示例数据#
3个基因集列表,为list格式
1
2
3
4
5
|
genes <- paste0("gene",1:1000)
set.seed(20210302)
gene_list <- list(A = sample(genes,100),
B = sample(genes,200),
C = sample(genes,300))
|
一、VennDiagram#
-
VennDiagram包是绘制韦恩图的一个经典包。
-
它不是基于ggplot体系,但可以转换为ggplot对象(个人更习惯些)。
-
另外每次绘图时,但会生成一个日志log文件,比较麻烦。
参考教程:https://www.r-graph-gallery.com/14-venn-diagramm.html
1 基本使用#
关键是设置filename 参数;但无论哪一种方法都会生成log文件。
(1)如果为正常名,则直接保存文件至本地;
(2)如果设置为NULL值,那么会得到gList图像,可配合grid.draw {grid}在R界面显示
1
2
3
4
5
6
7
8
9
10
11
12
13
|
# install.packages("VennDiagram")
library(VennDiagram)
#(1)保存图片至本地
venn.diagram(gene_list, filename = "test.pdf",
height = 3000, width = 3000)
#(2)在R界面显示
venn.plot = venn.diagram(gene_list,
filename = NULL)
grid.draw(venn.plot)
file.remove(list.files(pattern = "*log"))
dev.off()
|
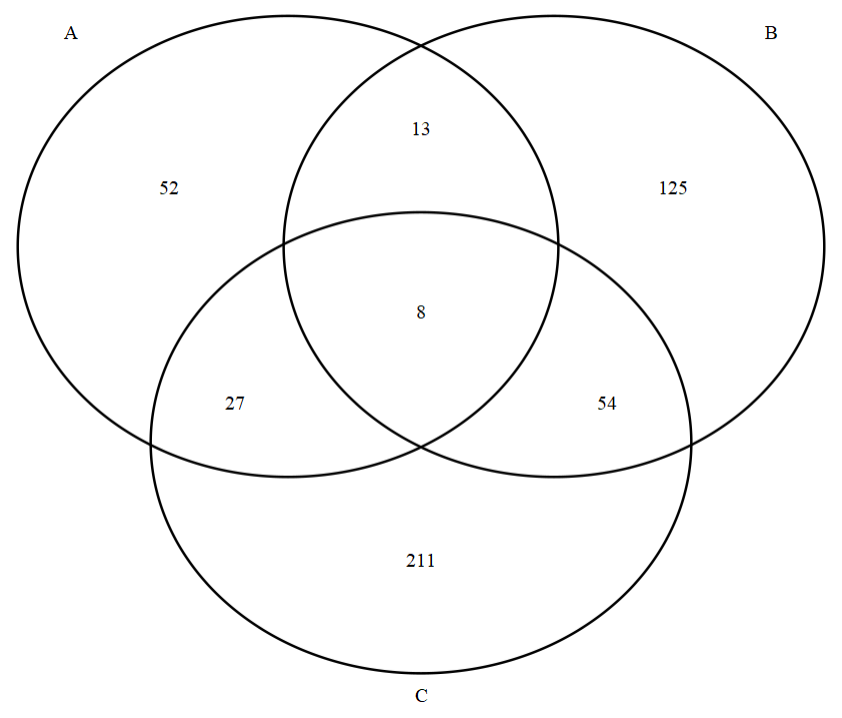
2 细节调整#
2.1 类别名#
即上图中的A、B、C,包括如下调整方面
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
|
#(1) 类别名标签
category.names
#(2) 字体大小(相对1)
cat.cex = 0.6
#(3) 字体格式
cat.fontface = "bold" #粗体
cat.fontfamily = "sans" #宋体
#(4) 字体颜色
cat.col = c("#FF0000", "#00FF00", "#0000FF")
#(5) 标签围绕图中心的角度位置
cat.pos = c(-27, 27, 135) #0度为12点方向,顺时针旋转
#(6) 类别名距离圆的距离
cat.dist = c(0.055, 0.055, 0.085)
#(7) 标签自旋转角度(逆时针)
rotation.degree = 60
rotation = 1 #适合3 set的情况
##示例
venn.plot = venn.diagram(gene_list[1:3], filename = NULL,
category.names = LETTERS[1:3],
cat.cex = 1.5,
cat.fontface = "bold",
cat.pos = c(-20, 20, 180),
cat.col = rainbow(3))
grid.draw(venn.plot)
|
2.2 圆的格式#
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
#(1) 填充颜色
fill = rainbow(3)
#(2) 边的颜色
col = rainbow(3)
#(3) 边的宽度
lwd = 2
#(4) 边的类型
lty = 'blank' # dashed dotted
##示例
venn.plot = venn.diagram(gene_list, filename = NULL,
fill = RColorBrewer::brewer.pal(4, "Pastel2"),
col = RColorBrewer::brewer.pal(4, "Pastel2"),
lwd = 2, lty = "dashed")
grid.draw(venn.plot)
|
我认为VennDiagram与ggVennDiagram包最大的不同就在于圆的填充颜色
2.3 交集数值格式#
1
2
3
4
5
|
venn.plot = venn.diagram(gene_list,
cex = 2, #设置大小
fontface = "bold", #设置格式
filename = NULL)
grid.draw(venn.plot)
|
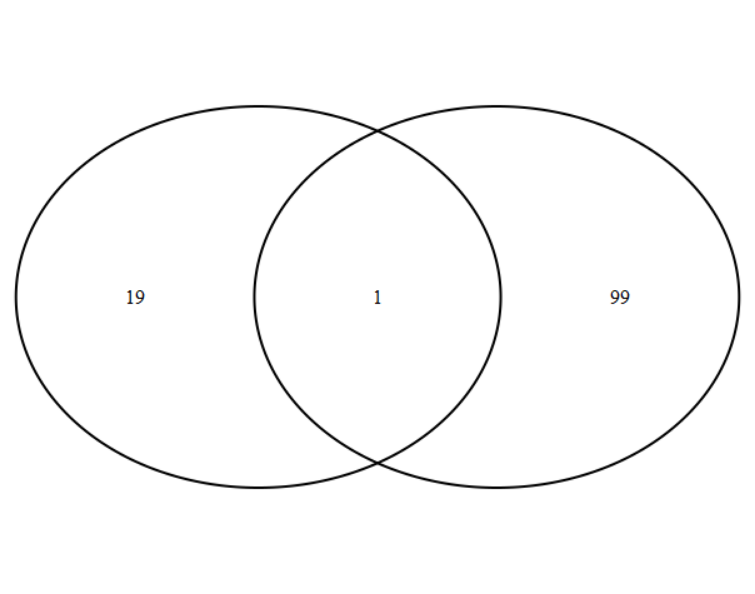
3 特殊的2 sets#
- 当绘制两个set的交集情况时,圆的面积大小与对应数目成正比;
- 而且当交集数目过小时,会自动使用引导线注释
1
2
3
4
5
6
7
|
gene_list2 <- list(A = sample(genes,20),
B = sample(genes,100))
venn.plot = venn.diagram(gene_list2,
filename = NULL)
grid.draw(venn.plot)
file.remove(list.files(pattern = "*log"))
dev.off()
|
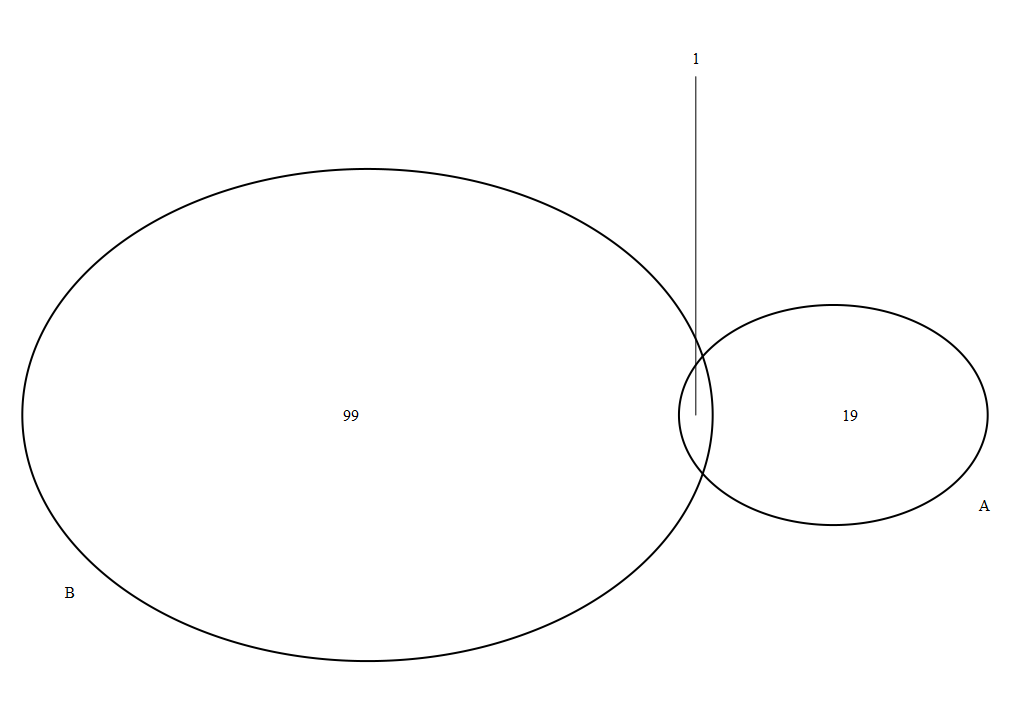
- 可通过
ext.系列参数修改这根引导线格式,例如直接取消~
1
2
3
4
5
6
7
8
9
10
|
# ext.text = F
# ext.percent = 0 重叠数目低于多少百分比,使用线
# ext.pos
# ext.line.lwd
venn.plot = venn.diagram(gene_list2,
ext.text = F,
filename = NULL)
grid.draw(venn.plot)
file.remove(list.files(pattern = "*log"))
dev.off()
|
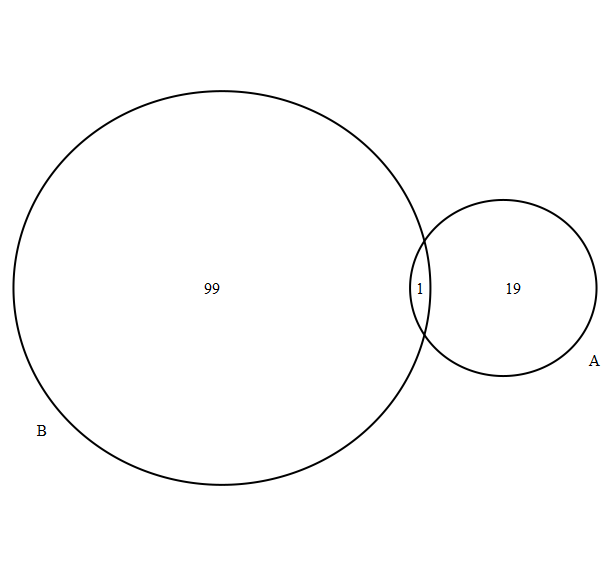
- 如果不想让圆的面积与数值存在这种对应关系,可通过如下方式进行绘制
1
2
3
4
5
6
7
8
9
10
11
12
13
|
venn.plot <- draw.pairwise.venn(
area1 = 50,
area2 = 50,
cross.area = 20)
dev.off()
#修改展示标签内容
venn.plot[[5]]$label = length(setdiff(gene_list2[[1]], gene_list2[[2]]))
venn.plot[[6]]$label = length(setdiff(gene_list2[[2]], gene_list2[[1]]))
venn.plot[[7]]$label = length(intersect(gene_list2[[1]], gene_list2[[2]]))
grid.draw(venn.plot)
file.remove(list.files(pattern = "*log"))
dev.off()
|
4 转为ggplot2对象#
1
2
3
4
5
|
library(ggplot2)
library(ggplotify)
library(cowplot)
as.ggplot(plot_grid(grobTree(venn.plot))) +
ggtitle("This is title")
|
二、ggVennDiagram#
ggVennDiagram包是基于ggplot绘图体系的,因此适用于ggplot的语法结构。
主要参考教程:https://mirror.linux.duke.edu/cran/web/packages/ggVennDiagram/vignettes/using-ggVennDiagram.html
1 基本使用#
1
2
3
4
5
6
7
8
9
|
# install.packages("ggVennDiagram")
library(ggVennDiagram)
packageVersion("ggVennDiagram")
# [1] ‘1.2.0’
library(ggplot2)
library(patchwork)
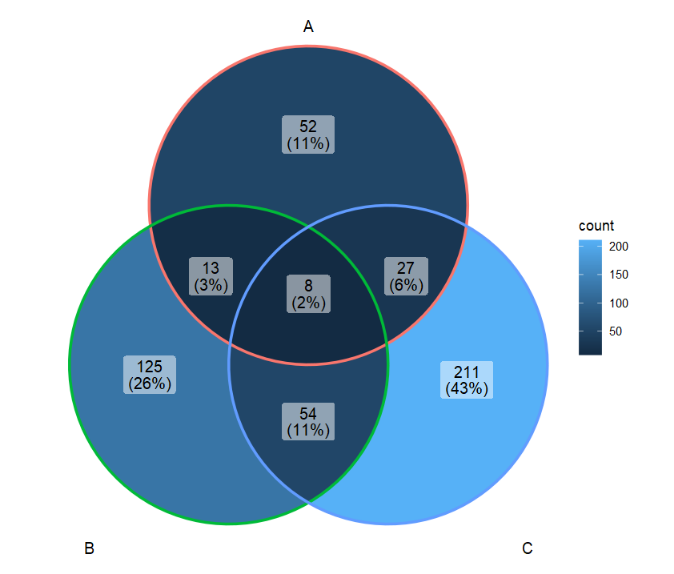
ggVennDiagram(gene_list)
|
2 细节调整#
2.1 类别名#
1
2
3
4
5
6
7
8
9
10
11
12
13
14
|
#(1) 类别名标签
category.names = c("AA","BB","CC","DD")
#(2) 字体大小
set_size = 8
#(3) 字体颜色
set_color = rainbow(4)
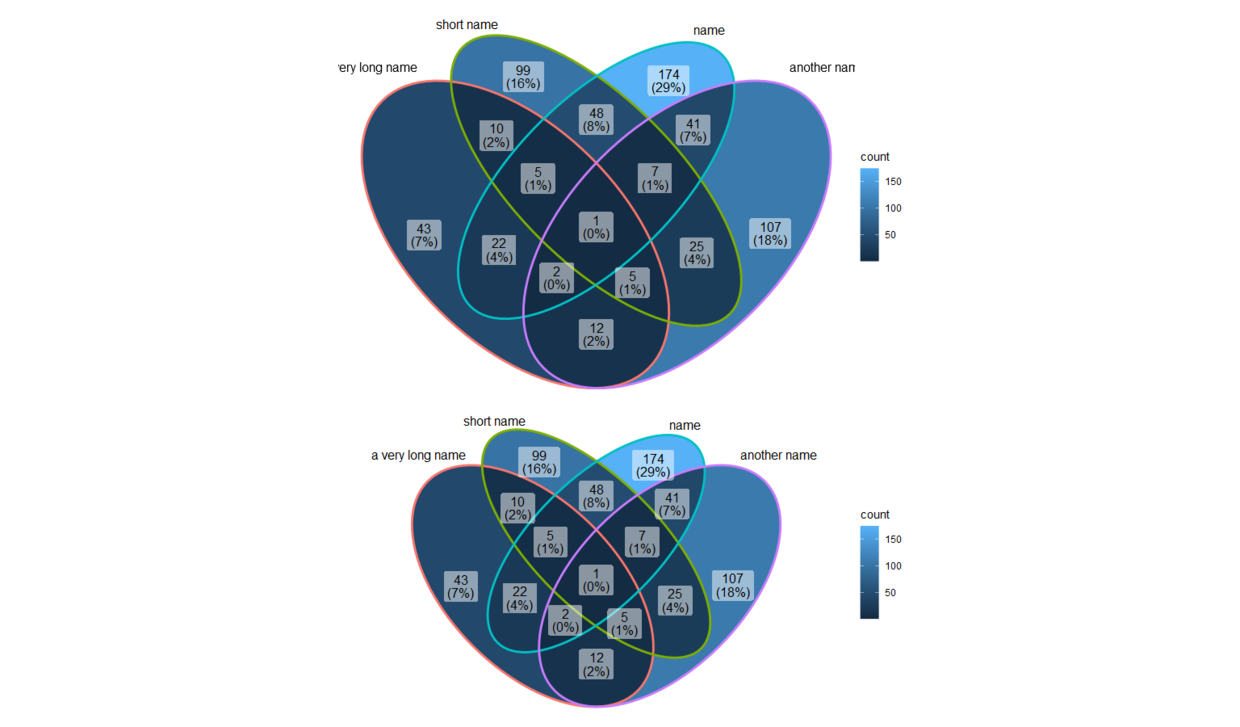
##有时名字太长,可能会显示不全,可以通过ggplot2函数拉宽画布
p1=ggVennDiagram(gene_list,
category.names = c("a very long name","short name","name","another name"))
p2=ggVennDiagram(gene_list,
category.names = c("a very long name","short name","name","another name")) +
scale_x_continuous(expand = expansion(mult = .2))
p1 / p2
|
2.2 圆的格式#
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
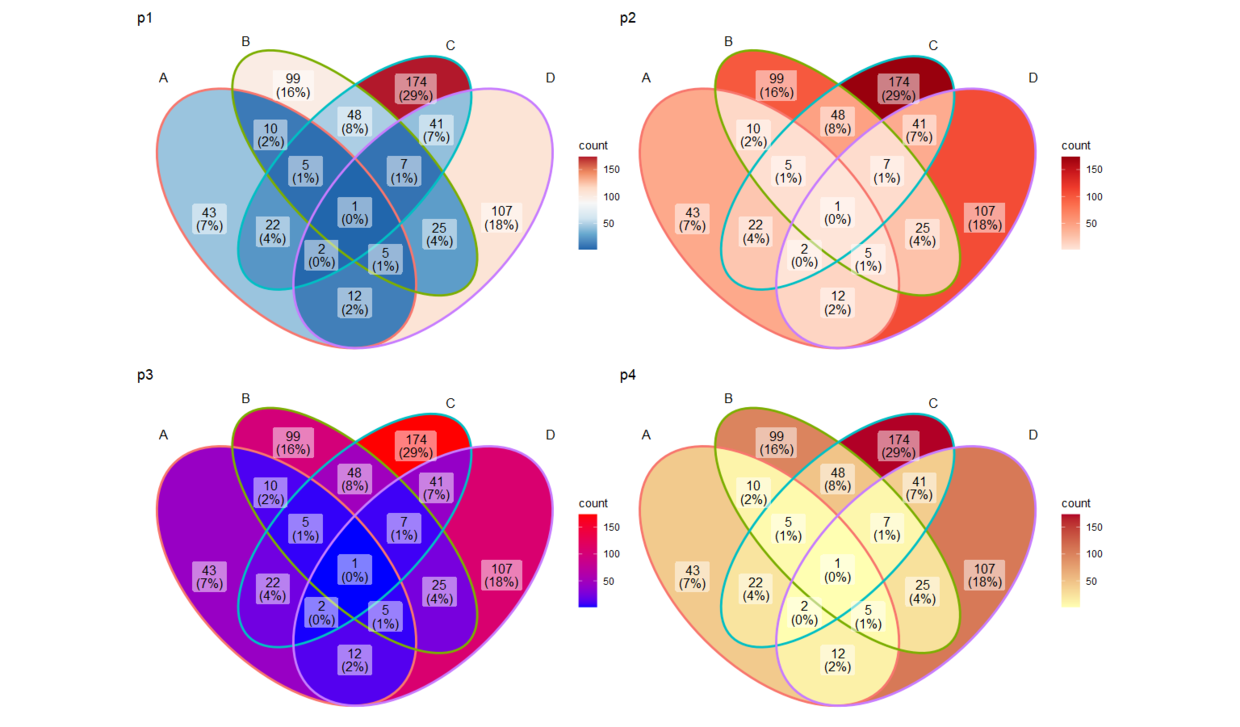
#(1) 填充颜色与数值大小相关,为渐变填充色。
#可通过ggplot的相关函数进行配色
RColorBrewer::display.brewer.all()
p1 = ggVennDiagram(gene_list) +
scale_fill_distiller(palette = "RdBu") + ggtitle("p1")
p2 = ggVennDiagram(gene_list) +
scale_fill_distiller(palette = "Reds", direction = 1) + ggtitle("p2")
# If -1, the order of colours is reversed.
p3 = ggVennDiagram(gene_list) +
scale_fill_gradient(low="blue",high = "red") + ggtitle("p3")
# https://colorbrewer2.org/
p4 = ggVennDiagram(gene_list) +
scale_fill_gradient(low="#ffffb2",high = "#b10026") + ggtitle("p4")
(p1 | p2) / (p3 | p4)
|
1
2
3
4
5
6
|
#(2)边的相关设置参数
scale_color_* #设置颜色
edge_size #设置粗细
edge_lty #设置类型
ggVennDiagram(gene_list, edge_lty = "dashed", edge_size = 1) +
scale_color_brewer(palette = "Set1")
|
2.3 交集数值格式#
参看上图可知,默认为数值加百分比格式,且为白色透明度背景
1
2
3
4
5
6
7
8
|
#(1)交集标签内容
label = # c("both", "count", "percent", "none")
#(2)背景透明度
label_alpha = 0
#(3)标签字体颜色
label_color = "red"
#(4)标签字体大小
label_size = 5
|
三、UpSetR#
当可视化多个(>5)集合的交集情况时,传统韦恩图的效果就比较复杂,不够直观。
UpSetR包可进行相对更加高效的展示,而且用法也很简单。
参考:https://cran.r-project.org/web/packages/UpSetR/vignettes/
1、基本使用#
1
2
3
4
5
6
|
# install.packages("UpSetR")
library(UpSetR)
#`upset()`绘图函数默认接受的集合对象为表格;
# 如果提供为list,包装在`fromList()`函数内部即可
upset(fromList(gene_list))
|
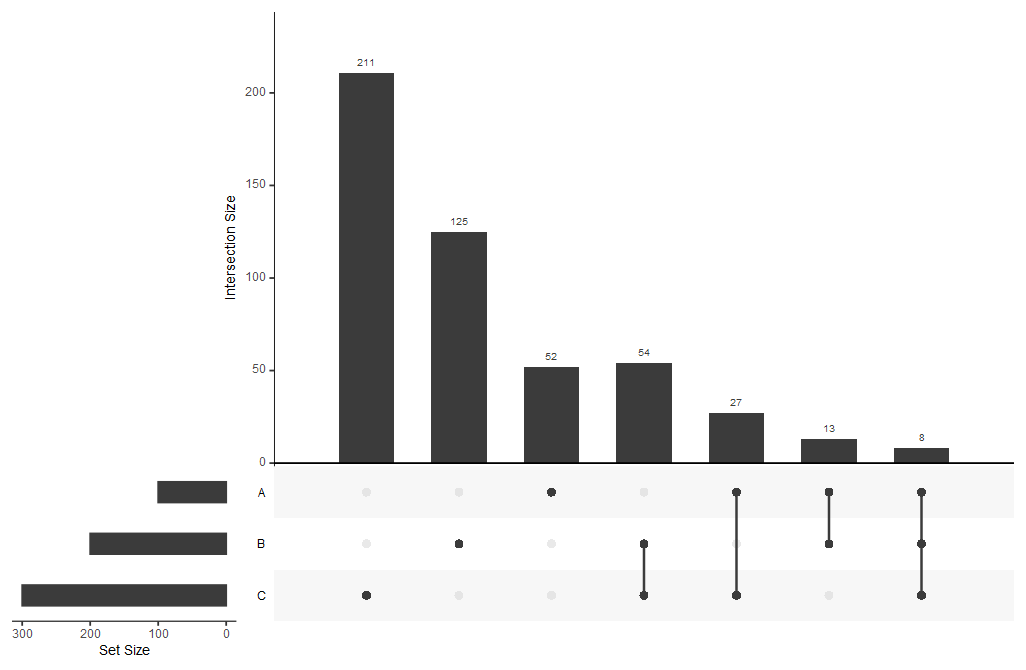
2、细节调整#
2.1 左下角柱状图#
每一列表示一类集合,柱高表示集合大小。
1
2
3
4
5
6
|
#(1) 指定选择哪些set集合纳入分析
sets = names(gene_list)
#(2) 设置展示集合的数目默认选择Size Top 5的集合。
nsets = 5
#(2) 集合排列顺序, 默认为 从上到下为size从小到大的顺序
keep.order = TRUE #设置是否按照提供数据的集合顺序展示
|
2.2 右上角柱状图#
-
nintersects=40 :默认最多展示40列;设置为NA值则展示所有可能组合
-
empty.intersections = "on" :设置展示为0的交集组合
-
cutoff=3 筛选大于一定数量的交集类别
-
order.by = "degree" 表示先按照组成集合的类别数排序
order.by = "freq" 表示仅按照集合大小排序
decreasing = T 默认均为降序排列,
2.3 文本标签设置#
1
2
3
4
5
6
7
8
|
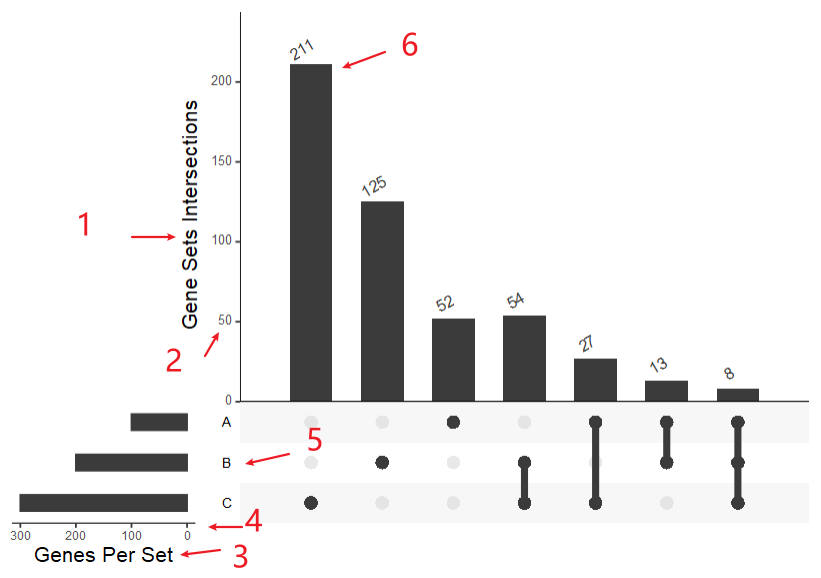
upset(fromList(gene_list),
number.angles = 30,
point.size = 3.5, #左下角的点
line.size = 2, #左下角的线
mainbar.y.label = "Gene Sets Intersections",
sets.x.label = "Genes Per Set",
mb.ratio = c(0.7, 0.3),
text.scale = c(1.5, 1.1, 1.5, 1, 1.2, 1.5)) #见下图箭头)
|