1、关于RWR#
1.1 算法简介#
-
Random Walk with Restart,RWR重启随机游走算法
-
在给定的一个由节点和边组成的网络结构中(下面均已PPI蛋白相互作用网络为例),选择其中一个或者一组基因。我们想知道其余的哪些基因与我们先前所选择的一个或者一组基因最相关。此时可以用到RWR,简单原理如下:
-
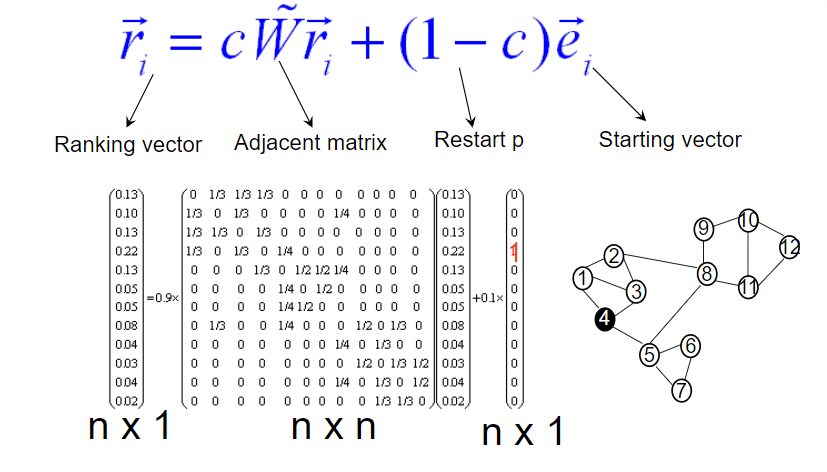
RWR的公式如下。其中1-c表示重启概率,e表示起始节点,W表示网络邻接矩阵
$$
\vec{r_i} = cW*\vec{r_i} + (1-c) \vec{e_i}
$$
-
如下例图所示,假设对于有12个蛋白互作关系组成PPI网络。我们想知道蛋白4与网络中其它蛋白的相关性排名如何。
(1)选取蛋白4作为起始节点,随机向邻居节点走一步(蛋白1、3、5)。但是在每一次走向邻居节点时,都有一定概率返回到起始节点(蛋白4)。
(2)假设走到了节点1,然后继续向邻居节点走一步(蛋白2,3),也仍然有一定概率返回起始节点。
(3)如此往复循环,最终从蛋白4走到网络其余每个蛋白的节点概率趋于收敛,作为与蛋白4的相关性度量指标。
1.2 RandomWalkRestartMH包简介#
RandomWalkRestartMH R包2018年最初发表,可用于多种复杂生物网中应用RWR算法,下面的笔记主要参考自官方文档。
按照文档中的定义,有三种类型的网络–
- Monoplex Network:只有一种类型节点,一个类型边。例如常规的PPI网络:节点为蛋白,边为互作关系。
- Heterogeneous Network:本质上是两个网络(例如PPI网路与疾病相似性网络),节点类型不相同(前者为蛋白,后者为疾病)。依靠两类节点的关系(某疾病与某蛋白功能异常密切相关)合并而成。

- Multiplex Network:本质上也是两个网络,但节点类型相同。例如PPI网络与通路网络

1
2
3
4
5
6
7
|
#安装,建议从github安装
#BiocManager::install("RandomWalkRestartMH")
devtools::install_github("alberto-valdeolivas/RandomWalkRestartMH")
packageVersion("RandomWalkRestartMH")
# [1] ‘1.13.1’
#检查包的版本与官文示例文档的版本要一致
#该包要求为igraph定义的网络结构,也需要安装。
|
2、分析实例#
2.1 Monoplex Network#
- 在给定PPI网络中,寻找与指定基因(集)最相关的基因
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
|
library(RandomWalkRestartMH)
library(igraph)
##(1) 加载数据:PPI网络的igraph对象
#使用RandomWalkRestartMH的示例数据,下同
data(PPI_Network)
PPI_Network #包含4317个蛋白的18062条互作关系
# IGRAPH 3bec0a4 UN-- 4317 18062 --
# 这个PPI网络不包含互作强度
edge_attr(PPI_Network)
# named list()
##(2) 创建Multiplex对象,计算出邻接矩阵
PPI_MultiplexObject <- create.multiplex(list(PPI=PPI_Network))
PPI_MultiplexObject
AdjMatrix_PPI <- compute.adjacency.matrix(PPI_MultiplexObject) #稀疏矩阵,1表示对应行和列的蛋白存在互作
colSums(AdjMatrix_PPI[,1:4])
# A2M_1 AAGAB_1 AAMDC_1 AAMP_1
# 45 2 2 4
AdjMatrixNorm_PPI <- normalize.multiplex.adjacency(AdjMatrix_PPI) #标准化,使蛋白到所有邻居蛋白的概率和=1
##(3) 确定起始节点,应用RWR算法,计算相关节点分数
SeedGene <- c("PIK3R1")
RWR_PPI_Results <- Random.Walk.Restart.Multiplex(AdjMatrixNorm_PPI,
PPI_MultiplexObject,SeedGene)
# Top 10 ranked Nodes:
# NodeNames Score
# 1 GRB2 0.006845881
# 2 EGFR 0.006169129
# 3 CRK 0.005674261
# 4 ABL1 0.005617041
# 5 FYN 0.005611086
# 6 CDC42 0.005594680
# 7 SHC1 0.005577900
# 8 CRKL 0.005509182
# 9 KHDRBS1 0.005443541
# 10 TYRO3 0.005441887
#
# Seed Nodes used:
# [1] "PIK3R1"
|
有权重的Monoplex Network#
- 基本步骤相同,只需要给igraph对象添加边的权重weight信息。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
|
#STRING蛋白相互作用数据可使用STRINGdb包下载
#这里就参考文档,随机模拟
##(1)
library(RandomWalkRestartMH)
library(igraph)
data(PPI_Network)
#随机模拟PPI互作强度,设置igraph edge的weight属性
PPI_Network <- set_edge_attr(PPI_Network,"weight",E(PPI_Network),
value = runif(ecount(PPI_Network)))
edge_attr(PPI_Network) %>% names()
# weight
##(2) 创建Multiplex对象,计算出邻接矩阵
PPI_MultiplexObject <- create.multiplex(list(PPI=PPI_Network))
PPI_MultiplexObject
AdjMatrix_PPI <- compute.adjacency.matrix(PPI_MultiplexObject)
colSums(AdjMatrix_PPI[,1:4])
# A2M_1 AAGAB_1 AAMDC_1 AAMP_1
#22.4898374 0.4895032 1.4202684 1.2030568
AdjMatrixNorm_PPI <- normalize.multiplex.adjacency(AdjMatrix_PPI)
##(3) 确定起始节点,应用RWR算法,计算相关节点分数
SeedGene <- c("PIK3R1")
RWR_PPI_Results <- Random.Walk.Restart.Multiplex(AdjMatrixNorm_PPI,
PPI_MultiplexObject,SeedGene)
# Top 10 ranked Nodes:
# NodeNames Score
# 1 EGFR 0.010790996
# 2 CRKL 0.010145105
# 3 GSPT1 0.009818236
# 4 AXL 0.009428844
# 5 NFKBIA 0.009418182
# 6 ITSN1 0.009385254
# 7 ZDHHC17 0.009369438
# 8 GAB2 0.009291153
# 9 CXCL2 0.009027423
# 10 STAT3 0.008956187
#
# Seed Nodes used:
# [1] "PIK3R1"
|
2.2 Heterogeneous Network#
- 已知疾病SHORT综合征(OMIM的id为269880)的关键基因为PIK3R1。据此发现与之最相关的疾病、基因
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
|
library(RandomWalkRestartMH)
library(igraph)
##(1) 构建两个网络
#构建PPI网络
data(PPI_Network)
PPI_MultiplexObject <- create.multiplex(list(PPI=PPI_Network))
#构建疾病相似度网络
data(Disease_Network)
Disease_MultiplexObject <- create.multiplex(list(Disease=Disease_Network))
##(2) 疾病与蛋白的关系数据
data(GeneDiseaseRelations)
head(GeneDiseaseRelations)
# hgnc_symbol mim_morbid
# 3 A2M 614036
# 4 A2M 104300
# 6 A4GALT 111400
# 根据两个网络,进一步筛选
GeneDiseaseRelations_PPI <-
GeneDiseaseRelations[which(GeneDiseaseRelations$hgnc_symbol %in%
PPI_MultiplexObject$Pool_of_Nodes),]
GeneDiseaseRelations_PPI <-
GeneDiseaseRelations_PPI[which(GeneDiseaseRelations_PPI$mim_morbid %in%
Disease_MultiplexObject$Pool_of_Nodes),]
dim(GeneDiseaseRelations_PPI)
# [1] 1330 2
##(3) 创建Multiplex对象,计算出邻接矩阵
PPI_Disease_Net <- create.multiplexHet(PPI_MultiplexObject,
Disease_MultiplexObject,
GeneDiseaseRelations_PPI)
PPI_Disease_Net
PPIHetTranMatrix <- compute.transition.matrix(PPI_Disease_Net)
dim(PPIHetTranMatrix)
# [1] 11264 11264
##(4) 每一层确定一个起始节点,应用RWR算法,计算相关节点分数
SeedGene <- c("PIK3R1")
SeedDisease <- c("269880")
RWRH_PPI_Disease_Results <-
Random.Walk.Restart.MultiplexHet(PPIHetTranMatrix,
PPI_Disease_Net,
Multiplex1_Seeds = SeedGene,
Multiplex2_Seeds = SeedDisease)
# RWRH_PPI_Disease_Results
# Top 10 ranked global nodes:
# NodeNames Score
# 1 PIK3R1 0.414259752
# 2 269880 0.372567325
# 3 615214 0.020817435
# 4 616005 0.020785895
# 5 194050 0.005868407
# 6 309000 0.005687206
# 7 262500 0.005681162
# 8 138920 0.005655559
# 9 223370 0.005655117
# 10 608612 0.005649291
#
# Top 10 ranked nodes from the first Multiplex:
# NodeNames Score
# 1479 GRB2 0.001965500
# 1081 EGFR 0.001754048
# 797 CRK 0.001636329
# 603 CDC42 0.001630575
# 19 ABL1 0.001623304
# 798 CRKL 0.001605543
# 1360 FYN 0.001598567
# 3405 SHC1 0.001597720
# 2583 PDGFRB 0.001596830
# 4027 TYRO3 0.001589911
#
# Top 10 ranked nodes from the second Multiplex:
# NodeNames Score
# 6352 615214 0.020817435
# 6705 616005 0.020785895
# 1699 194050 0.005868407
# 3625 309000 0.005687206
# 2901 262500 0.005681162
# 686 138920 0.005655559
# 2150 223370 0.005655117
# 4770 608612 0.005649291
# 4464 606176 0.005641777
# 1411 180500 0.005639919
#
# Seeds used:
# [1] "PIK3R1" "269880"
|


