1、层次聚类简介
1.1 计算步骤
层次聚类hierarchical clustering常用的是自下而上的聚合法(Agglomerative)。与之相对的是自顶而下的分裂法(Division)。
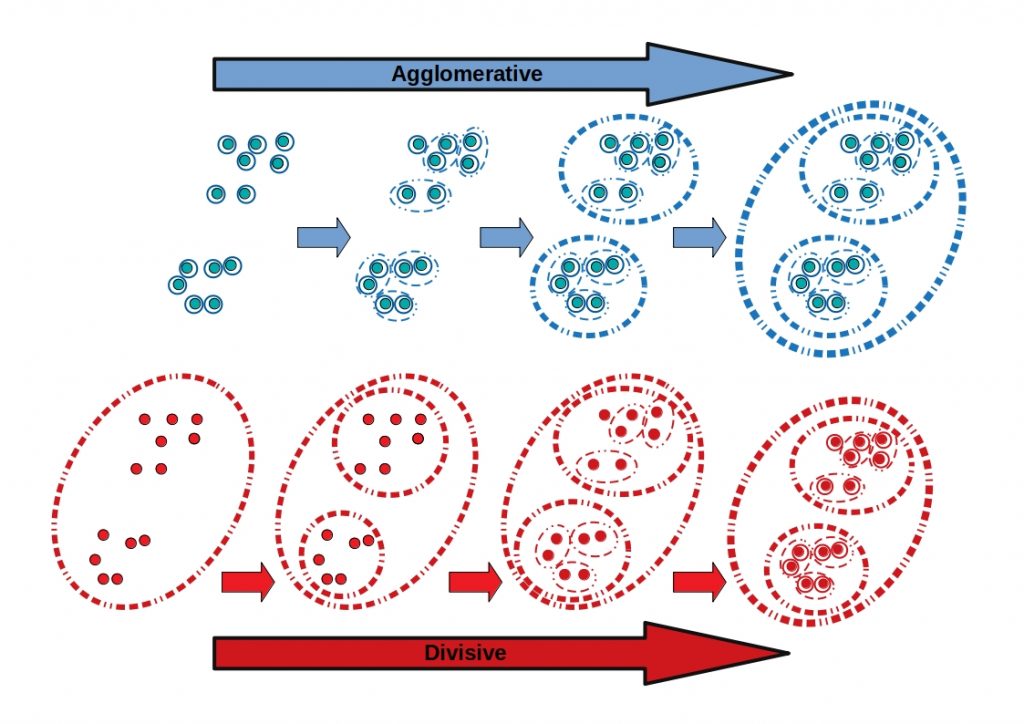
以聚合法为例:最初将数据集中每个单独样本视为一个聚类
(1)计算所有聚类两两之间的距离,通常为欧几里得距离。
(2)将最相似(距离最近)的两个聚类合并成一个聚类。
(3)重复上述步骤1、2,直至所有的所有样本都位于单个聚类中。
1.2 聚类距离
计算聚类之间的距离,有如下方式可供选择
- 质心链接:两聚类质心之间的距离;
- 单链接:两聚类最近样本之间的距离;
- 全链接:两聚类最远样本之间的距离;
- 均链接:两聚类所有样本之间距离的平均值;
- Ward方法:
- 首先计算每个聚类的类内距离平方和(类内方差)
- 然后对于所有聚类两两组合中,类内方差增加最少的组合方式。

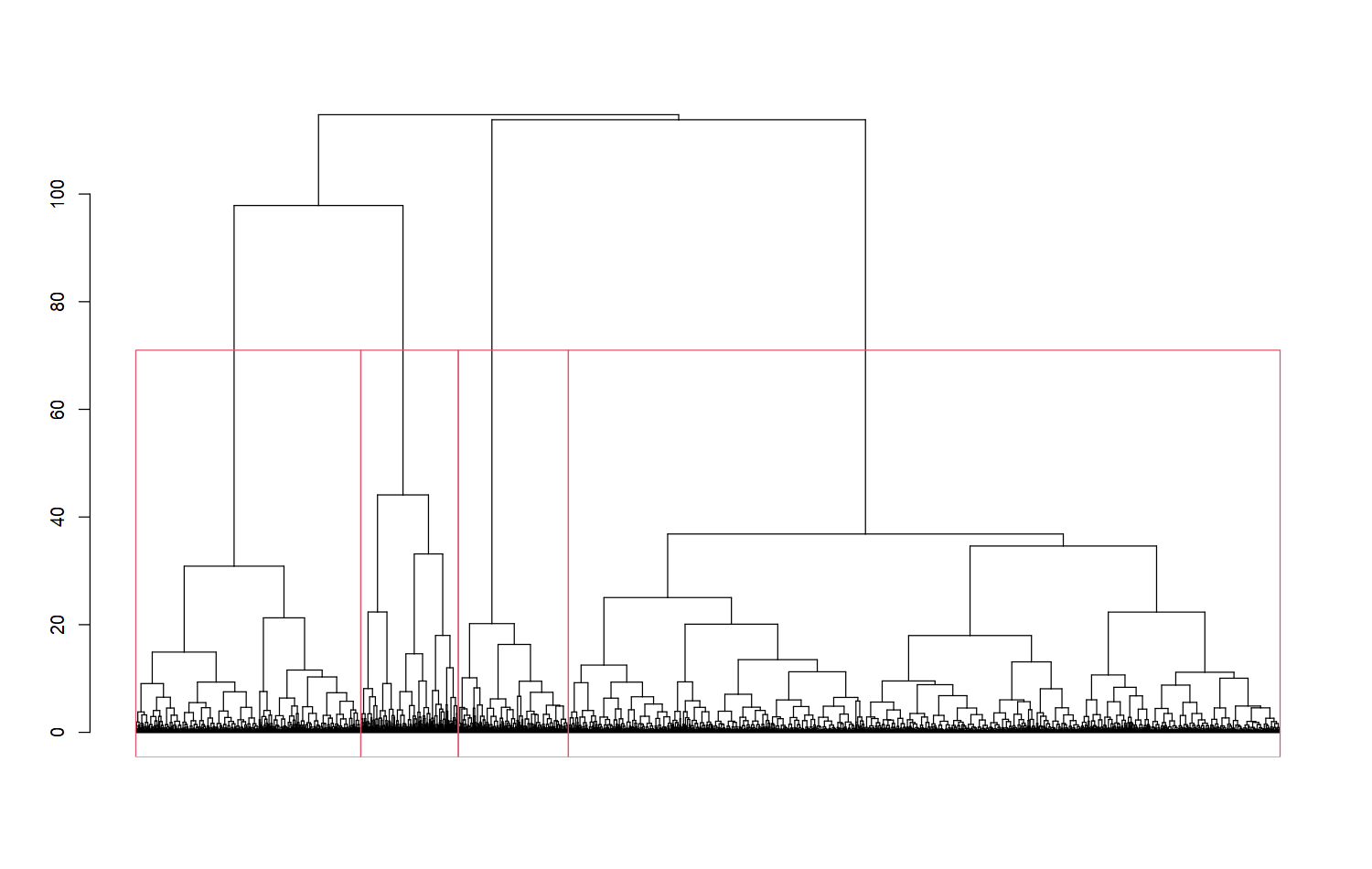
1.3 树状图切割聚类
对于层次聚类的结果通常以树状图(dendrogram)的方式展示。如下左图表示原始样本的分布情况;右图表示层次聚类的结果
- 水平线表示相应的两个聚类被合并在一起,而这两个聚类之间的距离对应水平线的高度。
层次聚类的优势在于保留了样本之间的层次结构,即样本与哪些样本更相似性,而又与哪些样本的关系比较远。
- 如下示例,从层次聚类结果来看,样本C更接近DEF,与AB较远
基于层次聚类结果,确定样本聚类的划定方式就是在树状图使用水平线切割,有如下两种思路
- 给定指定的距离进行切割;
- 给定指定的聚类数进行切割。

如上的聚类确定方式需要对数据集有预期的把握。如不确定,可比较不同的聚类结果的性能(上一节K均值算法中提到的聚类性能评价指标),选择最优的聚类结果。
2、代码实操
- (1) 原始数据归一化
|
|
- (2) 计算样本两两间距离
|
|
- (3) 层次聚类
|
|
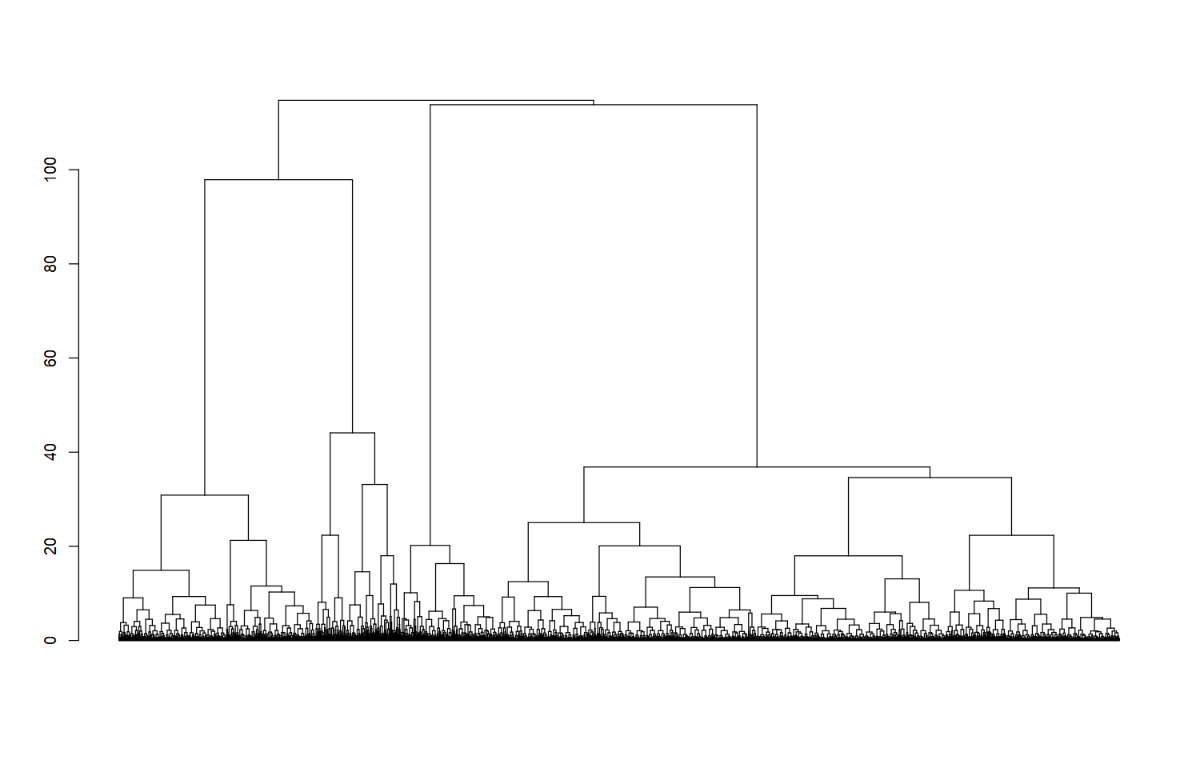
- (4) 确定聚类
|
|