算法简介#
t-SNE#
正态分布密度函数 ,其中σ表示标准差,μ表示均值
第一步:计算高维空间中任意两样本点的欧几里得距离。
第二步:对于任一特定节点,将其余节点与之的距离转换为以该节点为中心的正态分布的概率值。
正态分布的标准差与该样本点周围数据的密度成反比,但是具体关系依赖perplex超参数。该值越大表明越关注数据的全局特征,反之越关注数据的局部结构。
第三步:将对任一特点节点的其余节点概率处于它们的和,从而使得数据集中每个样本的概率总和为1。
$$
正态分布密度函数 \quad f(x) = \frac{1}{\sigma \sqrt{2\pi}}exp(\frac{-(x-\mu)^2}{2\sigma^2})
$$
$$
样本点j到点i的概率 \quad P_{j|i} = \frac{exp(-(||x_i-x_j||)^2 /2\sigma_i^2)}{\sum_{k\neq i}exp(-(||x_i-x_j||)^2 /2\sigma_i^2)}
$$
其中||Xi-Xj||表示高维空间中数据点i与数据点j的欧几里得距离

如上以所以样本点均为中心样本进行正态分布密度转换之后,可以得到一个概率矩阵p,用以描述每个样本与其它样本的相似程度。
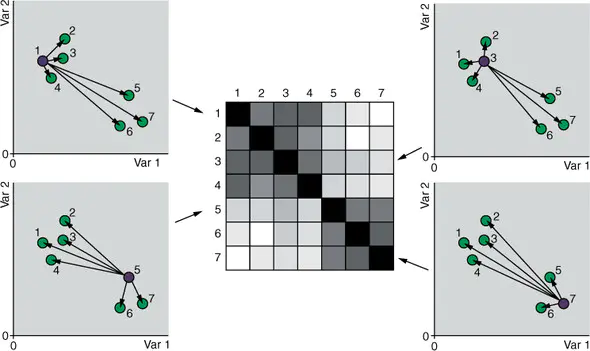
在二维的空间中,对所有数据点重新排布。使用上述方法再次计算新的概率矩阵与q。使用损失函数KL散度评价两个概率矩阵的相似性。采用梯度下降的方法,不断迭代的、”移动“二维空间中数据点的位置,使得KL散度指标逐渐降低,直至达到预期水平。
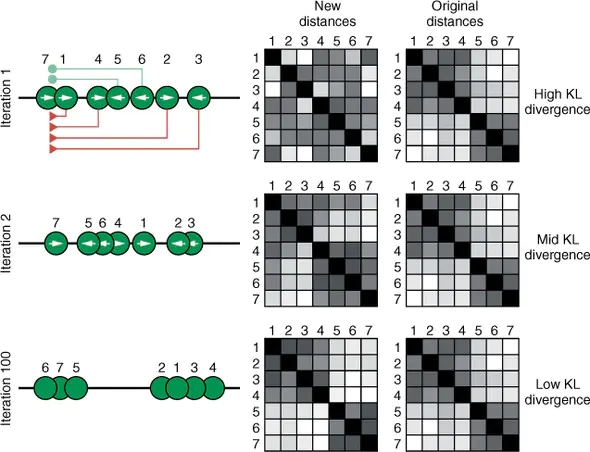
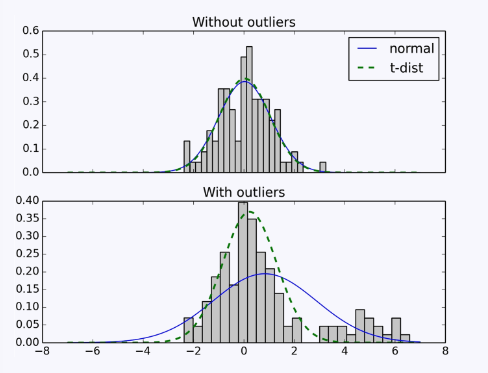
t-SNE中的t的含义是指在计算新的、二维空间中样本间的概率矩阵时,使用T分布将样本距离转换为概率值可以更好的兼容离群点的影响,识别出真正距离相近的样本。
UMAP#
UMAP的思路与tSNE类似,将样本在高维空间中的距离转换到二维平面中。与tSNE不同的是UMAP采用了所谓流形的概念,测量流形中样本之间的距离。
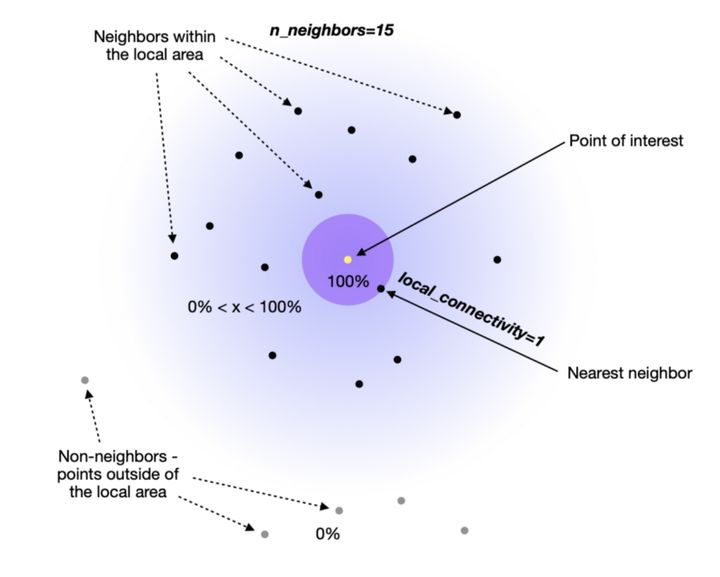
在学习流形时,有两个关键步骤
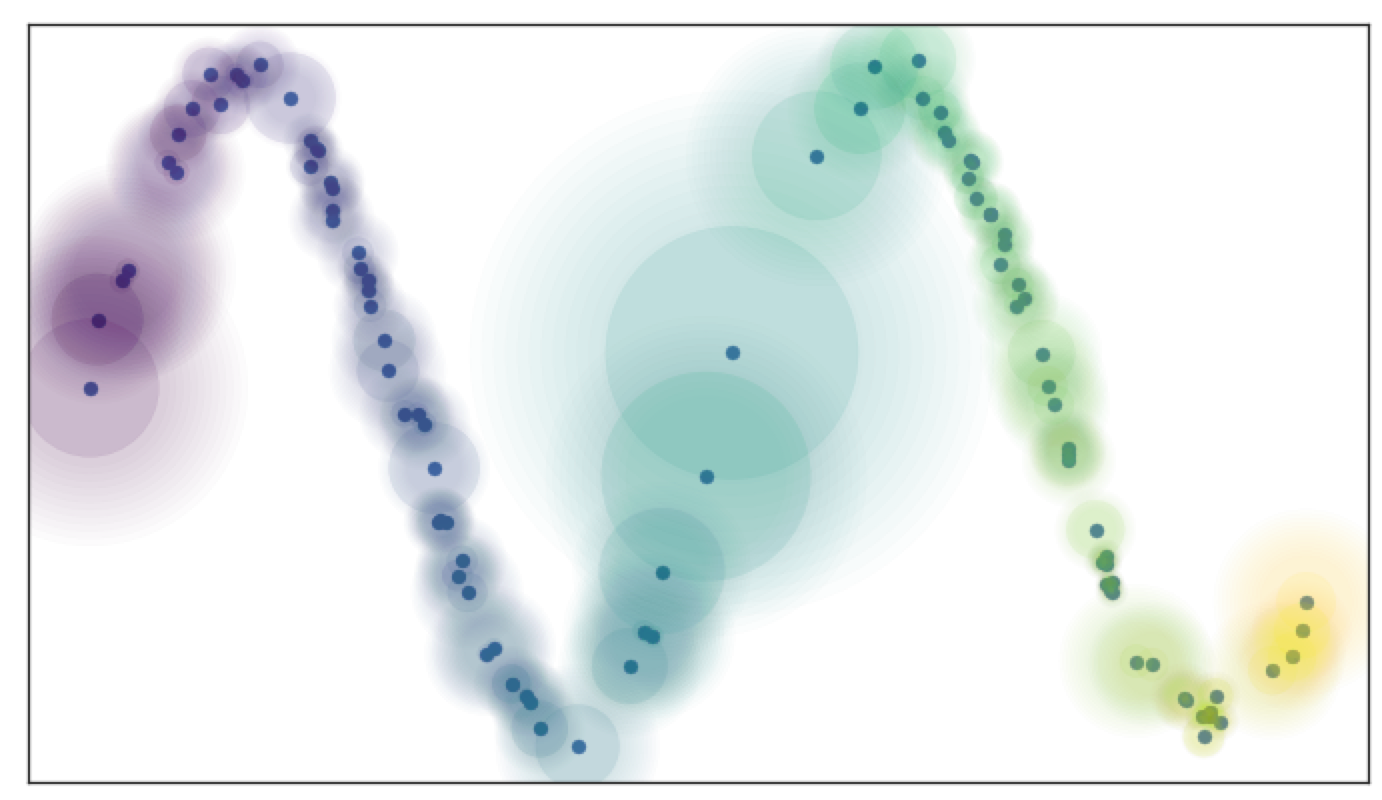
(1)对于既定的样本点进行局部连接,扩展最近的样本(local_connectivity=1),这样可以确保不存在孤立的样本。
(2)然后以局部连接样本为边界进行模糊扩展,确定近邻样本。可通过超参数设置,较大的值表示包含更多的近邻样本,更关注全局结构;反之更关注局部结构。
计算如此得到的流形中,得到所有样本与近邻样本之间的距离。下一步将数据放在两个维度的流形中,并迭代地移动数据点,直至新流形中样本间的距离等于沿着原始高维流形中样本间的距离。损失函数为交叉熵。
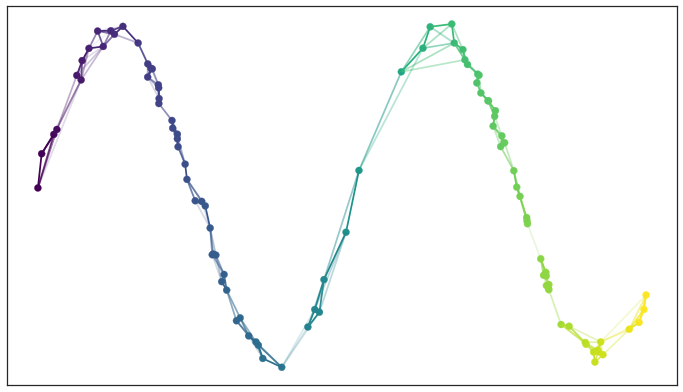
二者比较#
(1)tSNE与UMAP相对于PCA均属于非线性降维方式,获得的二维平面新轴不能用原始变量直接解释
(2)相比于tSNE,UMAP的具有更多的优势:计算开销小、同时保留了局部与全局结构;而且作为一种确定性算法,给定相同的输入可以给出相同的输出。
(3)对于tSNE,关注的更多是局部结构,即聚为一类的数据点具有很好的相似性;但是不同聚类之间的距离并没有任何含义。
(4)在实际分析时,往往先对高维数据进行PCA分析,然后对于保留的若干主成分再进行tSNE/UMAP降维
代码实操#
1
2
3
4
5
6
7
8
9
10
11
12
13
|
library(tidyverse)
data(banknote, package = "mclust")
swissTib = as_tibble(banknote)
# # A tibble: 6 x 7
# Status Length Left Right Bottom Top Diagonal
# <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 genuine 215. 131 131. 9 9.7 141
# 2 genuine 215. 130. 130. 8.1 9.5 142.
# 3 genuine 215. 130. 130. 8.7 9.6 142.
# 4 genuine 215. 130. 130. 7.5 10.4 142
# 5 genuine 215 130. 130. 10.4 7.7 142.
# 6 genuine 216. 131. 130. 9 10.1 141.
dat = swissTib[,-1]
|
1、t-SNE#
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
|
library(Rtsne)
?Rtsne
##(1) 相关参数[一般使用默认参数即可,这里主要了解一下]
# perplexity = 30 # 5~50 值越大表示越保留全局特征(相对而言)
# theta = 0. # 0~1 速度与精确度的平衡,越大越关注速度
# max_iter = 1000 # 迭代次数
# eta = 200 # 每次迭代,点移动的距离
# num_threads = 1 # 使用线程数
# pca = TRUE # 进行tSNE分析前,是否进行PCA分析
# initial_dims = 50 # 若进行PCA分析,保留的主成分数
# dims = 2 # 输出的tSNE维度数
##(2) 示例分析
set.seed(123) #设置随机种子,确保分析结果的一致性
tsne.list = Rtsne(dat)
tsne = tsne.list$Y %>% as.data.frame()
colnames(tsne) = c("tSNE_1","tSNE_2")
head(tsne)
# tSNE_1 tSNE_2
# 1 -8.127869 5.0456539
# 2 -14.330835 4.5799939
# 3 -13.687125 5.9405066
# 4 -13.203127 -0.3737462
# 5 -12.773563 8.5912141
# 6 -8.431319 4.1007125
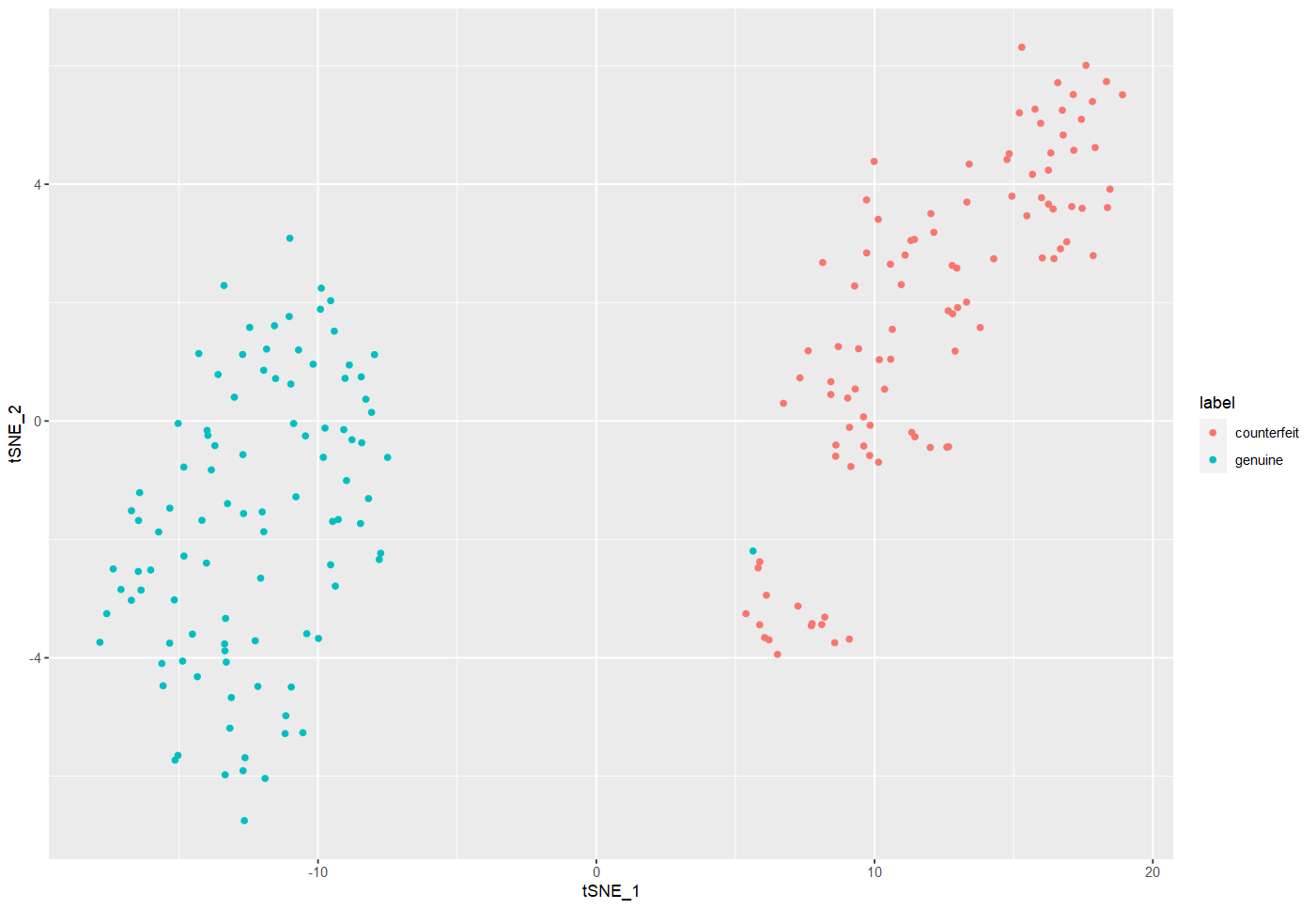
tsne$label = swissTib$Status
ggplot(tsne, aes(x=tSNE_1,y=tSNE_2,color=label)) +
geom_point()
|
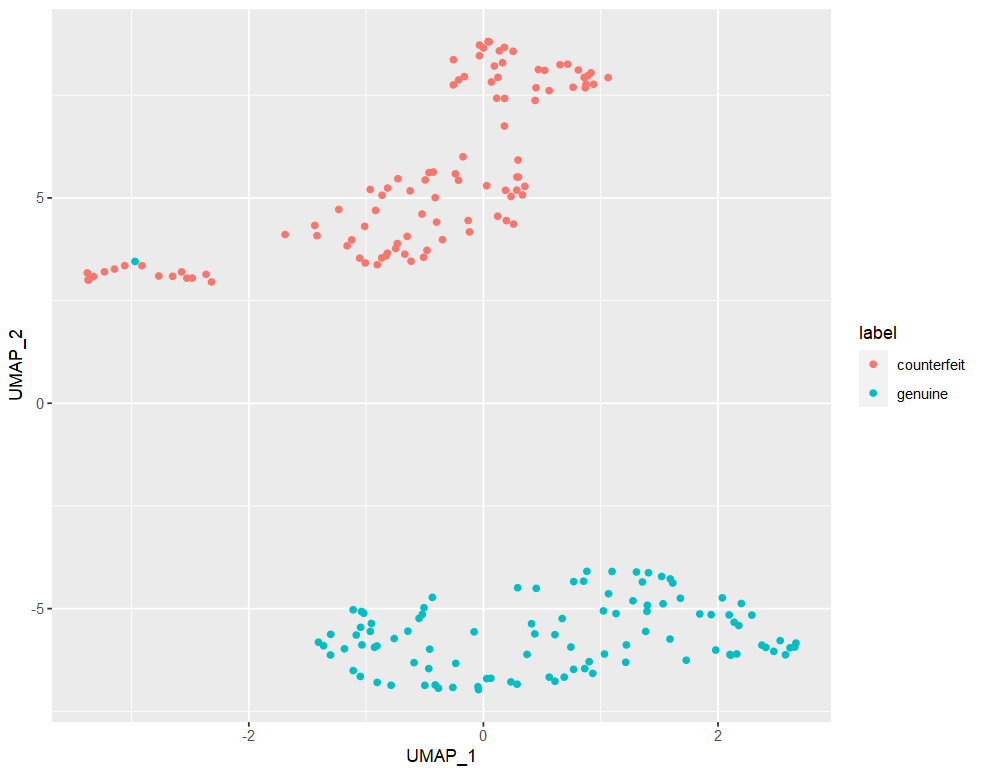
2、UMAP#
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
|
library(umap)
##(1) 查看默认参数
umap::umap.defaults
# umap configuration parameters
# n_neighbors: 15 #控制模糊搜索区域的半径,较大值的将包含更多的近邻样本,即更考虑全局结构
# n_components: 2 #输出维度数
# metric: euclidean #测量流形上样本的距离
# n_epochs: 200 #迭代次数
# input: data
# init: spectral
# min_dist: 0.1
# set_op_mix_ratio: 1
# local_connectivity: 1
# bandwidth: 1
# alpha: 1
# gamma: 1
# negative_sample_rate: 5
# a: NA
# b: NA
# spread: 1
# random_state: NA
# transform_state: NA
# knn: NA
# knn_repeats: 1
# verbose: FALSE
# umap_learn_args: NA
##(2) 示例分析
#不需要设定随机种子也能保证结果的一致性
umaps.list = umap(dat)
umaps = umaps.list$layout %>% as.data.frame()
colnames(umaps)=c("UMAP_1","UMAP_2")
head(umaps)
# UMAP_1 UMAP_2
# 1 1.407095 -4.126555
# 2 1.213177 -6.306884
# 3 1.980743 -6.008990
# 4 -1.303874 -6.129523
# 5 2.664741 -5.839394
# 6 1.355476 -4.351818
umaps$label = swissTib$Status
ggplot(umaps, aes(x=UMAP_1, y=UMAP_2, color=label)) +
geom_point()
|