Github仓库:https://github.com/openbiox/UCSCXenaShiny
Online App:https://shiny.zhoulab.ac.cn/UCSCXenaShiny/
Tutorial Book:https://lishensuo.github.io/UCSCXenaShiny_Book/
文章发表:Communications Biology, https://www.nature.com/articles/s42003-024-06891-2
-
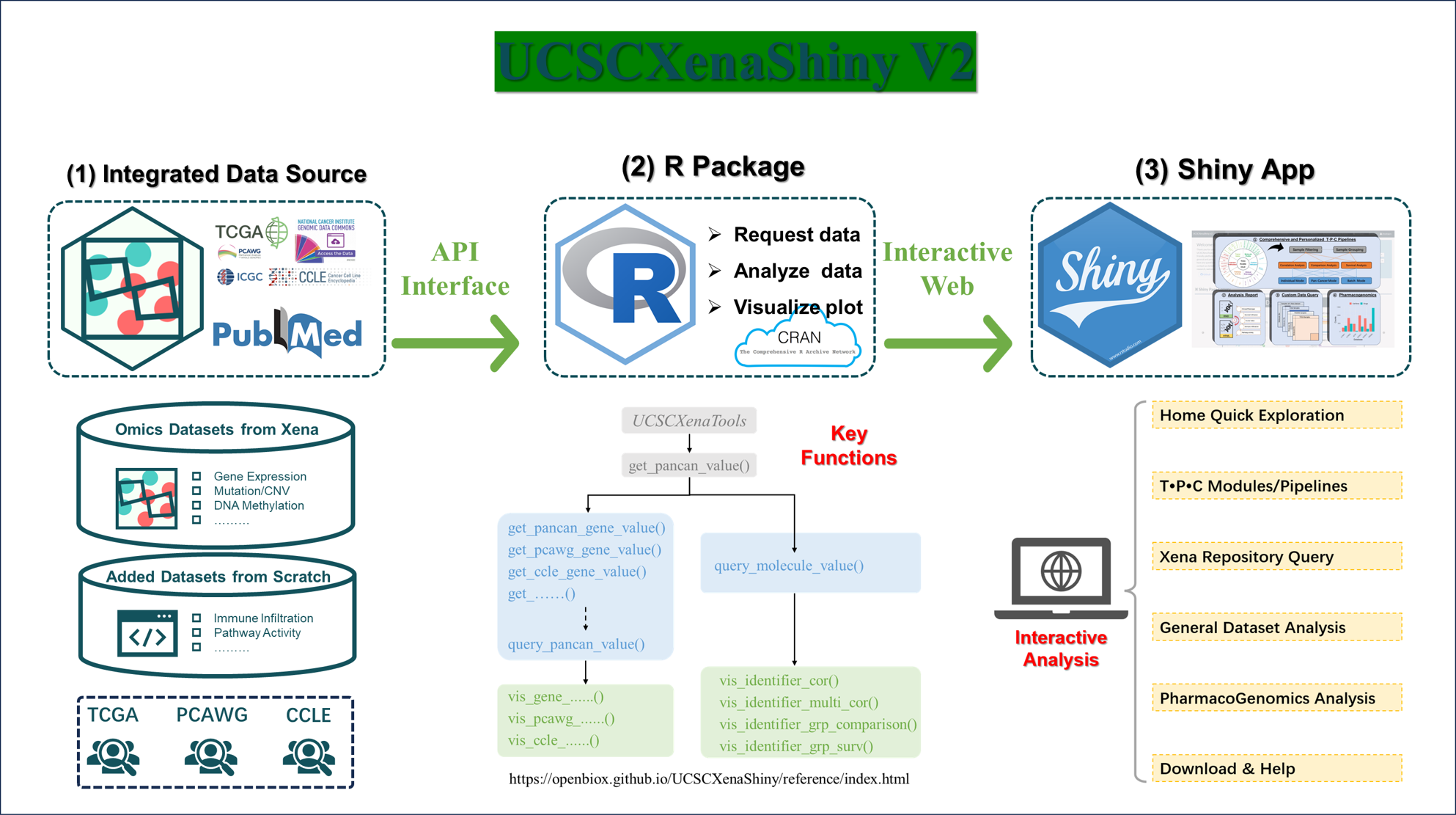
2024年9月,UCSCXenashiny v2版本正式发表在Communication Biology期刊上。此时更新旨在给用户以更加个性化、多元化的角度,高效地探索TCGA等大型肿瘤多组学队列数据。
-
关于UCSCXenashiny的详细介绍与功能用法,用户可参考我们精心准备的教程文档。下面将简要介绍工具各个板块的快速入门使用帮助。
1. 安装/访问
UCSCXenashiny是基于R语言开发的Shiny项目。用户有如下两种方式进入Shiny网页
(1)直接在线访问我们已经部署好的Shiny网站:https://shiny.zhoulab.ac.cn/UCSCXenaShiny/ 或者 https://shiny.hiplot.cn/ucsc-xena-shiny
- 优点:简单、直接
- 缺点:由于网络拥堵等原因时,偶尔可能会无法访问(如果长时间遇到这个问题,可通过Github或者邮件联系我们)
(2)在用户的R环境安装UCSCXenashiny包后,再本地启动Shiny网页
- 优点:相对比较稳定,R包也提供很多分析函数可供调用
- 缺点:需要对R语言操作有一定了解,对电脑内存有一定要求
对于第二种方式,我们提供了多种安装方式,包括CRAN、Conda、docker等。下面以CRAN为例:
|
|
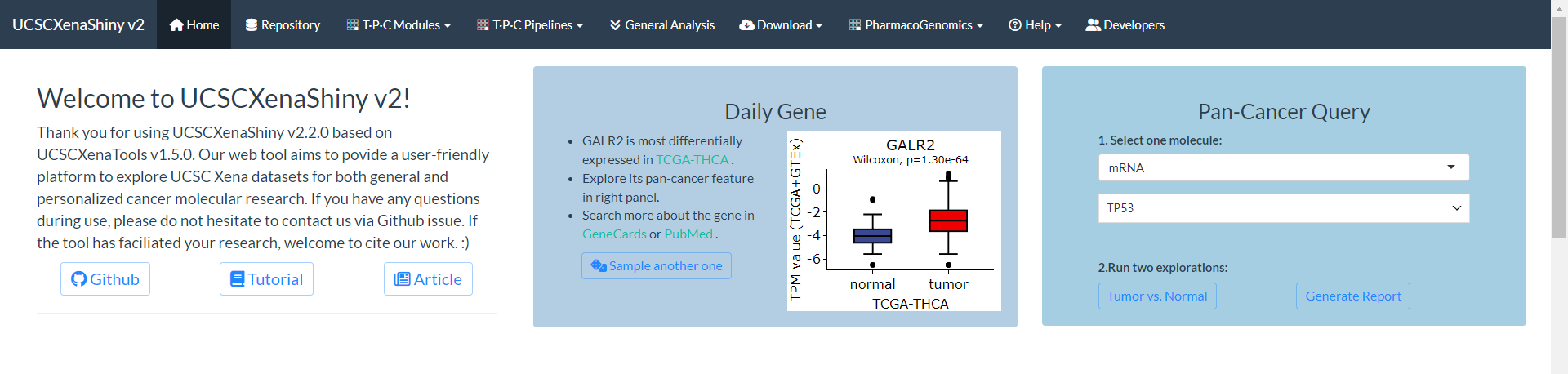
2. 首页导航
- 进入Shiny App的首页面后,最上方的导航栏展示了目前UCSCXenashiny支持的全部功能。
- 接下来是关于 V2版本的概述,以及两个特别设计的快速功能面板
- Daily Gene:每天展示一个基因,介绍它在哪一种TCGA肿瘤中相对正常样本差异表达最显著;
- Pan-Cancer Query:用户输入一个多组学分子后,可生成一个TCGA泛癌分析报告。
- 再往下分别是关于UCSCXenashiny的核心功能动态展示、TPC功能快速链接、以及更新日志。
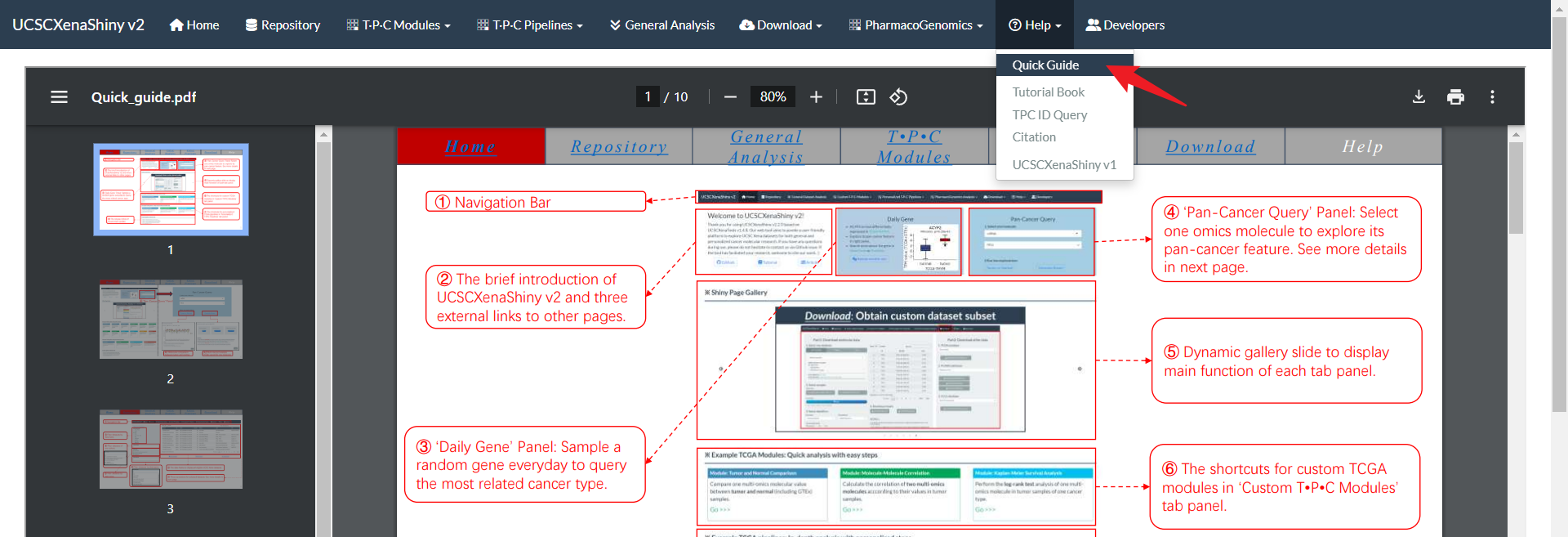
- 此外,用户可在导航栏的Help部分进入Quick Guide页面,查看每个模块的布局介绍。
3. TPC分析
- TPC 表示三个常见的大型肿瘤多组学项目,分别是TCGA, PCAWG, CCLE
- 基于UCSC Xena平台的数据优势,我们设计了两个系列的交互式分析页面,作为UCSCXenashiny的核心功能
3.1 TPC Modules
特点:功能相对具体,页面布局简洁,用户仅需要较少的步骤得到结果
以TCGA为例,我们共提供了3类模块:
-
比较分析类:① Tumor VS Normal (Box plot); ② Tumor VS Normal (Anatomy plot); ③ Mutation VS Wild
-
相关性分析类:① Molecule-Molecule; ② Molecule-Tumor Immune Infiltration; ③ Molecule-Immune Signature; ④ Molecule-TMB/Stemness/MSI; ⑤ Molecule-Pathway
-
生存分析类:① Kaplan-Meier; ② Cox regression
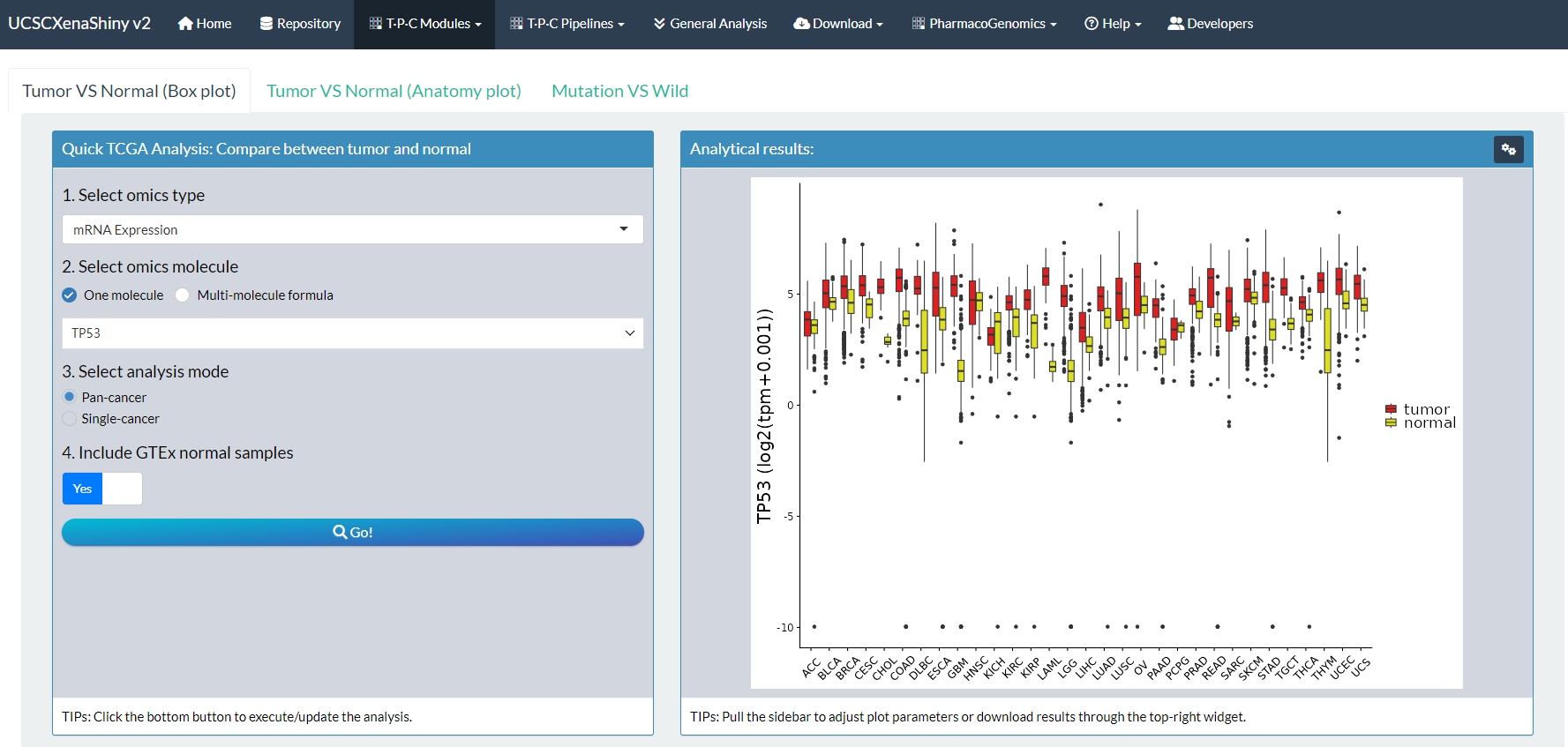
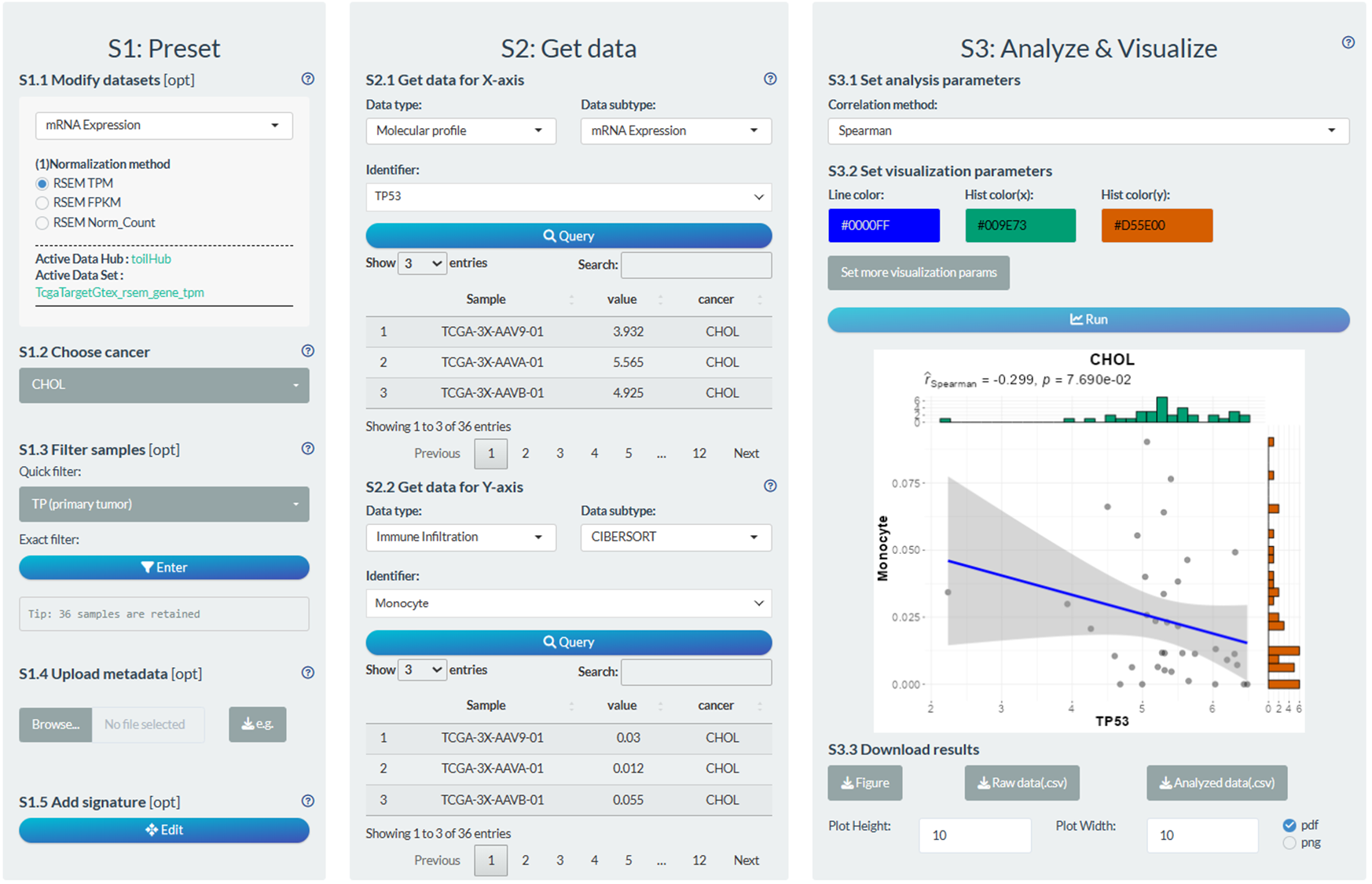
3.2 TPC pipelines
TPC分析流程的设计是本次V2更新的核心之一。以TCGA为例,共包括相关性分析、分组比较分析、生存分析三类流程,具有如下特点:
-
数据层面:大量的数据整合于一体,增加了分析的广度
- 7种组学分子:Gene Expression, Transcript Expression, DNA Methylation, Protein Expression, miRNA Expression, Gene Mutation, Copy Number Variation
- 4种非组学分子:4类Clinical Phenotype,3类Pathway Activity,7类Immune Infiltration,5类Tumor Index
-
操作层面:多元的操作设计,增加了分析的深度
- 个性化的数据样本过滤与分组功能,使得用户可以针对具有某种分子特征的肿瘤亚群进行特定分析;
- 除了基础分析模式外,我们还支持泛癌分析模式以及批量筛选模式,扩大了Pipeline的应用场景。
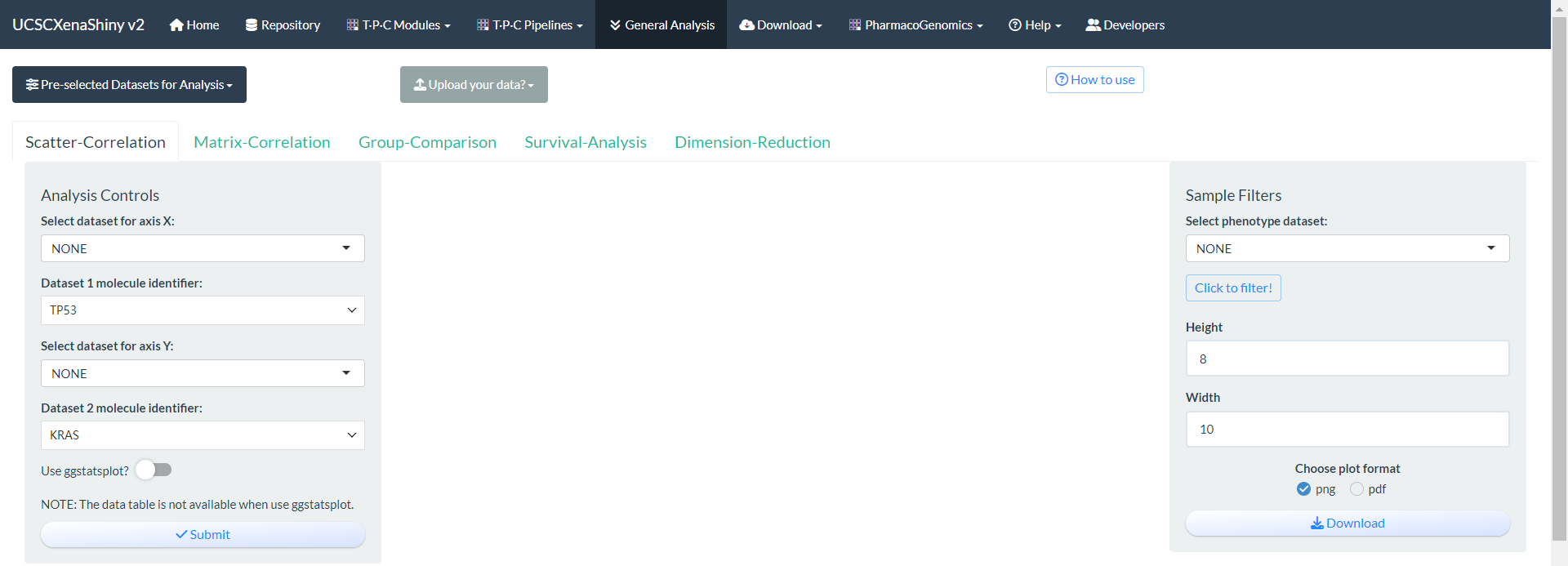
4. 其它功能
4.1 General Analysis
- 除了TPC数据以外,Xena平台还整理了其它很多队列的肿瘤组学数据。
General Analysis页面则允许用户以更通用的角度,探索Xena平台大部分的数据集。
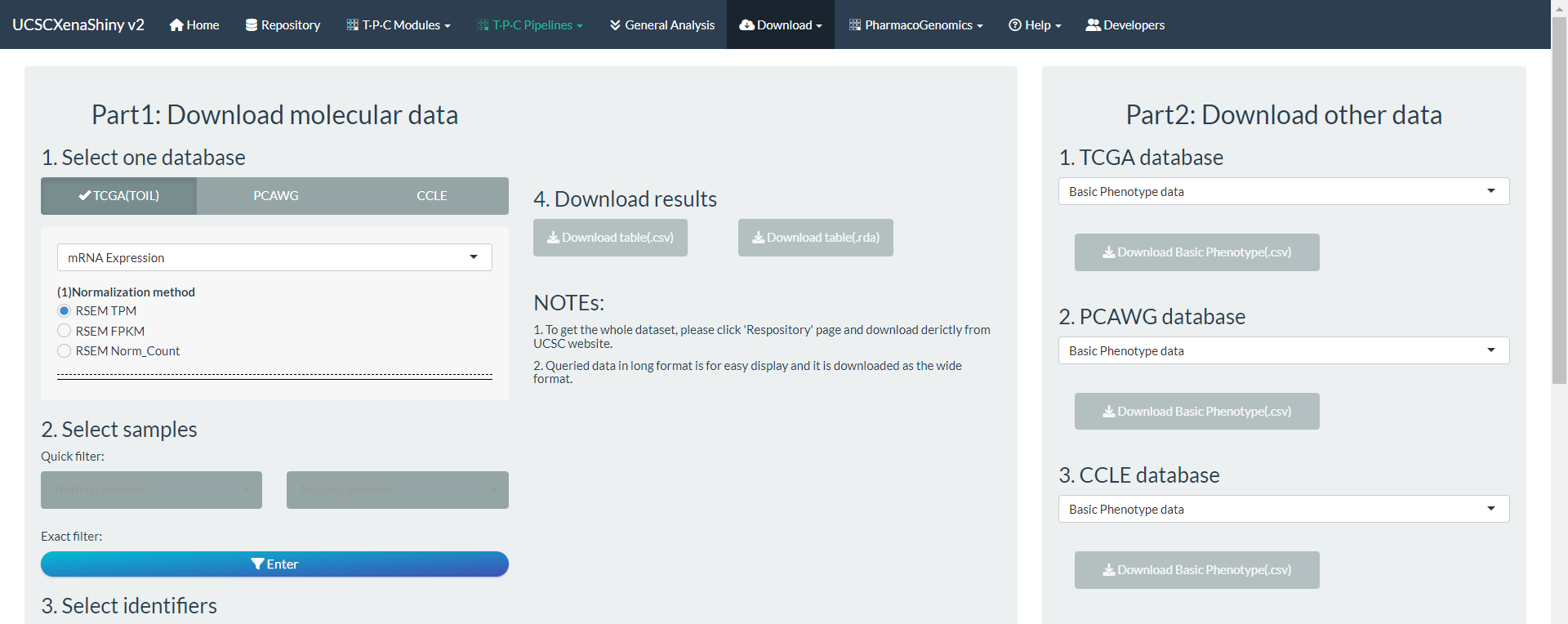
4.2 Download
除了分析功能以外,UCSCXenashiny提供了数据下载的功能。
Repository页面可以查询Xena大部分的数据集,以及多样的下载方式;Download页面可以帮助用户从特定数据集中,下载指定的一个或多个分子的数据。
4.3 PharmacoGenomics
- 此外,肿瘤细胞系的药物敏感性数据在治疗相关的分子机制研究和药物筛选中具有重要作用。
- 在V2版本的开发过程中,我们集成了彭宇中博士开发的OmicsPharDB工具 (https://github.com/mugpeng/OmicsPharDB)
- 其收集了来自GDSC1/2,CTRP1/2,PRISM,gCSI等药物数据,实现了多个具有特色的组学联合分析模块。
综上,简要介绍了UCSCXenaShiny v2版本更新后的使用方式与功能概述。我们的教程文档以及操作视频(https://lishensuo.github.io/UCSCXenaShiny_Book/)都提供了详细的帮助信息,使得用户可以快速上手。在后续,我们也会针对上述提到的每项功能,结合实例分享相关的使用经验。
最后,如果你在操作过程中,碰到意外的报错或Bug,欢迎随时通过Github或者邮件联系我们。