UCSCXenaShiny是基于集成了多种肿瘤数据库的UCSCXena平台,进行数据下载、分析、可视化的Shiny工具(以及同名R包),由上海科技大学王诗翔博士等共同开发;于2021年6月发表于Bioinformatics。下面主要学习其R包的相关函数,了解其核心功能。
1、数据关系#
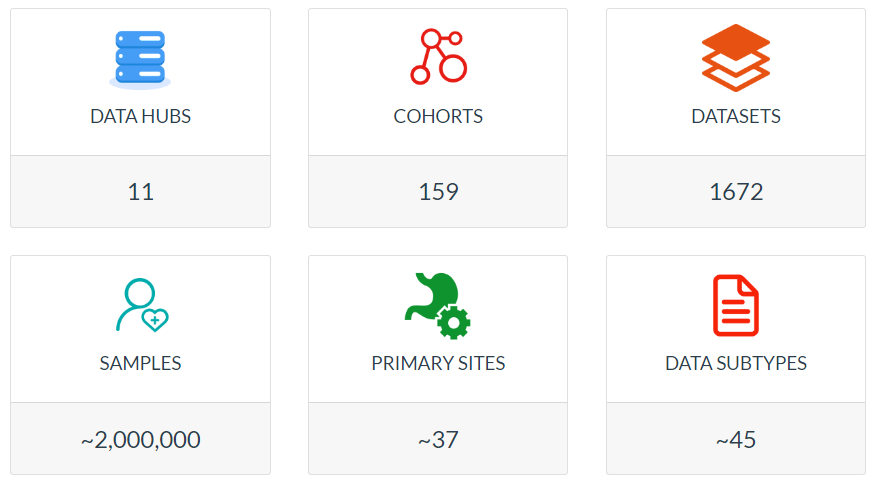
1.1 Hub/Cohort等
- Hub:不同的大型项目,例如TCGA、GTEx
- Cohort:不同的队列,可以表示一独立的研究,例如一种肿瘤
- Subtype:数据类型,例如表达矩阵/表型/突变数据
- Dataset:最终的数据实体,例如不同标准化方式的表达矩阵
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
|
data_stat = UCSCXenaTools::XenaGenerate()
## hosts → cohorts → datasets
data_hosts = data_stat@hosts # 11
data_cohorts = data_stat@cohorts # 159
data_datasets = data_stat@datasets # 1681
data_meta = UCSCXenaTools::XenaData
t(data_meta[1,])
# [,1]
# XenaHosts "https://ucscpublic.xenahubs.net"
# XenaHostNames "publicHub"
# XenaCohorts "Breast Cancer Cell Lines (Neve 2006)"
# XenaDatasets "ucsfNeve_public/ucsfNeveExp_genomicMatrix"
# SampleCount "51"
# DataSubtype "gene expression"
# Label "Neve Cell Line gene expression"
# Type "genomicMatrix"
# AnatomicalOrigin "Breast"
# SampleType "cell line"
# Tags "cell lines,breast cancer"
# ProbeMap "probeMap/affyU133_ucscGenomeBrowser_hg18.probeMap"
# LongTitle "Cell Line Gene Expression (Neve et al. Cancer Cell 2006)"
# Citation "Cancer Cell. 2006 Dec;10(6):515-27."
# Version "2011-11-01"
# Unit NA
# Platform NA
table(data_meta$Type)
# clinicalMatrix genomicMatrix genomicSegment mutationVector
# 345 981 152 203
|
clinicalMatrix主要包括survival data与phenotype data;
genomicMatrix包括所有可以表示为:行名是molecule,列名是sample的矩阵数据,例如基因表达矩阵等
genomicSegment主要针对CNV数据;mutationVector主要针对SNV数据
1.2 常用Hub
- TCGA与TARGET均是由NIH与NCI等机构完成,二者均涉及多种癌症的多组学分析,而后者更专注于少年儿童肿瘤研究;
- GTEx主要关注正常组织的相关测序数据;
- CCLE是针对癌症细胞系的多组学研究以及药物敏感性的相关数据;
- PCAWG是ICGC的子项目之一,也是一项大规模的癌症基因组学研究;
- TOIL是一个数据计算框架,对上述数据进行单独/整合分析,例如TCGA+TARGET+GETx

1.3 两个R包
UCSCXenaShiny网站主要以两个R包作为底层基础:
(1)UCSCXenaTools包是从UCSCXena下载数据/查询数据的工具包;
(2)UCSCXenaShiny包基于获取到的数据进行多种生信分析以及可视化。下面主要学习下这个R包的系列函数
2、UCSCXenaShiny包#
2.1 内置数据#
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
|
TCGA.organ # TCGA缩写
tcga_gtex # 样本所属的组织,分组(肿瘤/正常)
tcga_clinical # 肿瘤所属样本年龄性别,肿瘤分期等
tcga_subtypes # 肿瘤亚型
tcga_surv # 肿瘤的4种生存数据
## 肿瘤其它的不同属性
tcga_genome_instability
tcga_purity
tcga_tmb
ccle_info # 细胞系基本信息
toil_info # toil样本基本信息(TCGA+TARGET+GTEX)
pcawg_info #pcawg肿瘤基本信息(包括肿瘤分析,样本OS生存资料等)
## ccle、pcawg的肿瘤纯度信息
ccle_absolute
pcawg_purity
|
此外还有部分数据由于体量大等原因放在了Zenodo数据库,相关分析用到时可另行下载到R包安装路径的extdata文件夹内。
2.2 查询单个数据#
- 查询任一ense (Genomic) Matrix Dataset(上述1.1)的某个molecule/signature的样本(表达)数据
1
2
3
4
5
6
|
query_molecule_value()
query_molecule_value(dataset=, molecule=)
dataset <- "ccle/CCLE_copynumber_byGene_2013-12-03"
x <- query_molecule_value(dataset, "TP53")
head(x)
|
- 直接查询3大肿瘤Hub(toil/ccle/pcawg)中的某个molecule/signature的样本(表达)数据
1
2
3
4
5
6
7
8
9
|
query_pancan_value()
query_pancan_value(molecule=,
data_type= c("mRNA", "transcript", "protein", "mutation", "cnv", "cnv_gistic2",
"methylation", "miRNA", "fusion", "promoter", "APOBEC"),
database= c("toil", "ccle", "pcawg"))
query_pancan_value("KRAS")
query_pancan_value("KRAS", database = "ccle")
query_pancan_value("KRAS", database = "pcawg")
|
2.3 肿瘤相关可视化#
这是Shiny网页版的Quick PanCan Analysis模块的分析相关函数

(1)差异基因分析可视化
1
2
3
4
|
vis_toil_TvsN()
vis_toil_TvsN_cancer()
vis_pcawg_dist()
|
(2)基因表达相关性
1
2
3
4
5
6
|
vis_gene_cor() # TCGA
vis_gene_cor_cancer()
vis_ccle_gene_cor()
vis_pcawg_gene_cor()
|
(3)基因表达与肿瘤指标的相关性
1
2
3
4
5
|
vis_gene_TIL_cor()
vis_gene_immune_cor()
vis_gene_stemness_cor()
vis_gene_tmb_cor()
vis_gene_msi_cor()
|
(4)生存相关分析
1
2
3
4
5
6
7
8
9
10
11
12
|
# survival分析
## step1:获取数据:分子表达+生存资料
sur_data = tcga_surv_get(item=, TCGA_cohort=, profile=)
sur_data = tcga_surv_get("TP53", "BRCA", "mRNA")
## step2 绘图可视化:默认取最佳分组,可自定义阈值
tcga_surv_plot(
sur_data,
time = "OS.time", status = "OS")
# 单变量Cox回归
vis_unicox_tree()
vis_pcawg_unicox_tree()
|
(5)药物敏感度相关分析
1
2
3
4
5
6
7
|
# 在多种肿瘤细胞系中,基因表达与药物敏感性的相关性分析
analyze_gene_drug_response_asso("TP53")
vis_gene_drug_response_asso("TP53")
# 在多种肿瘤细胞系中,根据某基因高低表达分组,药物敏感性的差异
data = analyze_gene_drug_response_diff("TP53")
vis_gene_drug_response_diff("TP53")
|
共涉及20余个药物。
2.4 一般分析#
这是Shiny网页版的General Analysis模块的分析相关函数

1
2
3
4
5
6
7
8
9
10
11
|
# 两两相关性可视化(点图)
vis_identifier_cor()
# 多个间相关性(热图)
vis_identifier_multi_cor()
# 分组表达差异
vis_identifier_grp_comparison()
# 分组生存分析
vis_identifier_grp_surv()
|



