ConsensusClusterPlus包是肿瘤分型研究的常用工具,其于2010年发表于Bioinformatics。
- Paper:https://academic.oup.com/bioinformatics/article/26/12/1572/281699
- Tutorial:https://bioconductor.org/packages/release/bioc/vignettes/ConsensusClusterPlus/inst/doc/ConsensusClusterPlus.pdf
|
|
1、示例表达矩阵
- 行名为基因,列名为样本的基因表达矩阵;根据需要,进行标准化处理
- 基因集的选择是关键的一步,可根据统计学(高变基因)或者生物学(特定功能相关基因)进行选择
- 如下是参考教程的数据
|
|
2、样本亚型鉴定
- 实际就是一个函数即可:
ConsensusClusterPlus() - 其中涉及到较多参数的选择,具体如下
|
|
- R对象结果:list格式
|
|
- 图形输出结果,主要两类图
(1)特定聚类结果的热图

(2)所有聚类结果的consensus value累计分布图
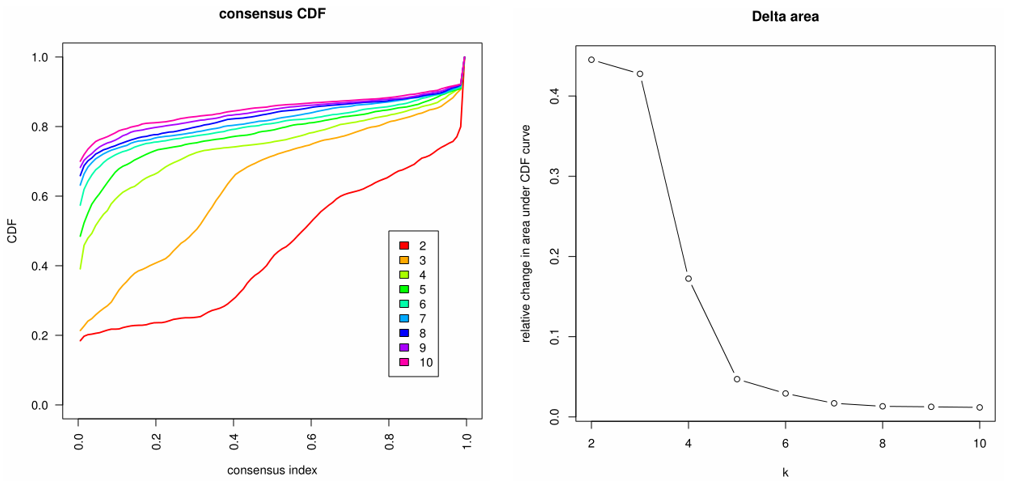
根据上图结果,可以优先考虑分为4~6类亚型的结果。