1、HPA
- HPA:the Human Protein Atlas,由2003年来自瑞典的科研机构发起,旨在绘制综合性人类蛋白质图谱。
- https://www.proteinatlas.org/
- 蛋白质表达数据库,常见用途包括:
- (1)查看特定基因在不同组织、脑区,细胞类型,组织细胞类型,疾病(癌症),免疫细胞,肿瘤细胞系等表达情况。
- (2)数据挖掘类文章常使用其进行比较基因在肿瘤部位与相应正常部位的蛋白水平表达差异。
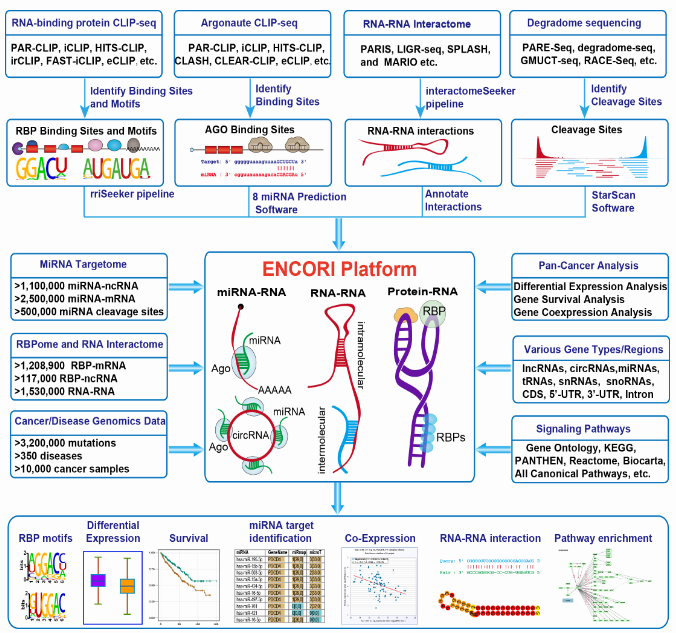
2、ENCORI
- ENCORI,The Encyclopedia of RNA Interactomes,由中山大学生命科学学院屈良鹄团队开发,于2014年发表于Nucleic Acids Res。
- https://starbase.sysu.edu.cn/index.php
- miRNA、lncRNA、RBP等多维相互作用网络,常见用途包括
- miRNA/RBP的靶标查询(mRNA,lncRNA…)
- RNA interaction, ceRNA network
- TCGA肿瘤的差异表达,生存分析,相关性等
3、TCIA
- TCIA,The Cancer Immunome Database,由来自奥地利的因斯布鲁克大学医学院Zlatko Trajanoski团队开发,于2017年发表于Cell Reports
- https://tcia.at/home
- 20种solid cancer的免疫相关分析,例如
- 免疫基因表达、细胞浸润、肿瘤亚克隆等
- 亮点之一是提出Immunophenoscore指标用于预测免疫治疗响应
- 样本临床信息也整理的较为完整
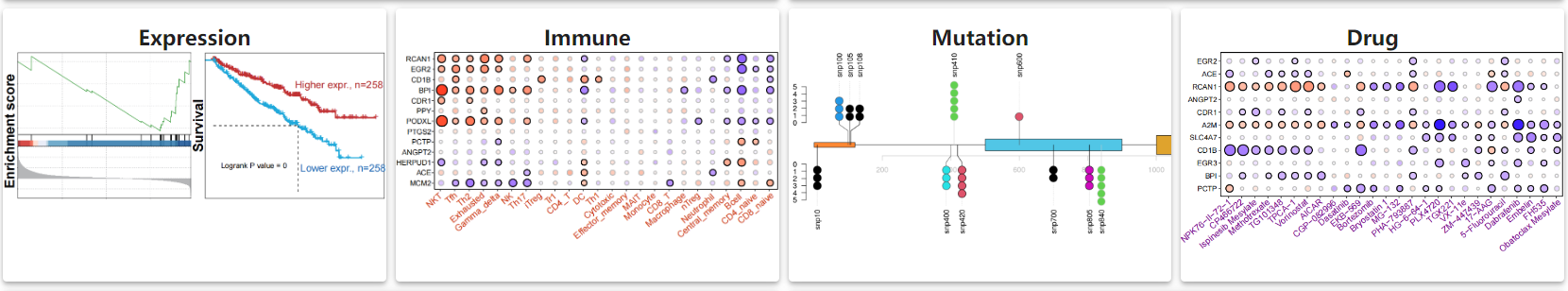
4、GSCA
- GSCA,Gene Set Cancer Analysis,由来自华中科技大学的郭安源团队开发/更新,于2022年11月发表于Briefings in Bioinformatics
- http://bioinfo.life.hust.edu.cn/GSCA/#/
- 单基因/基因集与33种癌症在基因表达、免疫浸润、突变变异、药物敏感度等4个方面的综合分析平台
- 基因集分析主要通过GSVA/GSEA两种方式进行打分,再与临床信息相关联
- 比较适合基因/基因集的泛癌分析,出图较为精美
- 提供原始数据可供下载
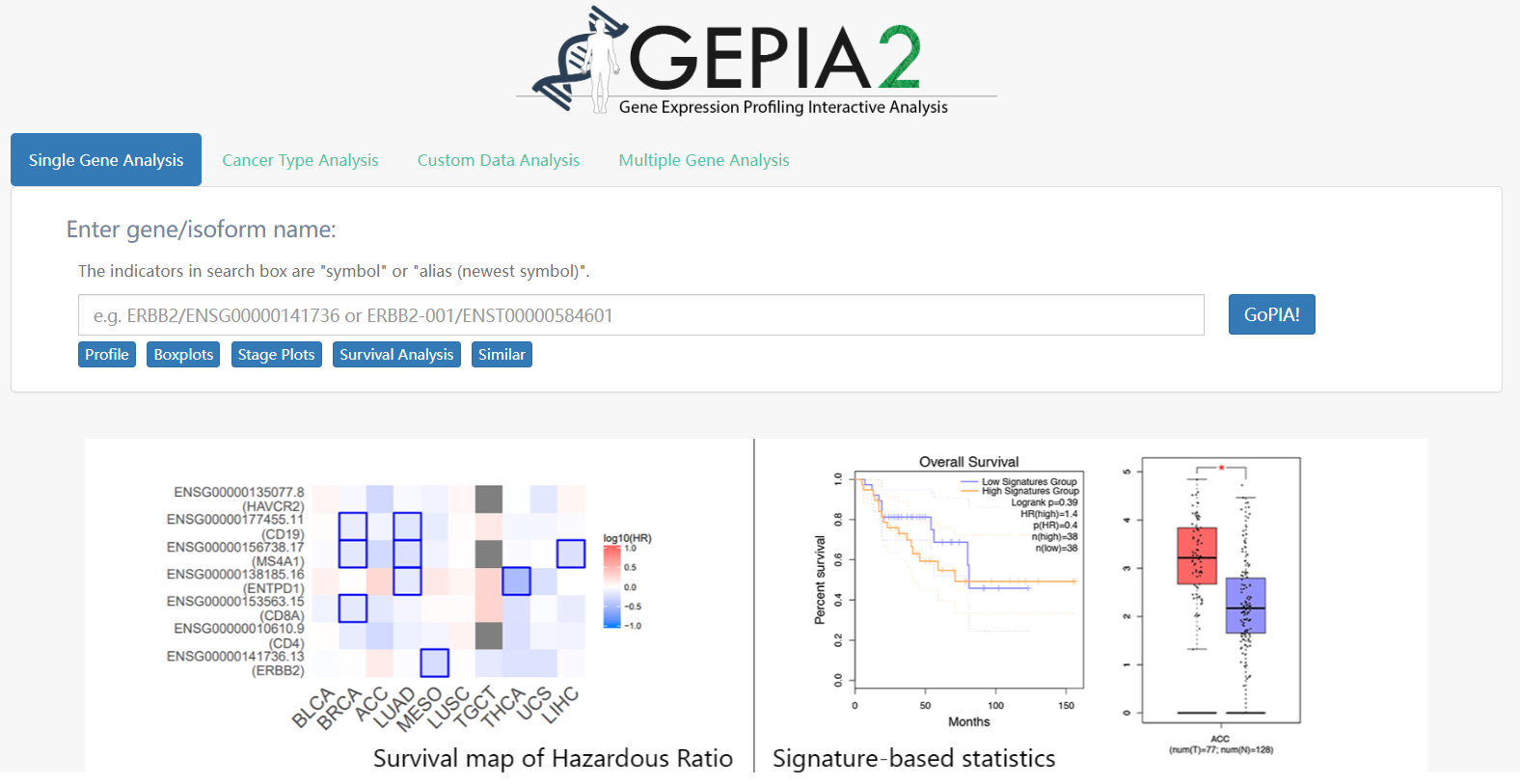
5、GEPIA2
- Gene Expression Profiling Interactive Analysis (V2),由北京大学张泽民研究团队开发,于2019年7月发表于Nucleic Acids Research
- http://gepia2.cancer-pku.cn/
- 主要功能在基因在多肿瘤/正常组织(TCGA+GTEx)的常规分析
- 基因为主体
- 差异分析、生存分析
- 基因对相关性分析/基因最相关基因分析
- 特定基因集降维信息
- 肿瘤为主体
- 差异基因集、生存相关基因集
- 基因为主体