生存分析(survival analysis)的主要目的是发现与患者生存事件相关的指标因素,例如年龄性别、基因表达/突变等。如下学习相关基础知识及几种常见的生存分析方法。
参考教程:http://www.sthda.com/english/wiki/survival-analysis-basics

1、基础知识#
(1)event:表示“生存”结束的事件,最常见的形式是患者死亡,或者疾病复发等。
(2)survival time:从开始记录(通常是初次诊断患病),到event事件发生的时间。
- survival time的长短直接取决于对于事件的定义方法。
- 在UCSC xena整理的TCGA数据库中收集了4种事件定义方法,如下表所示。
|
开始日期 |
结束日期(事件发生) |
特征 |
| OS (overall survial) |
初次诊断 |
患者死亡(任何原因) |
longest |
| DSS (disease-specific survival) |
初次诊断 |
因该病导致死亡 |
/ |
| PFI (progression-free interval) |
初次诊断 |
经治疗后,该疾病首次恶化或导致患者死亡 |
shortest |
| DFI (disease-free interval) |
初次诊断 |
经治疗未发现肿瘤后,又复发或导致患者死亡 |
/ |
[1] PFI里的恶化可包括疾病严重/局部区域复发/远处转移/新生肿瘤等指标。PFS(progression-free survival)指标与PFI基本类似,区别在于不关注死亡原因。
[2] DFI里的复发可包括原位复发、远处转移、新生肿瘤(同器官)。DFS(disease-free survival)指标与DFI基本类似,区别在于同样不关注死亡原因。
参考来源:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6066282/ Definitions and derivation of clinical survival outcome endpoints部分
OS/PFS/PFI/DFS/DFI/DSS各种生存指标定义 – 王进的个人网站 (jingege.wang)
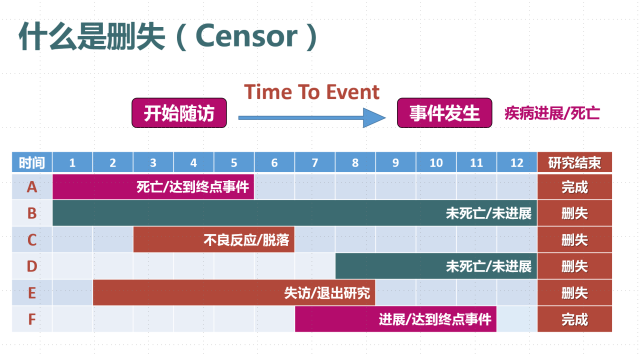
(3)Censor 删失值
在随访的过程中,可能无法全部病人都可以收集到准确地生存时间。
- 一方面由于随访项目时间限制,直至随访结束,病人仍在世;
- 一方面可能在随访期间,病人失联、或者因其它原因去世等,无法继续随访。
这样的数据就称之为Censor,而此时的生存时间即为所能记录到的最长生存时间。病人的status要么是事件发生了,要么就是Censor。
2、分析包与示例数据#
(1)分析R包
1
2
3
|
# install.packages(c("survival", "survminer"))
library("survival") #生存分析
library("survminer") #结果可视化
|
(2)示例数据 lung {survival}
- 228个被诊断患有肺癌的病人开始长期的随访
- 如果病人死亡;则随访结束,并记录生存时间(从确诊到死亡的时间)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
|
head(lung)
# inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
# 1 3 306 2 74 1 1 90 100 1175 NA
# 2 3 455 2 68 1 0 90 90 1225 15
# 3 3 1010 1 56 1 0 90 90 NA 15
# 4 5 210 2 57 1 1 90 60 1150 11
# 5 1 883 2 60 1 0 100 90 NA 0
# 6 12 1022 1 74 1 1 50 80 513 0
# inst: Institution code
# time: Survival time in days(*) 生存时间
# status: censoring status 1=censored, 2=dead(*) 事件是否发生
# age: Age in years
# sex: Male=1 Female=2
# ph.ecog: ECOG performance score (0=good 5=dead)
# ph.karno: Karnofsky performance score (bad=0-good=100) rated by physician
# pat.karno: Karnofsky performance score as rated by patient
# meal.cal: Calories consumed at meals
# wt.loss: Weight loss in last six months
|
如果挖掘TCGA的数据,通常1表示事件发生,0表示censored
3、Log-rank test分析#
(1)对于一种表型因素将患者分成两组,分析是否具有显著生存差异;
- 性别、基因突变、疾病阶段等可根据表型特性分组;
- 年龄、基因表达等可设置特定阈值进行分组。
(2)survfit()分析,后续可对其结果进行绘图:
n表示每组的病人数;events表示每组有多少病人死亡;median 表示中位生存率所对应的生存时间;- 最后两列表示第三列的95%置信区间。
1
2
3
4
5
6
7
8
9
10
11
|
fit <- survfit(Surv(time, status) ~ sex, data = lung)
print(fit)
# Call: survfit(formula = Surv(time, status) ~ sex, data = lung)
#
# n events median 0.95LCL 0.95UCL
# sex=1 138 112 270 212 310
# sex=2 90 53 426 348 550
res.sum <- surv_summary(fit)
head(res.sum[res.sum$sex==1,])
head(res.sum[res.sum$sex==2,])
|
(3)survdiff() 分析显著性P值
1
2
3
4
5
6
7
8
9
10
11
12
13
14
|
surv_diff <- survdiff(Surv(time, status) ~ sex, data = lung)
# Call:
# survdiff(formula = Surv(time, status) ~ sex, data = lung)
#
# N Observed Expected (O-E)^2/E (O-E)^2/V
# sex=1 138 112 91.6 4.55 10.3
# sex=2 90 53 73.4 5.68 10.3
#
# Chisq= 10.3 on 1 degrees of freedom, p= 0.001
#虽然如上结果是有P值的,但是尝试之后发现从对象中只能提取chisq的值,然后再进一步转换
p.val = 1 - pchisq(surv_diff$chisq, length(surv_diff$n) - 1)
p.val
# [1] 0.001311165
|
(4)最优分组阈值 ☆
- 对于连续型变量(例如基因表达),需要手动设置一个阈值对样本进行二分组(或多分组),再进行生存分析。最常见的选择是中位数。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
### 以其中的年龄为例
median_cut = median(lung$age)
# 57
lung$age_group = ifelse(lung$age > median_cut,
"Old", "Young")
table(lung$age_group)
# Old Young
# 111 117
surv_diff <- survdiff(Surv(time, status) ~ age_group, data = lung)
p.val = 1 - pchisq(surv_diff$chisq, length(surv_diff$n) - 1)
p.val
# [1] 0.1702206
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
|
# 使用survminer包的surv_cutpoint函数
res.cut <- surv_cutpoint(lung, time = "time", event = "status",
variables = c("age"))
summary(res.cut)
# cutpoint statistic
# age 70 2.013619
# best cut
best_cut = summary(res.cut)["age","cutpoint"]
# [1] 70
lung$age_group_best = ifelse(lung$age > best_cut,
"Old", "Young")
table(lung$age_group_best)
# Old Young
# 46 182
surv_diff <- survdiff(Surv(time, status) ~ age_group_best, data = lung)
p.val = 1 - pchisq(surv_diff$chisq, length(surv_diff$n) - 1)
p.val
# [1] 0.0312283
# 这里的p值结果相较上述的中位数分析更为显著
|
4、Log-rank test可视化#
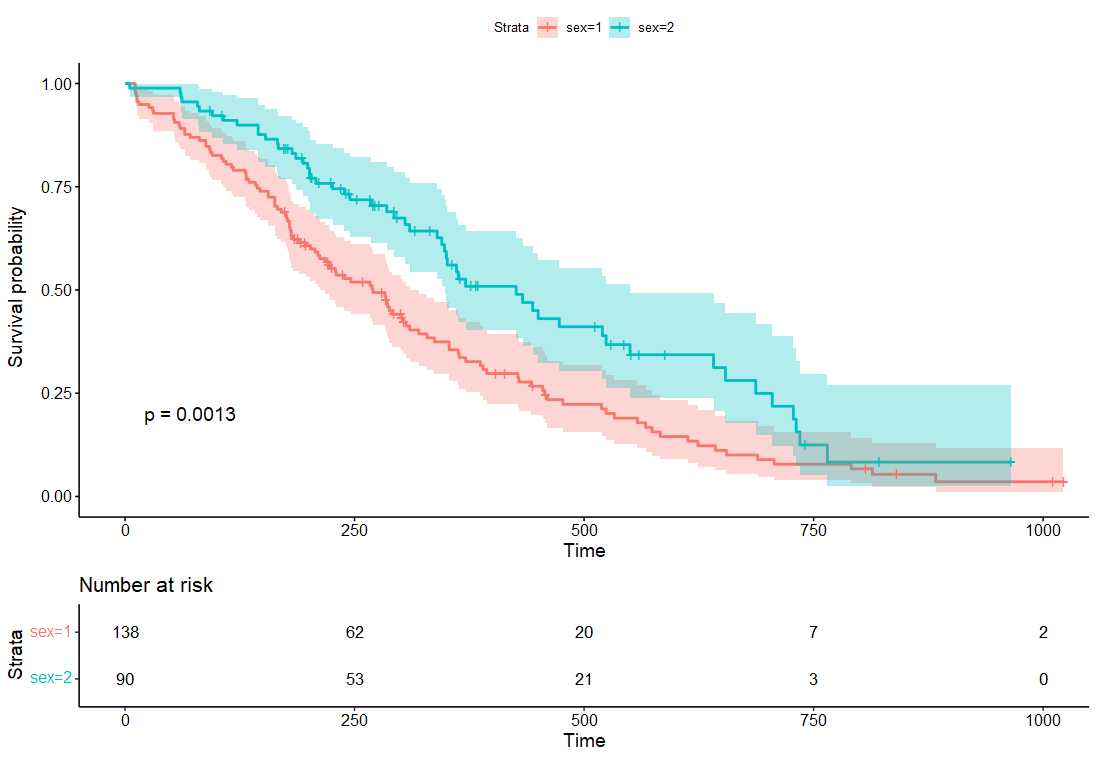
(1)绘制生存曲线,如下图所示默认绘制两个部分
- 上图:时间对应的生存率的生存曲线图,曲线中的短竖线表示在时间点有缺失值病人。不同颜色表示不同分组,阴影部分表示95%置信区间;左下角p值表示基于log-rank test计算得到的P值。
- 下表:不同时间点对应每组尚未发生事件(死亡)的数目。
1
2
3
|
ggsurvplot(fit,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE)
|
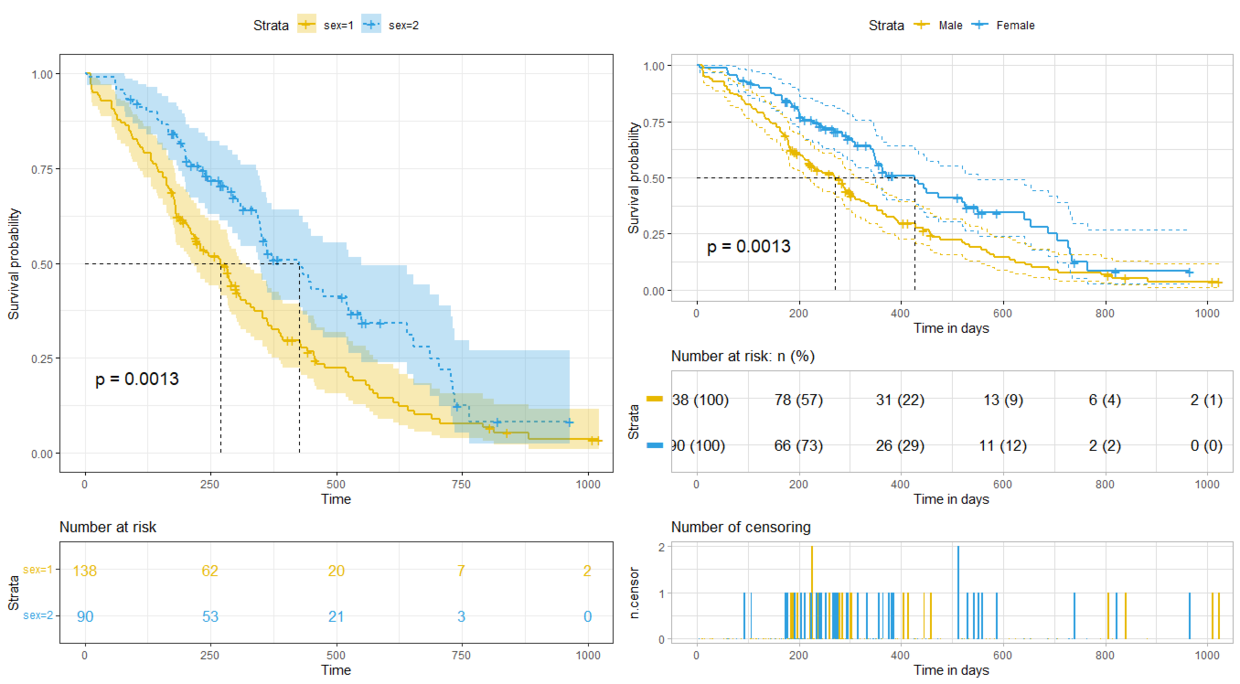
(2)可调整的图形参数(结合具体代码理解)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
|
p1 = ggsurvplot(
fit,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE, # Add risk table
risk.table.col = "strata", # Change risk table color by groups
linetype = "strata", # Change line type by groups
surv.median.line = "hv", # Specify median survival
ggtheme = theme_bw(), # Change ggplot2 theme
palette = c("#E7B800", "#2E9FDF")
)
p2 = ggsurvplot(
fit, # survfit object with calculated statistics.
pval = TRUE, # show p-value of log-rank test.
conf.int = TRUE, # show confidence intervals for
# point estimaes of survival curves.
conf.int.style = "step", # customize style of confidence intervals
xlab = "Time in days", # customize X axis label.
break.time.by = 200, # break X axis in time intervals by 200.
ggtheme = theme_light(), # customize plot and risk table with a theme.
risk.table = "abs_pct", # absolute number and percentage at risk.
risk.table.y.text.col = T,# colour risk table text annotations.
risk.table.y.text = FALSE,# show bars instead of names in text annotations
# in legend of risk table.
ncensor.plot = TRUE, # plot the number of censored subjects at time t
surv.median.line = "hv", # add the median survival pointer.
legend.labs = c("Male", "Female"), # change legend labels.
palette = c("#E7B800", "#2E9FDF") # custom color palettes.
)
arrange_ggsurvplots(list(p1,p2))
|
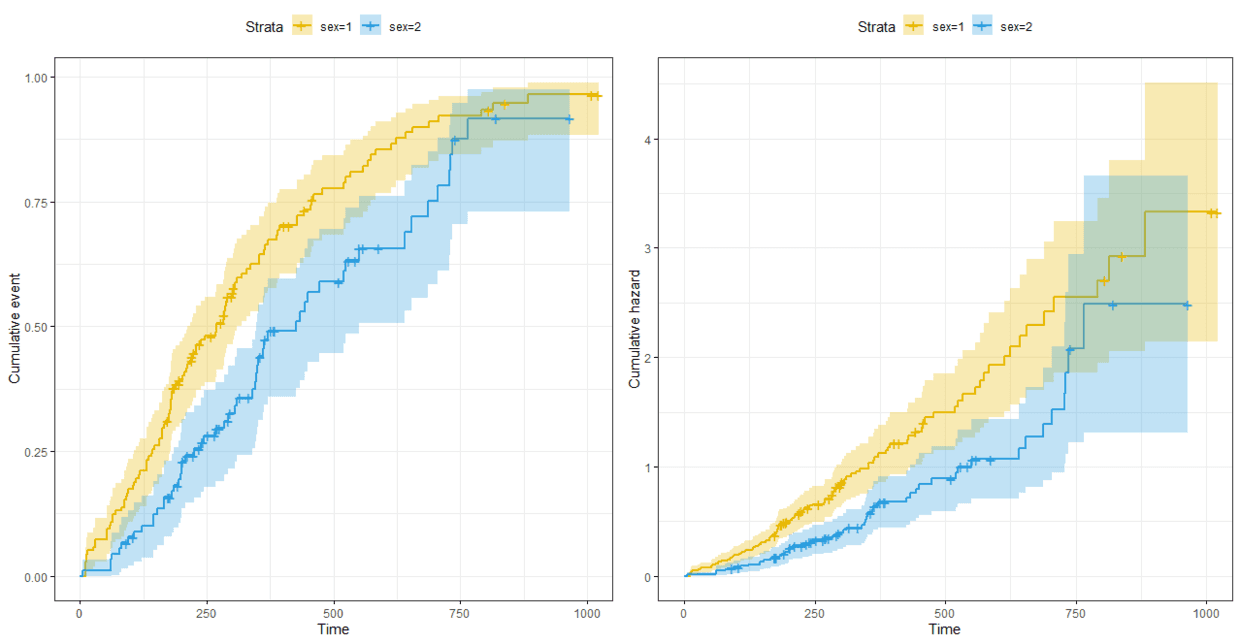
(3)如上是经典的生存曲线,同时也支持绘制其它类型曲线
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
|
# 参数`fun="event"`,表示cumulative event事件累计发生率
p3=ggsurvplot(
fit,
conf.int = TRUE,
risk.table.col = "strata", # Change risk table color by groups
ggtheme = theme_bw(), # Change ggplot2 theme
palette = c("#E7B800", "#2E9FDF"),
fun = "event"
)
# 参数`fun="cumhaz"`,表示cummulative hazard表示累计风险水平(在时刻t,事件发生的可能性)
p4=ggsurvplot(
fit,
conf.int = TRUE,
risk.table.col = "strata", # Change risk table color by groups
ggtheme = theme_bw(), # Change ggplot2 theme
palette = c("#E7B800", "#2E9FDF"),
fun = "cumhaz"
)
arrange_ggsurvplots(list(p3,p4))
|
5、比例风险模型分析#
比例风险模型又称Cox Proportional-Hazards Model,用于量化每个表型对于事件发生的影响大小。
- 表型可以是分类变量或者是连续变量;
- 衡量影响大小的指标称为Hazard rate(风险因子),简称HR
- Hazard ratio(HR)=exp(coef)
- HR<1(coef<0) 表明负相关—该变量值越大,事件发生风险越低,生存率越高;
- HR>1(coef>0) 表明正相关—该变量值越大,事件发生风险越高,生存率越小。
(1) 单变量Cox回归
1
2
3
4
5
6
7
8
9
10
11
12
13
|
res.cox <- coxph(Surv(time, status) ~ sex, data = lung)
res.cox
# Call:
# coxph(formula = Surv(time, status) ~ sex, data = lung)
#
# coef exp(coef) se(coef) z p
# sex -0.5310 0.5880 0.1672 -3.176 0.00149
#
# Likelihood ratio test=10.63 on 1 df, p=0.001111
# n= 228, number of events= 165
exp(res.cox$coefficients)
# sex
# 0.5880028
|
(2)对多个变量分别进行单变量Cox回归
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
|
#分别回归
covariates <- c("age", "sex", "ph.karno", "ph.ecog", "wt.loss")
univ_formulas <- sapply(covariates,
function(x) as.formula(paste('Surv(time, status)~', x)))
univ_models <- lapply( univ_formulas, function(x){coxph(x, data = lung)})
#提取各个变量的回归结果
univ_results <- lapply(univ_models,
function(x){
x <- summary(x)
p.value<-signif(x$wald["pvalue"], digits=2)
wald.test<-signif(x$wald["test"], digits=2)
beta<-signif(x$coef[1], digits=2);#coeficient beta
HR <-signif(x$coef[2], digits=2);#exp(beta)
HR.confint.lower <- signif(x$conf.int[,"lower .95"], 2)
HR.confint.upper <- signif(x$conf.int[,"upper .95"],2)
HR <- paste0(HR, " (",
HR.confint.lower, "-", HR.confint.upper, ")")
res<-c(beta, HR, wald.test, p.value)
names(res)<-c("beta", "HR (95% CI for HR)", "wald.test",
"p.value")
return(res)
#return(exp(cbind(coef(x),confint(x))))
})
res <- t(as.data.frame(univ_results, check.names = FALSE))
as.data.frame(res)
# beta HR (95% CI for HR) wald.test p.value
# age 0.019 1 (1-1) 4.1 0.042
# sex -0.53 0.59 (0.42-0.82) 10 0.0015
# ph.karno -0.016 0.98 (0.97-1) 7.9 0.005
# ph.ecog 0.48 1.6 (1.3-2) 18 2.7e-05
# wt.loss 0.0013 1 (0.99-1) 0.05 0.83
|
(3)多变量Cox回归(多元回归)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
|
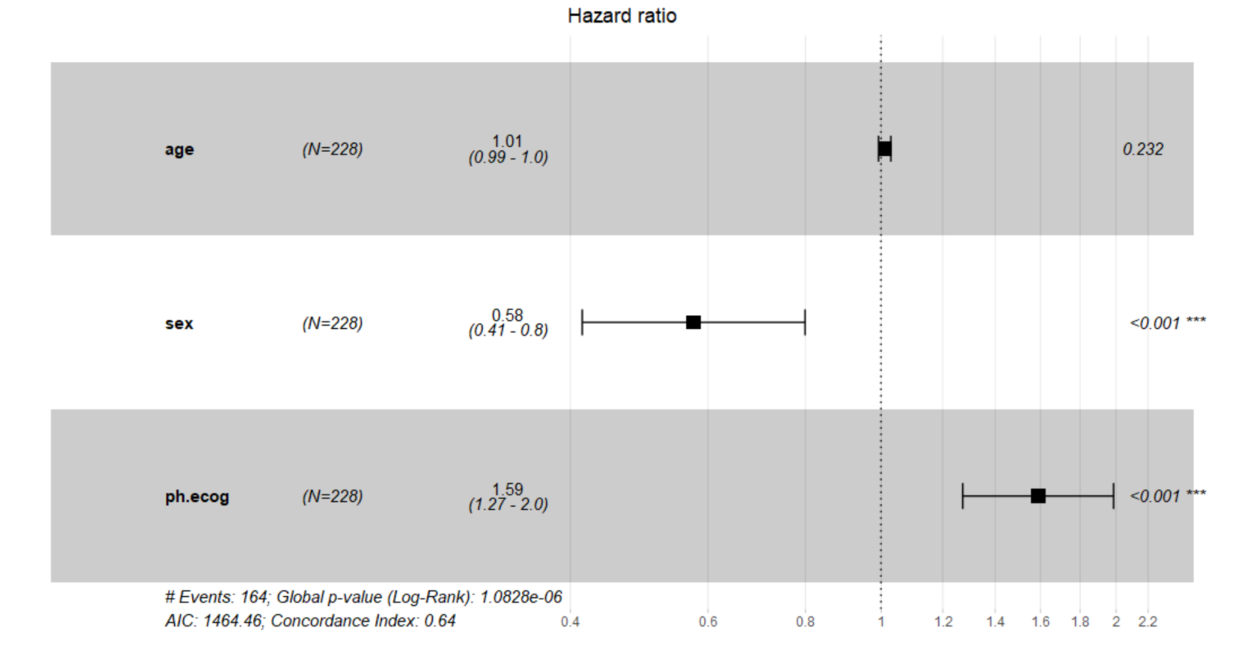
res.cox <- coxph(Surv(time, status) ~ age + sex + ph.ecog, data = lung)
summary(res.cox)
# Call:
# coxph(formula = Surv(time, status) ~ age + sex + ph.ecog, data = lung)
#
# n= 227, number of events= 164
# (因为不存在,1个观察量被删除了)
#
# coef exp(coef) se(coef) z Pr(>|z|)
# age 0.011067 1.011128 0.009267 1.194 0.232416
# sex -0.552612 0.575445 0.167739 -3.294 0.000986 ***
# ph.ecog 0.463728 1.589991 0.113577 4.083 4.45e-05 ***
# ---
# Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
#
# exp(coef) exp(-coef) lower .95 upper .95
# age 1.0111 0.9890 0.9929 1.0297
# sex 0.5754 1.7378 0.4142 0.7994
# ph.ecog 1.5900 0.6289 1.2727 1.9864
#
# Concordance= 0.637 (se = 0.025 )
# Likelihood ratio test= 30.5 on 3 df, p=1e-06
# Wald test = 29.93 on 3 df, p=1e-06
# Score (logrank) test = 30.5 on 3 df, p=1e-06
ggforest(res.cox, data = lung,
main = "Hazard ratio",
cpositions = c(0.10, 0.22, 0.4),
fontsize = 1.0)
|
6、cox模式lasso回归#
1
2
|
install.packages("glmnet")
library(glmnet)
|
(1)示例数据
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
|
library(survival)
data(CoxExample)
x <- CoxExample$x
y <- CoxExample$y
## 标签数据(生存资料)
dim(y)
# [1] 1000 2
head(y)
# time status
# [1,] 1.76877757 1
# [2,] 0.54528404 1
# [3,] 0.04485918 0
## 特征数据(类似基因表达数据)
dim(x)
# [1] 1000 30
x[1:4,1:4]
# [,1] [,2] [,3] [,4]
# [1,] -0.8767670 -0.6135224 -0.5675738 0.6621599
# [2,] -0.7463894 -1.7519457 0.2854590 1.1392105
# [3,] 1.3759148 -0.2641132 0.8872741 0.3841870
# [4,] 0.2375820 0.7859162 -0.8967028 -0.8339338
|
(2)lasso分析
1
2
3
4
5
6
|
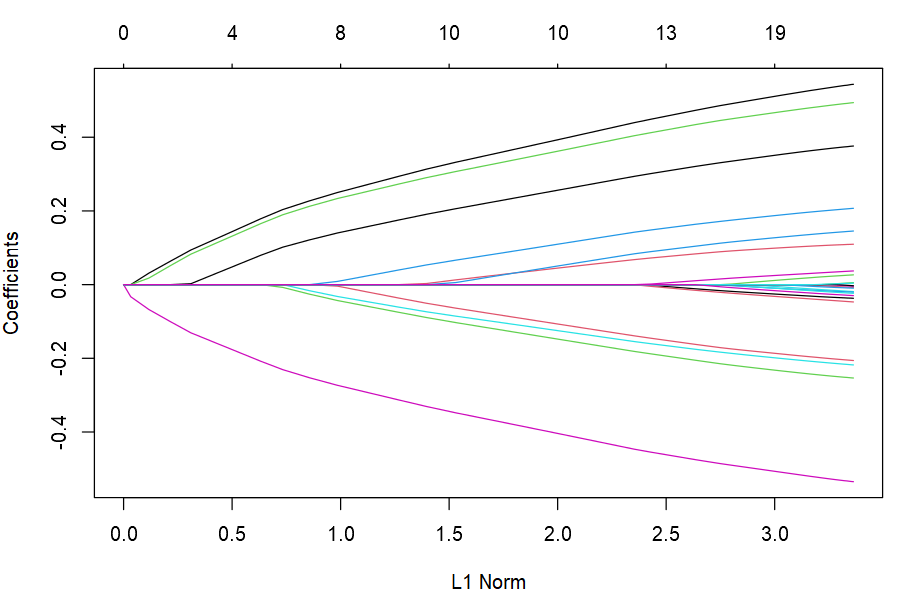
fit <- glmnet(x, y, family = "cox")
print(fit)
plot(fit, xvar = "norm", label = FALSE) # default
# plot(fit, xvar = "lambda", label = FALSE)
# plot(fit, xvar = "dev", label = FALSE)
|
如下图所示:
- 下轴:不同的L1 norm惩罚值;
- 上轴:在相应L1 norm惩罚值里,当前系数非0的变量个数;
- 左轴:变量的系数值
(3)交叉验证
在cox模式的lasso回归交叉验证中,支持两类评价指标
deviance:为默认指标;该值越小,模型预测效果越好C:即C-index,类似AUC;该值越大,模型预测效果越好
1
2
3
4
|
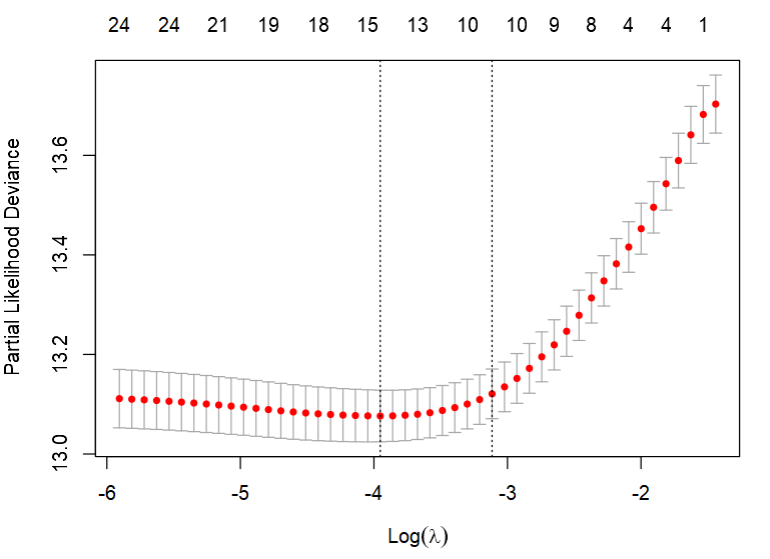
set.seed(1)
cvfit <- cv.glmnet(x, y, family = "cox", type.measure = "deviance")
# cvfit <- cv.glmnet(x, y, family = "cox", type.measure = "C")
plot(cvfit)
|
如下图,两条虚线分别表示两个λ值
- 左边的虚线→
lambda.min :表示评价指标最优的λ取值;
- 右边的虚线→
lambda.1se:表示lambda.min的一倍标准差下对模型更为严格惩罚的λ取值。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
|
cvfit$lambda.min
# [1] 0.01920429
cvfit$lambda.1se
# 1] 0.04436439
coef(cvfit, s = cvfit$lambda.min)
library(tidyverse)
coef(cvfit, s="lambda.min") %>%
as.matrix() %>% as.data.frame() %>%
tibble::rownames_to_column(var = "mRNA") %>%
dplyr::rename(coef=`1`) %>%
dplyr::filter(coef!=0)
# mRNA coef
# 1 V1 0.47972069
# 2 V2 -0.16674492
# 3 V3 -0.21014303
# 4 V4 0.16781912
# 5 V5 -0.17951289
# 6 V6 -0.48058218
# 7 V7 0.32603876
# 8 V8 0.08646499
# 9 V9 0.44014703
# 10 V10 0.10854409
# 11 V13 0.01304299
# 12 V17 -0.01388096
# 13 V25 -0.01830447
# 14 V30 -0.00301953
|







