scCODA(single-cell compositional data analysis)是由德国环境健康研究中心计算生物学研究所M Büttner等人基于python开发的单细胞数据分析工具,于2021年11月发表于Nature Communication;主要用于分析不同分组样本的细胞组成的差异。参考官方文档记录用法如下。
- Paper:https://www.nature.com/articles/s41467-021-27150-6
- Github:https://github.com/theislab/scCODA
- Tutorial:https://sccoda.readthedocs.io/en/latest/index.html
1、安装环境#
1
2
3
4
5
|
conda create -n sccoda python=3.9
conda activate sccoda
conda install rpy2
pip install sccoda
# conda install -c conda-forge notebook
|
2、分析流程#
1
2
3
4
5
6
7
8
9
10
11
12
13
|
import importlib
import warnings
warnings.filterwarnings("ignore")
import pandas as pd
import pickle as pkl
import matplotlib.pyplot as plt
from sccoda.util import comp_ana as mod
from sccoda.util import cell_composition_data as dat
from sccoda.util import data_visualization as viz
import sccoda.datasets as scd
|
- (2)读取数据
- pandas.Dataframe:第一列为样本名,其余每列各代表一种细胞类型,值表示细胞数量
- 使用scanny包转换为Anndata结构格式,obs表示样本信息
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
|
## 导入示例数据
cell_counts = scd.haber()
print(cell_counts)
# Mouse Endocrine Enterocyte Enterocyte.Progenitor Goblet Stem TA TA.Early Tuft
# 0 Control_1 36 59 136 36 239 125 191 18
# 1 Control_2 5 46 23 20 50 11 40 5
# 2 Control_3 45 98 188 124 250 155 365 33
data_all = dat.from_pandas(cell_counts, covariate_columns=["Mouse"])
data_all.obs
data_all.X
## 提取分组信息
data_all.obs["Condition"] = data_all.obs["Mouse"].str.replace(r"_[0-9]", "", regex=True)
# Mouse Condition
# 0 Control_1 Control
# 1 Control_2 Control
# 2 Control_3 Control
data_salm = data_all[data_all.obs["Condition"].isin(["Control", "Salm"])]
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
|
## 设置先验信息
model_salm = mod.CompositionalAnalysis(data_salm,
formula="Condition", #指定参考组
reference_cell_type="Goblet") #指定一种细胞类型作为已知组间比例不变的标准,如不确定设置为 "automatic"
## Markov-chain Monte Carlo (MCMC) inferrence
sim_results = model_salm.sample_hmc() # time consuming
# 备选方案:sample_hmc_da(), sample_nuts()
## 分析结果
# sim_results.set_fdr(est_fdr=0.4)
sim_results.summary()
sim_results.credible_effects()
sim_results.effect_df
|
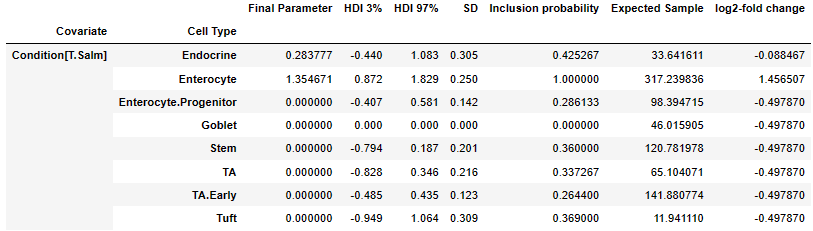
如上重点关注effect的Final Parameter列
- 若为0表示,该细胞类型比例在组间差异不大,可设置est_fdr设置判断的阈值标准
- 大于0,则表示相对于参考组,细胞比例提高;反之,则相反。
3、可视化#
此外scCODA也提供了一些可视化细胞比例的绘图函数,简单示例如下。
1
|
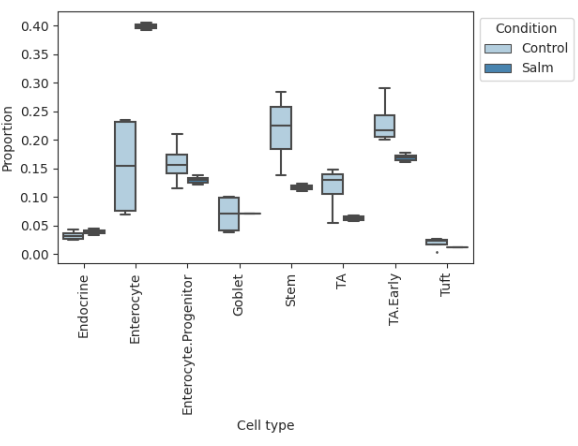
viz.boxplots(data_salm, feature_name="Condition")
|
1
|
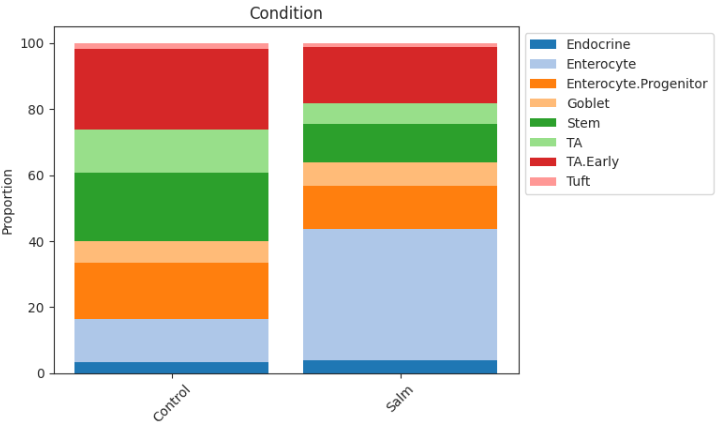
viz.stacked_barplot(data_salm, feature_name="Condition")
|