KNN–K近邻
1、KNN的步骤
- (1)计算输入数据与训练数据的距离(一般欧几里得距离);
- (2)从训练集中,选取距离输入数据点最近的k个数据;
- (3)对于分类任务【常见】,取这k个训练数据类别的众数;对于回归任务,取这k个训练数据值的平均数。
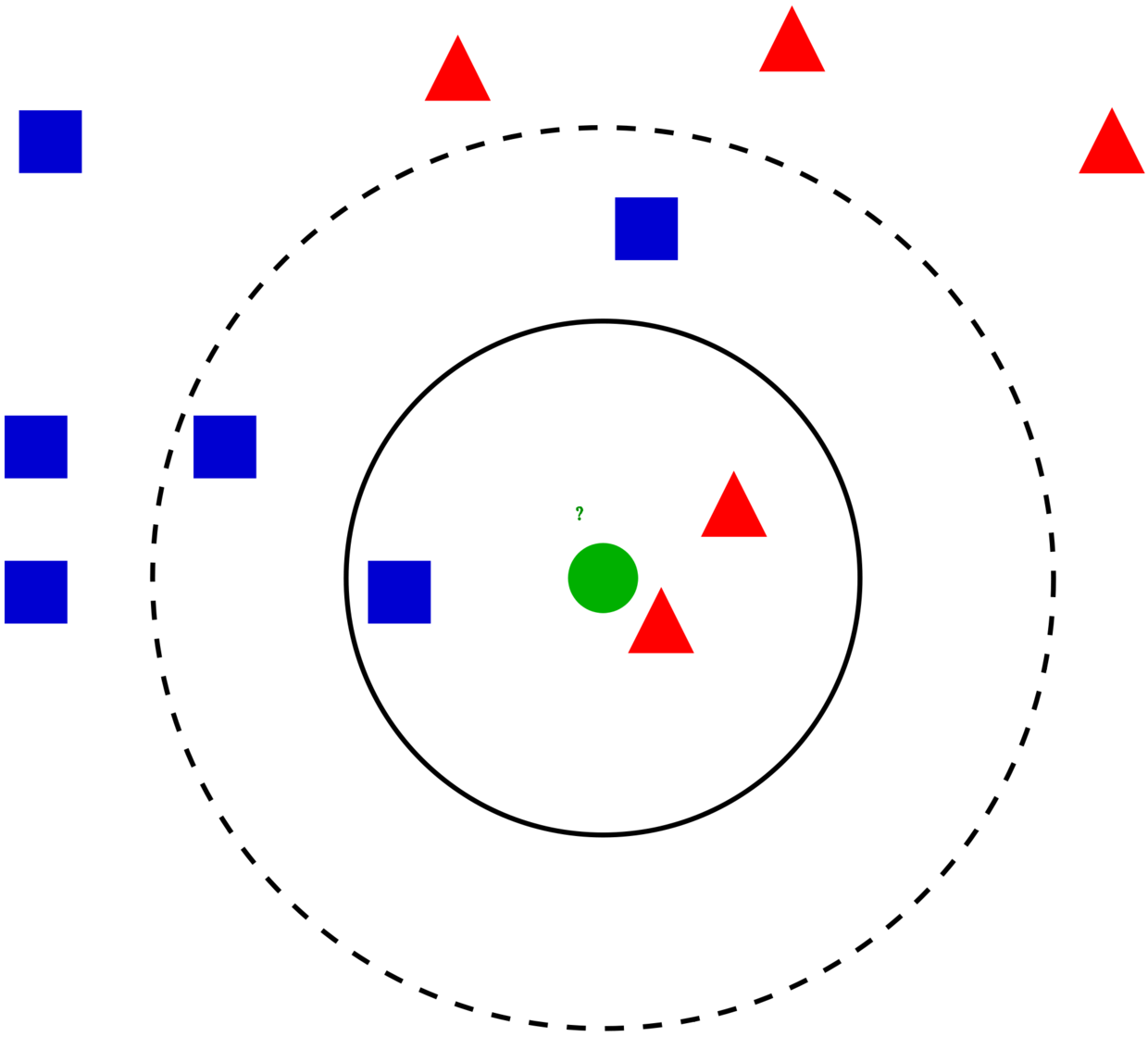
特点
- (1)如上步骤,KNN没有模型训练的过程。需要预测数据时,直接与训练数据集进行计算即可。
- (2)KNN算法中最重要的超参数就是K的选择,会在下面具体操作中介绍。
- (3)因为需要计算距离,所以需要进行数值变量标准化,以及类别变量转化(如果有分类变量的话)。
- (4)KNN在数据量小或者维度较小的情况下效果很好,但不适用于大规模的数据(计算量大)。
关于距离,欧几里得距离,归一化(中心化)
2、mlr建模
2.1 糖尿病数据
|
|
2.2 确定预测目标与训练方法
- (1)确定预测目的:根据三个指标
insulin,sspg,glucose对糖尿病状态class进行诊断
|
|
- (2)确定预测方法:使用KNN分类算法,超参数设为4
|
|
2.3 模型训练、预测
- (1)训练模型:KNN在训练阶段不进行任何计算,直到进入预测阶段之后才进行具体的计算。这在机器学习中是比较少见的,又称为“懒惰”的学习
|
|
2.4 交叉验证
- 交叉验证是将数据分为两部分:训练集+测试集。在训练集中训练模型,在测试集中评估模型的性能,从而避免过拟合的情况。
- 如果对交叉验证的结果满意,最后就可以使用所有数据(训练集+测试集)来训练模型。
- 有3种常见的交叉验证方法:(1)留出法;(2)K-折;(3)留一法。K折交叉验证更常用,演示如下。
|
|
2.5 调节超参数
- (1)设置可选的超参数范围以及搜索方法
|
|
- (2)比较不同超参数的结果
|
|
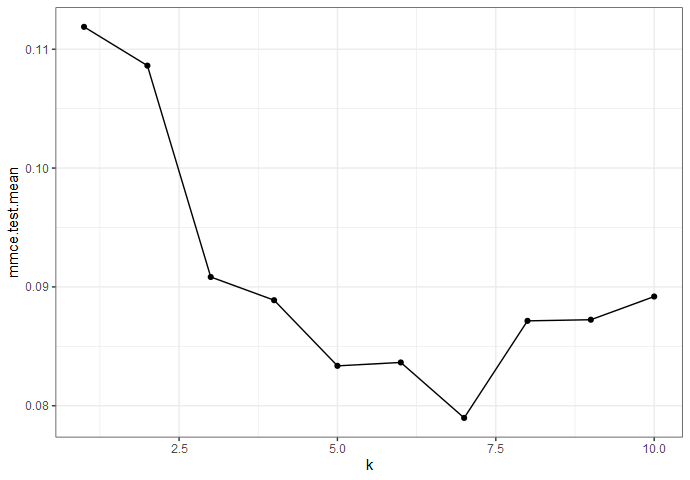
- (3)使用最佳超参数训练模型
|
|
2.6 嵌套交叉验证
- 相对于一般的交叉验证(2.4),嵌套交叉验证更适合于纳入超参数调整等预处理步骤。即在交叉验证中,也去验证超参数调整的步骤。
- 嵌套交叉验证包括内部循环与外部循环
|
|