GeneCompass: deciphering universal gene regulatory mechanisms with a knowledge-informed cross-species foundation model
Cell research | 2024.10
(最后)通讯作者 Xin Li


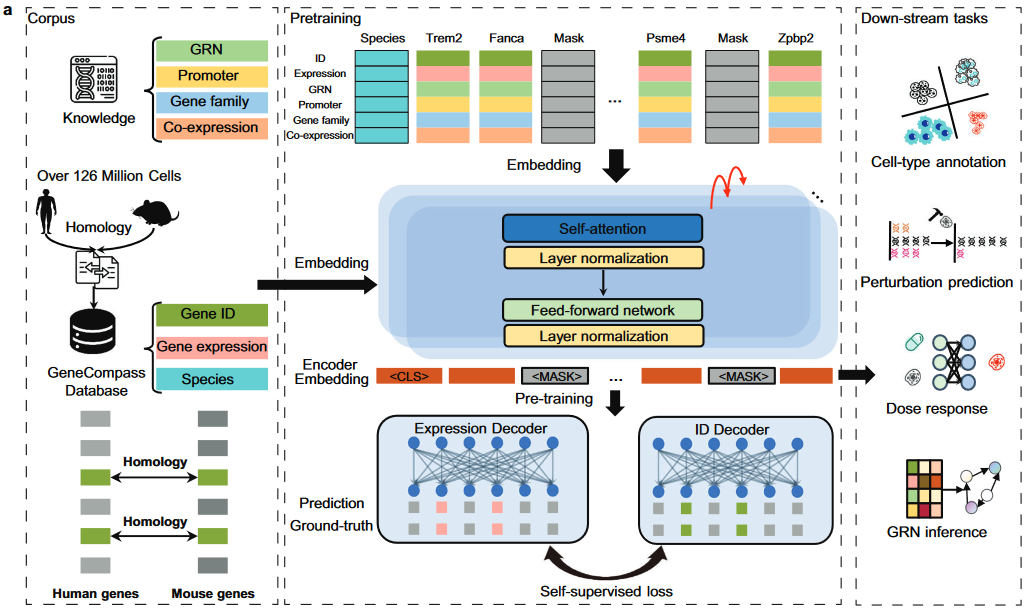
0. 模型预训练
0.1 数据规模
-
初始收集126M,经QC后,101M细胞用于训练(53M 人源 / 48M鼠源) [详见原文/Table S1, PS: 觉得其中一个描述有点问题]
-
包括了正常细胞以及disease cells/cancer cells/immortalize cell lines
-
词汇表包括36092个基因,其中17465个为人/鼠同源基因,其余species-specific基因使用Ensembl ID
0.2 模型框架
- 输入数据:每个细胞样本由2048个基因(Token)组成
- Token Embedding:将Gene ID,Expression Value,以及四种基因先验知识(promoter/GRN/gene family/co-expression)拼接在一起,再投射到768维
- Position Embedding表示每个基因的RANK位置,与上述进行矩阵加法
- 一个特殊的token,用于标记物种类型[推测应该是指cls标记]
存疑:Method并没有提到基因表达的标准化方式。Gene value与Position rank某种程度上是不是信息重复了
-
Transformer
- L =12; H = 12; D = 768 (参数量>100M)
- 两个自监督任务:预测掩码基因的Expression(MSE loss)与ID (Cross-entropy loss)
-
Train config
- Batch=10
- LR = 0.00005, Warm-up=10000
- 3 Epochs (直至loss不再降低|并没有设置验证集,Geneformer也没有)
- Cost 9 days using 4×8 A800 GPUs
-
小规模的消融实验
- 数据:human cells (55M) 模型: L = 6; H = 4; D = 256
- 比较是否加入先验知识对模型的下游任务影响
1. Gene embedding 初步探索
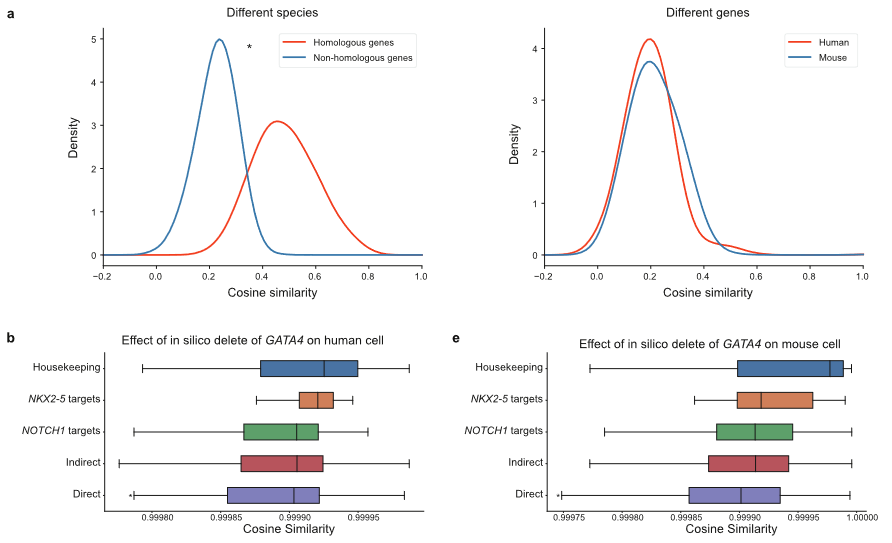
- 跨物种的同源基因的相似度较高(相比同物种的不同基因);
- 贡献:Attention > Prior knowledge
- 模拟基因(GATA4/TBX5)敲除会显著影响其直接靶基因的状态。
- 模拟敲除转录因子,观察其靶基因
- 不同物种观察到相似的结果
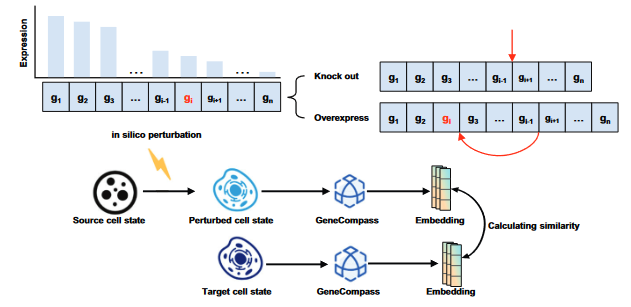
- In silico perturbation refer to scGPT(基于注意力)
2. Cell embedding 细胞注释
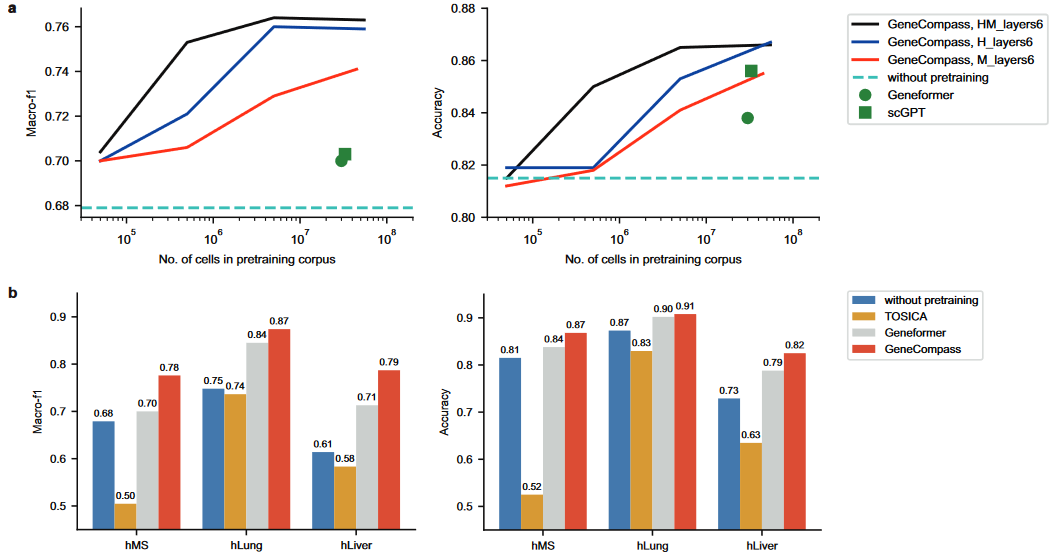
- 下游的细胞注释效果越好
- 预训练细胞量越多;
- 结合跨物种数据由于单物种数据
- 12注意力层优于6注意力
- 优于其它类型模型
- GeneCompass without pretraining
- TOSICA
- Geneformer
- 作为CAME(一个图神经网络,用于跨物种细胞类型注释)的初始输入,可以增强其最终的预测效果
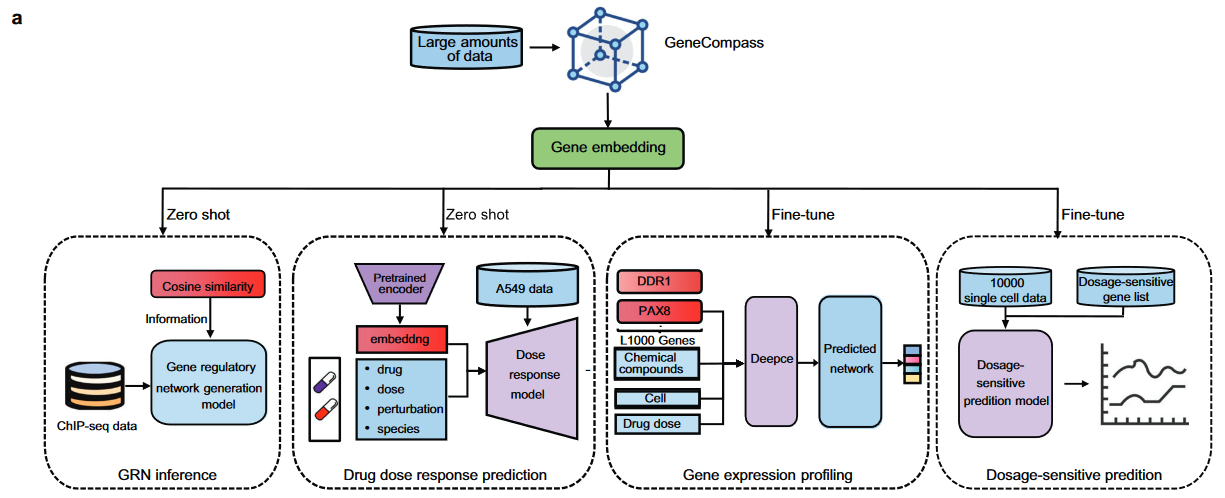
3. 基于Gene Embedding的衍生任务
3.1 GRN推断
- 根据Gene Embedding的相似度得到邻接矩阵,作为DeepSEM模型的输入
- 效果优于来自Geneformer、scGPT的Gene Embedding,以及vanilla DeepSEM
3.2 Drug Response预测
- 预测基因在不同种类和浓度扰动条件下的表达情况
- 分别用GeneCompass,Geneformer,scGPT计算的Gene Embedding替换CPA(单基因预测/drug dose response prediction)、DeepCE(全基因预测/gene expression profiling)的初始输入。Genecompass均能取得较好的结果。[药物扰动]
3.3 Dosage-Sensitive Gene预测
- 基于Gene Embdding的二分类微调任务
- 效果优于Geneformer,以及没有预训练的Genecompass
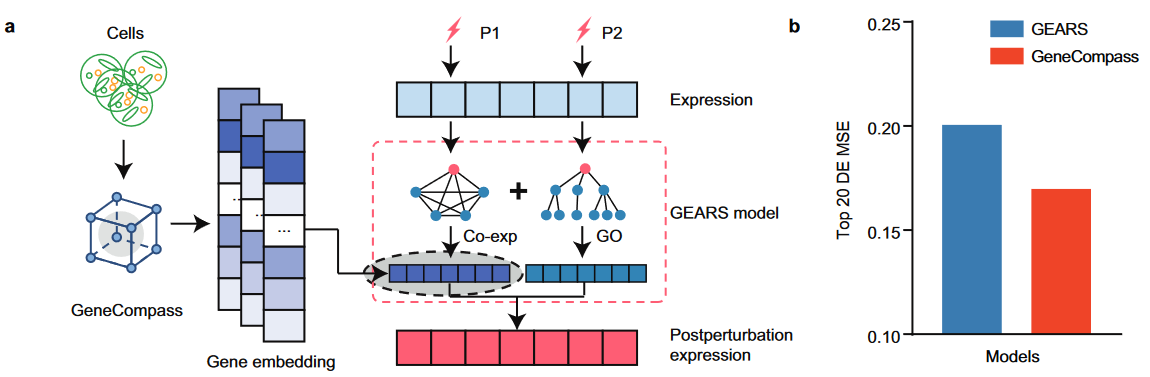
4. 基因扰动预测
- 预测特定基因突变条件下,其余基因表达的变化情况
- 基于GEARS框架,将Genecompass的Gene Embedding替换GEARS原有的基于共表达的Gene Embedding表示,观察到性能有明显提升。
5. ISP (In silico perturbation)
验证性分析
- 过表达OSKM使得成纤维细胞向iPSC状态变化
- 敲除维持干性的转录因子(Zbtb11, Zfp131),使得鼠源胚胎干细胞向内胚层变化。
筛选应用
- 逐一过表达ESC细胞的基因,筛选可以将ESC向Progenitor (前体细胞)或者更加成熟的Leydig (激素分泌细胞)状态变化的调控因子。取二者的交集,最终发现了5个。
- 通过湿实验/测序,证实了其中NR5A1与GATA4的效果