- Transfer learning enables predictions in network biology | Nature
- https://doi.org/10.1038/s41586-023-06139-9
- Received: March 2022 / Accepted: 27 April 2023
- Data & Code: https://huggingface.co/ctheodoris
通讯作者:Patrick T. Ellinor, M.D., Ph.D.

1. 预训练模型
1.1 scRNA-seq数据
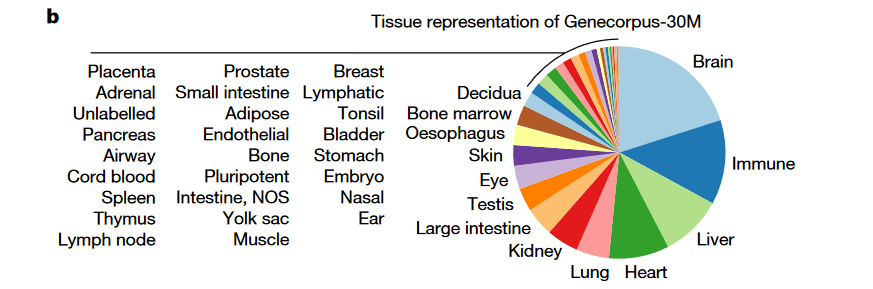
数据收集:Genecorpus-30M
- 561 datasets; 29.9 M cells
- 数据集详细信息见SP Table-1
数据质控:
- Exclude cells with high TMB
- Exclude cells via total read counts/线粒体 read counts
- Exclude cells with at least 7 features (7 * 0.15% = 1)
- Exclude possible doublets and/or damaged cells
- Select protein coding genes + miRNA
- Finally:~ 2.5w features of 27.4 M cells
表达值RANK encoding
-
先对每个细胞所有基因进行标准化 (10000 library size);
-
再对每个基因在所有细胞进行标准化 (scale factor : non-zero median value);
- 主要原因是不同基因在细胞中 基础表达水平的不同
- 统计发现 House keeping genes 有较高的scale factor,即标准化后值变小
- 而Genes like TFs 有较低的scale factor,即标准化后值变大
-
最后在每个细胞中,将基因按表达由高到低排序RANK。
1.2 模型架构
Transformer模型直接继承于Hugging face的BertForMaskedLM模型
Config参数
- 6 layers with each 4 heads
- gene embedding 256 (ffn: 512)
- max cell sentence length 2048 (dynamic padding),masked feature ratio: 15%
训练参数
- lr 0.001, linear warmup 10k steps
- Relu,Dropout 0.2, Adam with 0.001 weight decay
- Batch size 12, Epoch 3
- 12 V100 GPU (from 3 nodes), DeepSpeed 分布式训练,3 Days
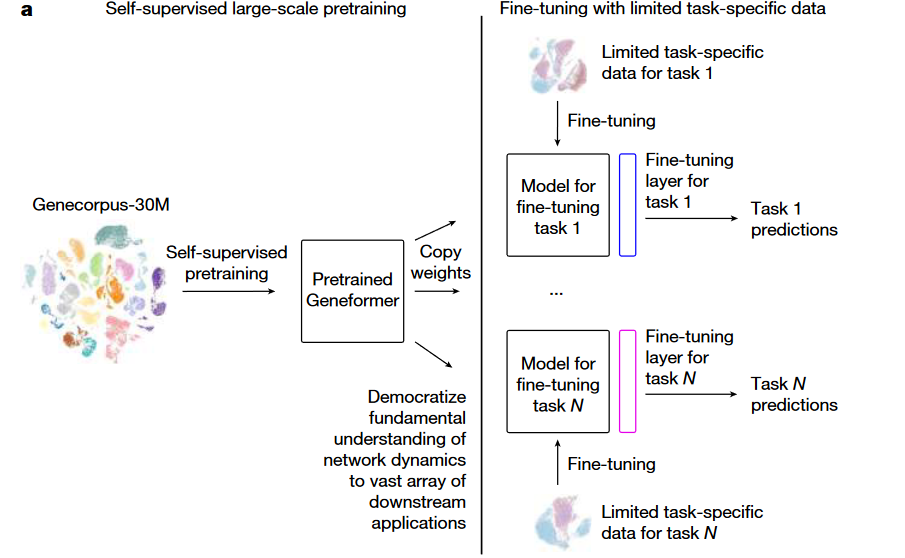
1.3 预训练效果
在具体介绍微调任务前,文章首先论述了其预训练模型的可应用性
Gene embedding
- Not affect by common batch-dependent technical artefacts
- 同一基因在不同Batch因素的Cosine相似度高
- Context awareness
- 人为地将Fibroblast的OSKM基因Rank提高 (In silico activation),使得其它基因的Embedding向iPSC状态变化。
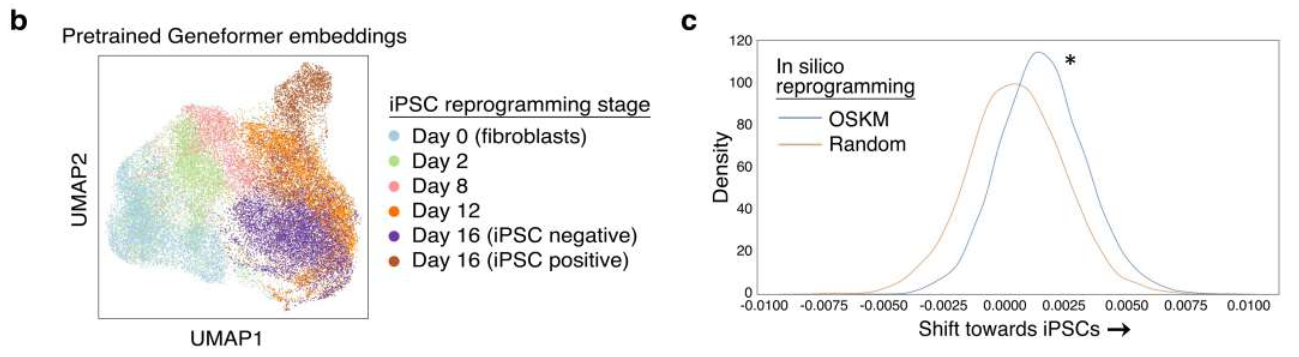
iPSC, induced Pluripotent Stem Cells: 已经分化的体细胞通过导入特定的转录因子重新编程而成的一种细胞类型
OSKM: 四个关键的基因重编程因子: Oct4 , Sox2 , Klf4 , c-Myc
Cell embedding (Cell embedding取每个细胞所有基因的Embedding的均值)
- Batch integration: cell cluster by cell type, not sample or platform
- 细胞类型注释微调任务:对比了两种已发表方法:CasTLe (XGBoost), scDeepSort
2. 微调任务
- 多为Gene classification/Cell classification任务,(详见SP Table2, 每个fine-tune task会freeze diff number of layers)
- AUC, F1 score
- 5折交叉验证
- 所有微调任务都使用相同的超参数组合,以说明预训练模型的性能。
- lr 0.00005,linear warmup 500,Adamw weight decay 0.001
- batch size 12,epoch 1 to avoid overfitting
It should be noted that hyperparameter tuning (超参数调优) for deep learning applications can generally significantly enhance learning effect. 但是文章中并没有这么做,表明后面微调模型性能都是underestimate。
2.1 Gene dosage sensitivity
基因对剂量的变化的高敏感性,即其少量表达变化可能引发显著的细胞或生物学后果。
- 目的:预测基因(TF)是否为剂量敏感性的TF
- 数据:一方面收集来自已报道的阴性或阳性标签数据,另一方面,使用30M中随机的10K的细胞作为训练数据。
- 结果:交叉验证的AUC 0.91,并在一个基于CNV的外部测试集数据也表现很好(微调数据与标签来源数据越接近,则效果越好)。
- In silico deleting(knockout): 在预训练模型中,模拟敲除心肌细胞的一个心脏疾病相关基因,使得cell Embedding发生较大改变(与敲除前的cell embedding similarity)
- 发现并验证了一个靶点 TEAD4
- 相关Genelist做通路富集分析
Smaller cosine similarity indicates more deleterious effect
2.2 Chromatin dynamics
同一个基因的启动子区域能同时携带 H3K4me3 活跃信号 和 H3K27me3抑制信号 的修饰,这种情况被称为“双价域”(bivalent domains),常见于胚胎干细胞(ESC)中。
- 目的:鉴别基因是否为双价修饰基因 or 非甲基化启动子基因或仅带有 H3K4me3 的基因
- 数据:一方面收集56个保守区域的标签数据,一方面取15K个 ESC作为训练数据
- 结果:AUC可以达到0.93 (bivalent vs unmethylated gene), 0.88 (bivalent vs H3K4me3-only)
- 衍生任务:预测long- versus short-range transcription factors
2.3 Network dynamics predictions
NOTCH1 是一个跨膜受体,在多种细胞类型中起关键作用,特别是在细胞命运决定、分化、增殖和凋亡等过程中。先前研究表明NOTCH1(N1)调控网络在心脏瓣膜疾病发挥重要作用。
- 目的:区分N1基因网络中的核心基因与外周基因
- 数据:标签数据来自先前研究报道,使用30K心脏内皮细胞作为微调数据
- 结果:AUC = 0.81
- 衍生分析:探索出最少需要多少微调训练数据(细胞量)可以达到良好的预测效果(884)
3. 模型深入分析
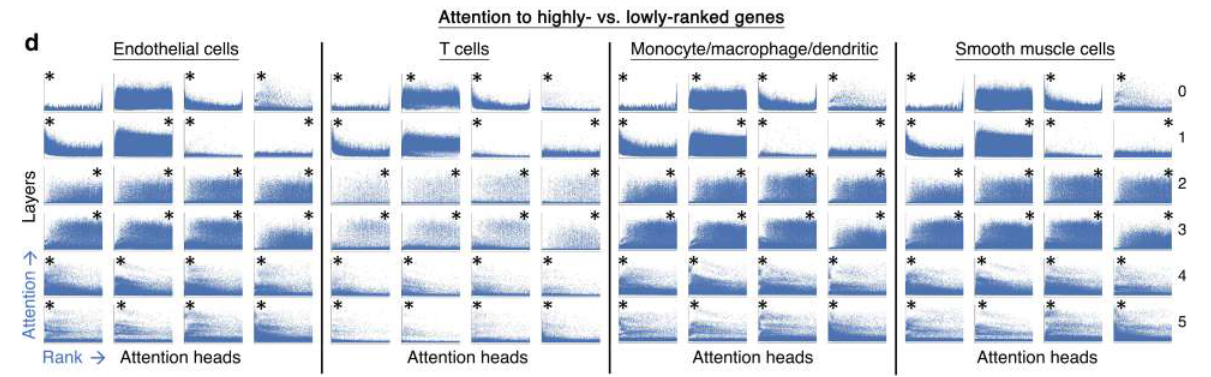
3.1 Network Hierarchy
即对于预训练Transformer,Attention weight的注意力权重分析。
- 6 layers, each 4 heads 都纳入考虑,每个预训练细胞的基因都有unique attention map
- > 20% heads ,TF genes have higher attention
- Genes with high rank have higher attention
- Early / Late layers also show diff patterns
3.2 TF Gene Network
-
方法类似2.1的In silico deletion,不过这里是为了发现转录因子(GATA4)的靶基因。
-
即计算GATA4删除后,其余基因的Embedding与未删除前的Embedding的相似度。越低表明被该TF影响的可能越高。
-
结果发现,先前报道具有高置信度的GATA4靶点,确实表现出更低的Cosine similarity。
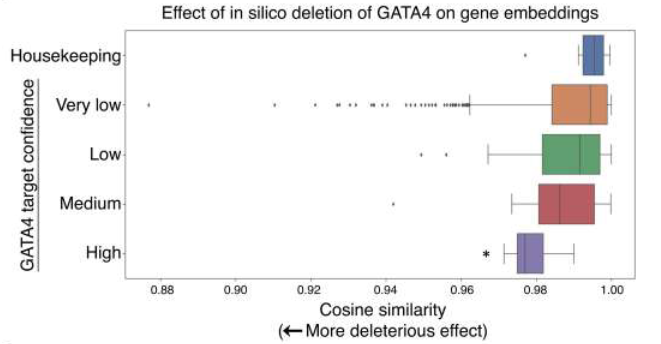
衍生分析发现也可以同时删除两个TF genes
3.3 Therapeutic Target
hypertrophic cardiomyopathy, HCM: 肥厚型心肌病;
dilated cardiomyopathy, DCM: 扩张型心肌病
-
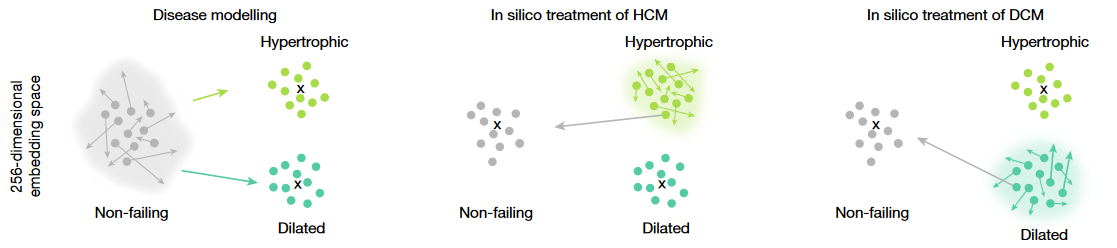
首先,微调两个模型,分别区分来自正常心脏和上述两种心肌病的心肌细胞。
-
“Normal”→“Disease”:Genes whose deletion or activation in normal status make cell embedding shift towards HCM/DCM.
- **Disease genes **– 通路富集分析
-
“Disease”→“Normal”:Genes whose deletion or activation in disease status make cell embedding shift towards normal cells.
- Therapeutic targets – 通路富集分析
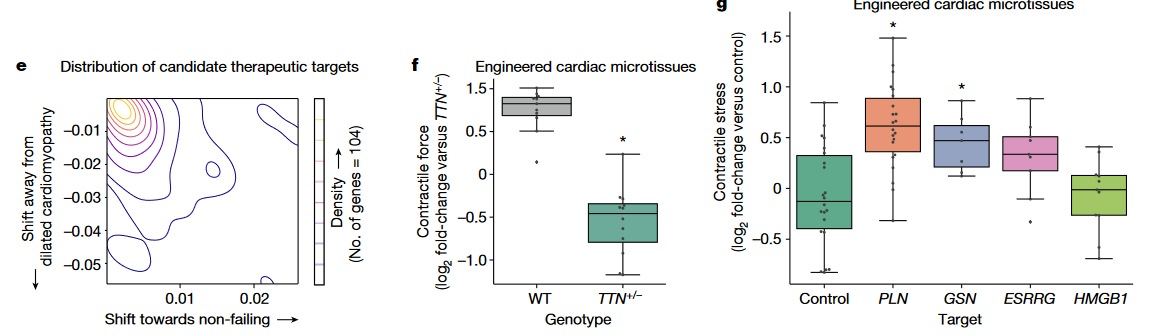
- 最后,对部分靶点进行实验验证
In silico deletion: remove the gene from the rank value of one cell
In silico activation: move the gene to the front of the rank value of one cell