标题 CELLPLM: PRE-TRAINING OF CELL LANGUAGE MODEL BEYOND SINGLE CELLS https://openreview.net/forum?id=BKXvPDekud
发表 ICLR (The International Conference on Learning Representations) 2024
通讯 Jiliang Tang | Computer science and engineering department | Michigan State University | https://www.cse.msu.edu/~tangjili/
1. 简介
(1)scRNA-seq data与natural language的区别
- Not sequential → Bag-of-genes (refer to bag-words)
- Cell-cell communication在细胞状态和发育过程中的重要性
- 单细胞数据的Quantity与Quality较差
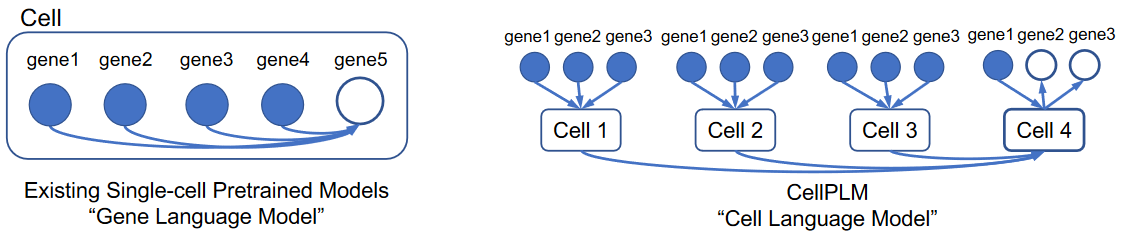
(2)CellPLM模型特点 (single-Cell Pre-trained Language Model)
- Cell language model to account for cell-cell relations
- 预训练过程使用了spatially-resolved transcriptomic (SRT) 空间转录组数据
- 使用高斯混合分布模型学习细胞的潜在分布空间
To the best of our knowledge, the proposed CellPLM is the first pre-trained transformer framework that encodes inter-cell relations, leverages spatially-resolved transcriptomic data, and adopts a reasonable prior distribution.
(3)CellPLM模型的核心假设
对于N个细胞,k个基因的单细胞表达矩阵X ∈ [N, k]
- 如下式1,O(i)代表细胞i的 unobserved (masked) genes, U(i) 代表细胞i的 known (unmasked) genes
- 即细胞i的某一基因表达的条件概率分布(conditional probability distribution)可由同一细胞内其它已知表达的基因推测得到。
- Gene tokens → 这在许多现有的单细胞Transformer模型常用(e.g. scBERT, scGPT)
- 如下式2,M表示unobserved genes from many cells, Mc表示表达矩阵X的补集。
- 即即预测细胞i的某一基因表达可以同一组织内多个细胞的known genes推测得到
- Cell tokens → 这是CellPLM模型的核心假设

2. 预训练
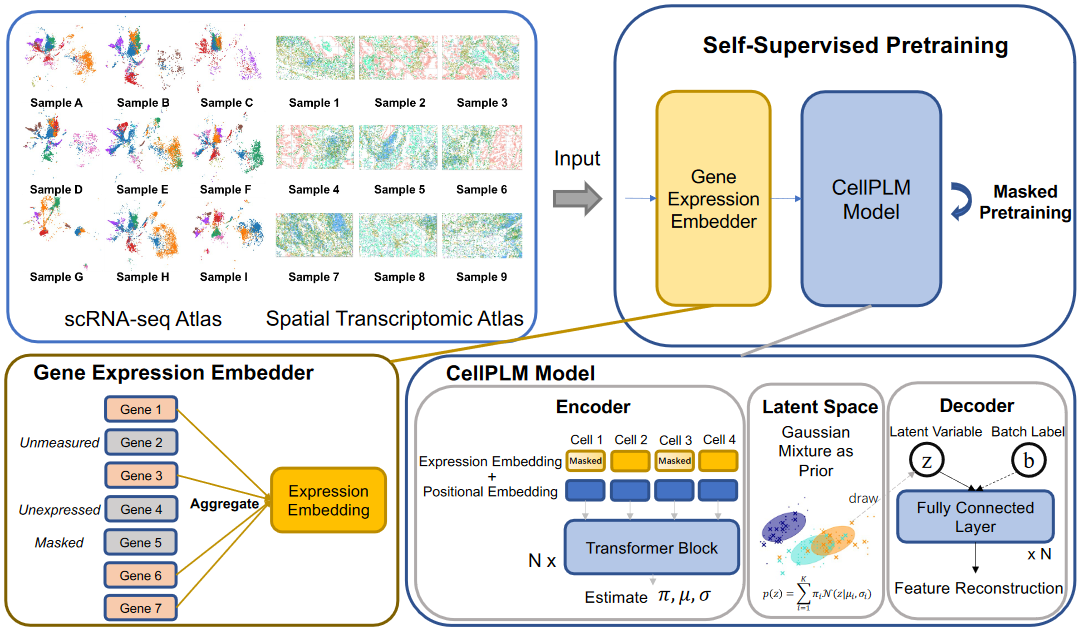
预训练模型分为4个组成部分
(1)Gene expression embedder (cell level)
参考词袋模型,将基因信息整合至细胞水平。
-
首先对gene token计算初始化的embedding表示 → (gene, embedding)
-
然后以细胞的基因表达(cell, gene)分别为系数计算加权和,作为初始化的Cell Expression Embedding (N, d)
表格矩阵基于Seurat进行了标准的library size normalization and log1p transformation处理。
一个批次的cells作为一个样本 (N, k),dataloader一次只迭代一个批次(batch_size = 1)
(2)Transformer encoder
首先Transformer的input cell embedding由两部分组成(Cat/Add)。除了上面的Expression Embed,还有Position Embed
- 对于SRT数据,position embed由一个FOV (fields of view) 内的细胞二维坐标生成。
- 对于普通scRNA-seq数据,由于没有相关信息,则通过随机生成进行表示。
然后将上述的合并 Cell embedding输入到Transformer层中
- 采用了具有线性复杂度的Flowformer计算注意力。
- 输出为(N, d)
(3)Gaussian mixture latent space
对注意力编码层的细胞表示(N, d),使用高斯混合分布模型学习其潜在分布空间 z
- 一方面学习得到潜在分布空间(N, z)
- 另一方面得到的变分重构损失(Latent loss)作为预训练损失的一部分
(4)Batch-aware decoder
预训练的最终输出是基因表达矩阵(N, k)。在输入到MLP预测前,需要进一步考虑其它因素(Covariates)对于表达量的影响。
- CellPLM考虑的协变量因素包括批次效应/测序平台/数据集
Loss
最后将细胞的潜在分布空间(z)以及协变量表示(b)合并后,输入到MLP网络,再经自然指数转换后,得到最终输出 (N, k)
使用NB负二项分布计算对于掩码基因的预测值与真实值之间的差异损失(Target loss)
最终预训练损失由两部分组成:Latent loss + Target loss,进行反向传播,更新模型。
预训练模型包含82M参数,对11M细胞数据进行训练,<24 hours via 8 v100 GPUs
- scRNA-seq
- HTCA (human tumor cell atlas):4.7M
- HCA (human cell atlas):1.4M
- GEO:2.6M
- SRT:2.7M
3. 微调训练
基于预训练模型,文章采用了多角度的微调任务,并与其它已有模型做对比,表明CellPLM的优势。
对于每个微调任务,文章基本都从Downstream Task Datasets,Evaluation Metrics,Baselines,Fine-tuning展开。
Cell-level tasks
- Zero shot:直接使用预训练模型,计算细胞的嵌入表示h (可用于聚类分群等)
- Cell Type Annotation:基于Latent输出的cell embed,连接MLP层预测细胞类型
通过查看仓库代码,上述两种微调任务都是直接使用的Latent layer输出的细胞嵌入表示执行相应任务。
Gene-level tasks
-
scRNA-seq denoising:将一部分非零表达基因置换为0后,训练模型预测其原有表达值。
-
Spatial Transcriptomic imputation:本质上,也是将一部分基因置换为0, 训练模型预测原有表达值。训练过程中,需要联合scRNA-seq作为Reference数据,帮助训练。
-
Genetic perturbation prediction:预测扰动基因表达,关键是如何在输入数据中标记出被扰动的基因
- For one perturbation, we set the input of perturbed genes to be −100 to mimic the gene perturbation action.
4. 其它方面
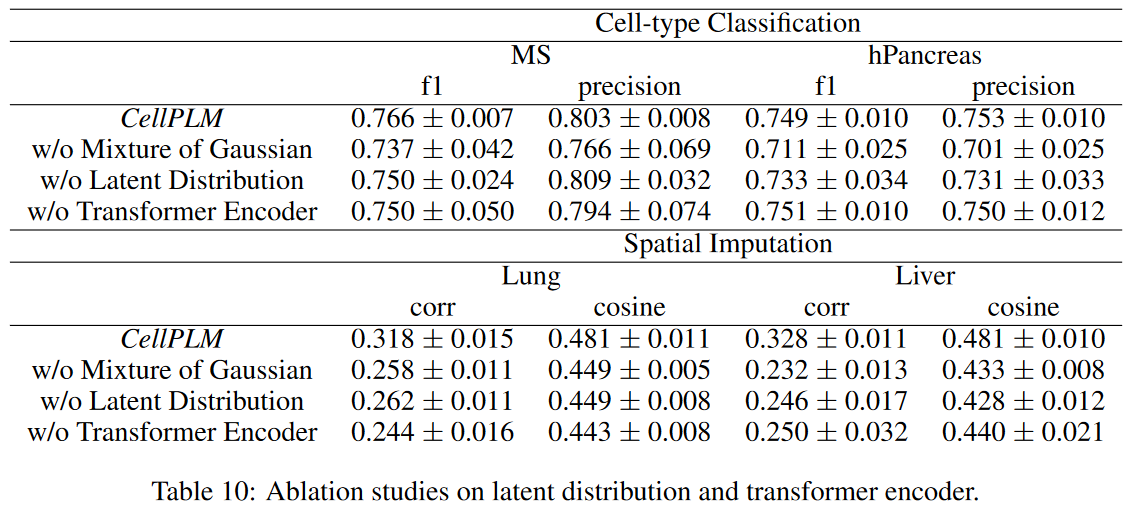
(1)消融实验
通过三个角度的消融实验,说明CellPLM模型component所发挥的作用
- 是否采用Mixture of Gaussian
- 是否采用Latent distribution layers
- 是否采用Transformer encoder
(2)代码借鉴学习
- 由于单细胞表达矩阵的稀疏性,torch支持相关高效的处理方式
- 例如 torch.sparse_csr_tensor,torch.sparse.mm等
- 通过
from abc import ABC, abstractmethod,使得更加容易地实现同一类型,不同变体的模型component- 例如 不同的微调 Pipeline
- 每个模型component都由一个文件夹组成,通过其中
__init__.py文件发挥关键作用。 - Transformer,VAE等经典模型的实现方式