标题: scBERT as a large-scale pretrained deep language model for cell type annotation of single-cell RNA-seq data
期刊|日期:nature machine intelligence, 2022/09
DOI:https://doi.org/10.1038/s42256-022-00534-z
作者:腾讯AI Lab 研究团队
1. 背景
(1)目前在scRNA-seq研究中,有三种常见的细胞类型注释方法。作者认为都存在一定的缺点。
- 基于Marker基因的手动注释:较为主观,marker基因缺少或不准确等
- 基于Atlas图谱的相关性注释:容易受到批次效应影响
- 基于有监督学习模型的预测:大部分使用HVG等部分基因建模,忽略全局角度
(2)受NLP领域的BERT模型启发,文章建立了scBERT模型专门用于单细胞数据的细胞类型注释任务。
- 预训练:基于数百万单细胞数据得到的预训练模型学习基因间相互作用;
- 微调:基于参考数据集,学习标注细胞类型相关的基因嵌入表示
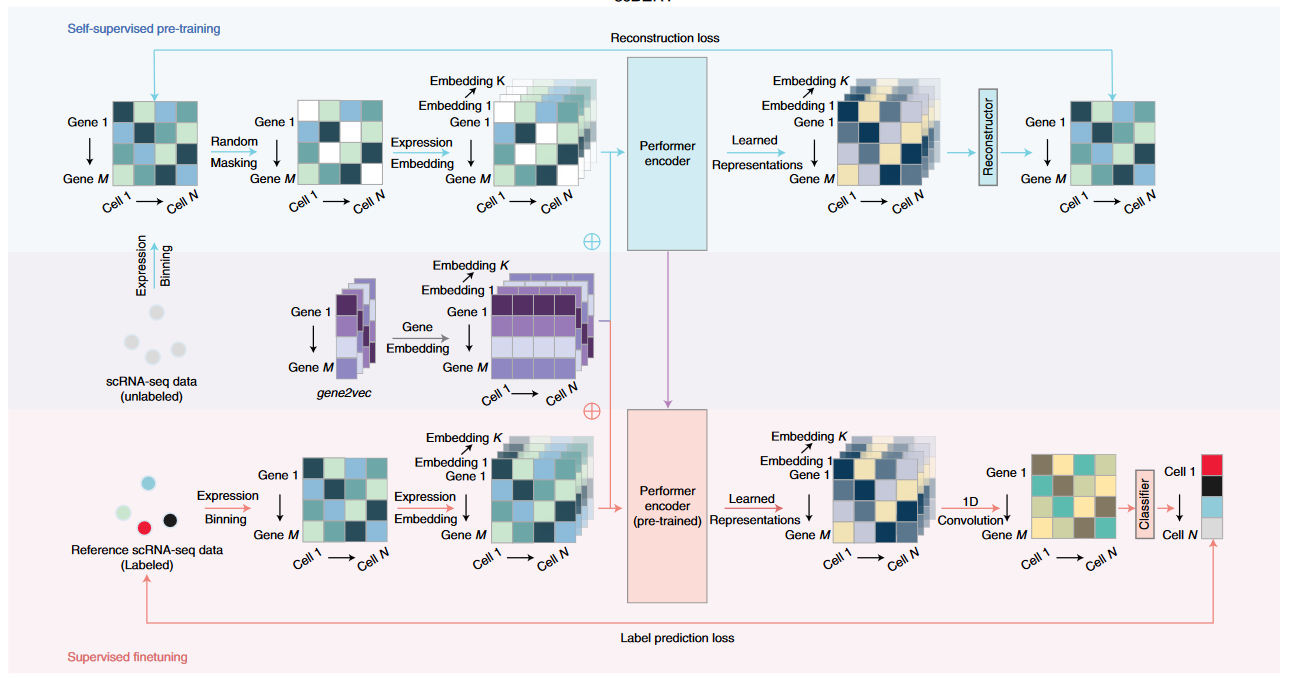
2. 建模过程
2.1 输入数据
表达矩阵预处理包括标准化,log转换,过滤表达基因小于200的细胞。
一个样本序列是一个细胞1.6w个基因表达情况,采用如下两种方式进行嵌入表示
- Expression embed: 将每个基因的表达值分bin处理为整型后,使用
nn.Embedding()提取表示 - Gene embed:基于gene2vec工具提取每个基因的高维表示
最后将上述每个基因的上述两种表示相加,得到每个基因最终的200维向量表示
Tips: 上面的Exp embed中相当于把离散化的表达值作为token处理。这与scGPT等将Gene id词汇表作为token处理方式不同。
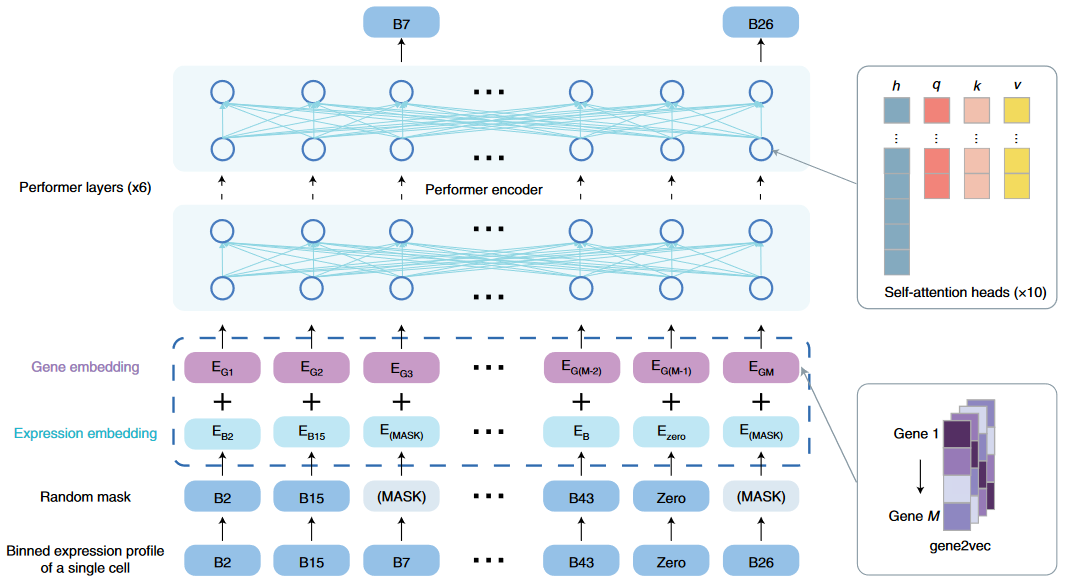
2.2 自监督训练
- 对每个细胞的15%的非零表达基因进行掩码,然后自监督输出的目的是重构掩码基因的表达。
- 预训练数据来自Panglao数据库, 包括209个数据集,74个组织,1M个细胞。
- scBERT采用了Performer用以高效计算针对长序列的注意力。这里处理的序列长度(Genes)是1.6w。(例如BERT模型处理的序列长度才是512)
- 10 heads each block, 6 block
尽管如此,文章提供实现的batch size也不大,只有3。
2.3 微调任务
- 基于参考数据集(有标注细胞类型)进行微调,根据细胞基因表达输入,预测其细胞类型。
- 框架简单来说(参看那边):
- 首先引用卷积层将基因的200维特征提取为1维
- 然后使用MLP将细胞的1.6维基因特征映射为单个值
- 最后应用softmax分类器,计算交叉熵损失
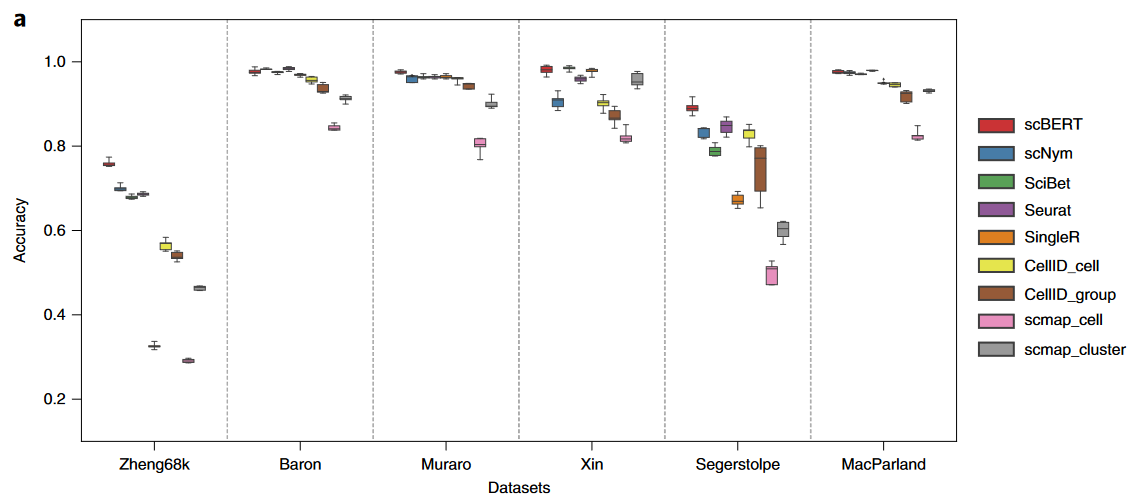
3. Benchmark
接下来,文章基于获得的预训练模型进行了大量的benchmark工作,说明scBERT模型的优势。
对比的方法包括如下——
- marker based : SCINA, Garnett, scSorter
- correlation based : Seurat, SingleR, CellID, scmap
- supervised classification : scNym, Scibet
对比的角度包括:
(1) Intra-dataset:在单个数据集内部进行5折交叉验证。
(2) Inter-dataset:跨多个相似数据集的应用。例如4个数据集中,数据集为单位的CV。
(3)其它方面:包括允许对未知细胞类型的预测,以及注意力矩阵的可解释性分析等