【01】ccRCC与免疫浸润
标题:Landscape of immune cell infiltration in clear cell renal cell carcinoma to aid immunotherapy
期刊|日期:Cancer Science | 13 March 2021
1 第一次分亚群
- TCGA 525样本(TPM)
- 29个 imunne signature:16 免疫细胞与13个免疫相关功能
- 使用ssGSEA方法计算每个样本的signature score
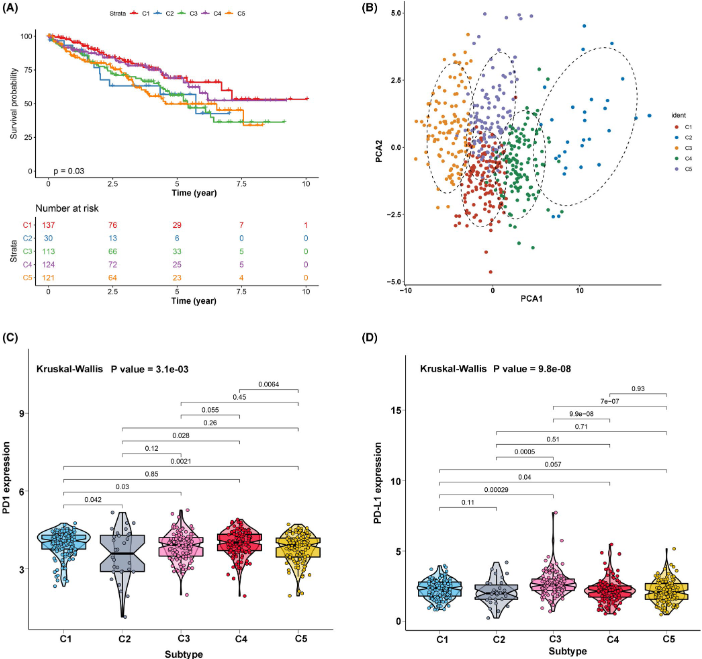
- 基于signature score,使用ConsensuClusterPlus包分得5个亚群
- PCA可视化轮廓
- PD-1与PD-L1表达差异
- C1+C4与C2+C3+C5间生存差异
2 第二次分亚群
- 两大组之间(C1+C4与C2+C3+C5)进行差异分析,得到658个差异基因
- 基于上述差异基因,再次使用ConsensuClusterPlus包重新分成3个亚群
- PCA可视化轮廓
- PD-1与PD-L1表达差异
- 三组间生存分析
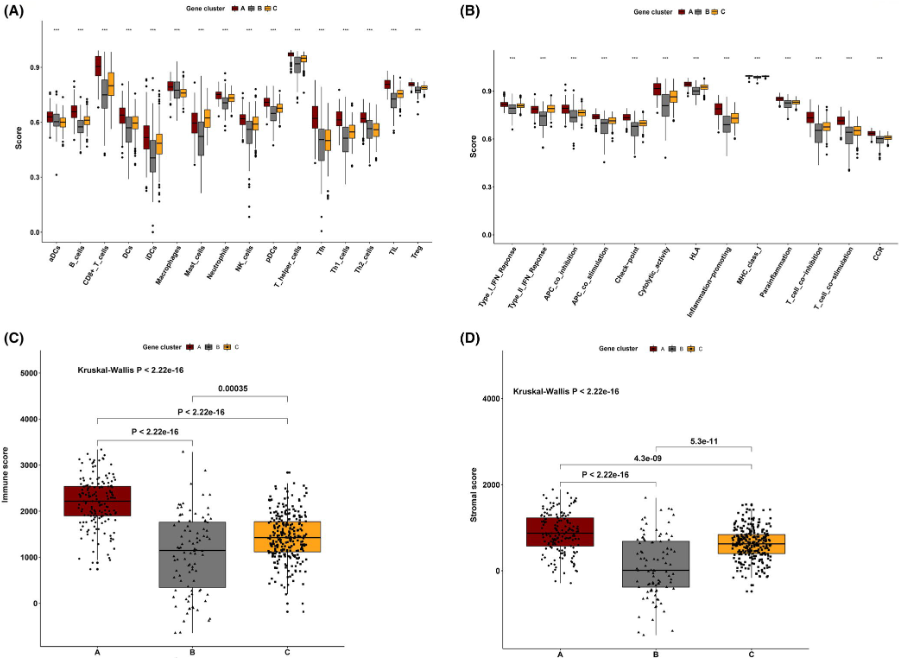
- 29种signature score在三组间的分布差异
- immune score与stromal score在三组间的分布差异(ESTIMATE)
- 根据与分组的相关性,将上述差异基因分成signature A/B
- 通路富集分析
3 TII分数及衍生分析
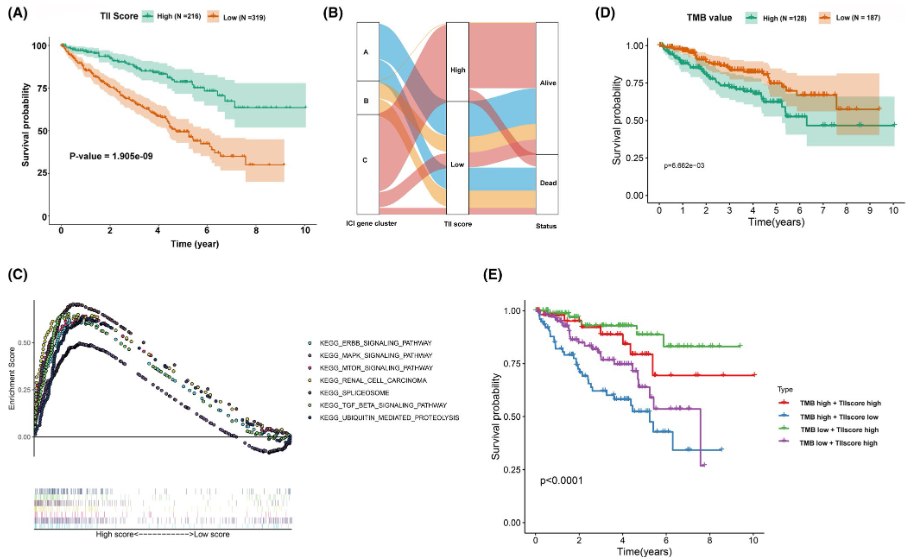
- 分别计算signatue A/B的第一主成分之和,然后相减作为每个样本的TII score
- 根据survmier包计算最佳阈值划分成high与low score两组,并进行一系列后续分析:
(1)生存分析
(2)与之间鉴定亚群的关系
(3)GSEA分析
(4)与TMB的独立性分析
(5)29种signature score在两组间的分布差异
(6)免疫检查点基因表达在两组间的分布差异
(7)使用pRRophetic进行药物敏感度分析
(8)基于两组间的差异基因【high vs low】进行CMAP药物匹配
(9)TII分组与临床信息的关系(fish exact test)
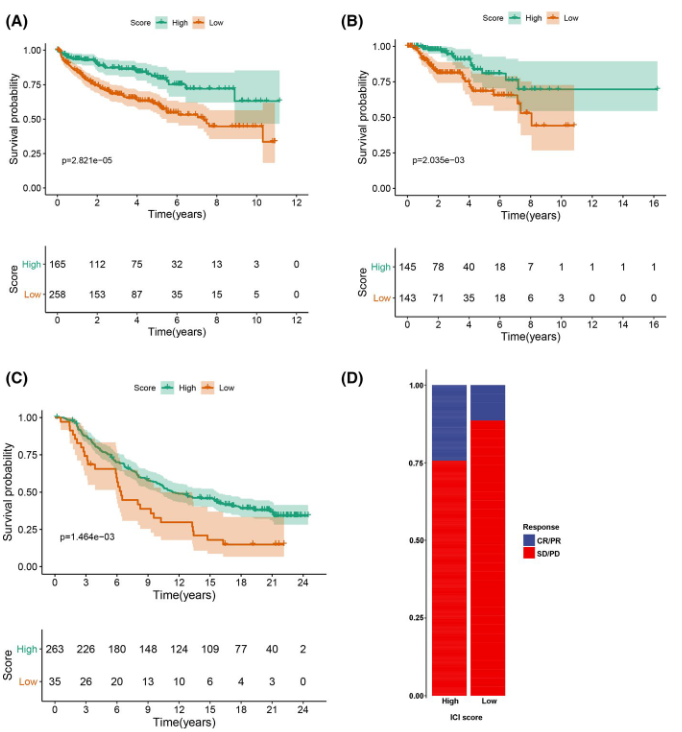
- 外部数据集验证(生存分析)
- PFS
- TCGA papillary renal cell carcinoma乳头状肾细胞癌
- IMvigor210 cohort(urothelial cancer,尿路上皮癌)
【02】ccRCC与IFN-γ相关基因
标题:Comprehensive Multi-Omics Identification of Interferon-g Response Characteristics Reveals That RBCK1 Regulates the Immunosuppressive Microenvironment of Renal Cell Carcinoma
期刊|日期:Cancer Science | 02 November 2021
1、整体批量分析
- 24个IFN-γ响应相关基因
- (1)TCGA:530 ccRCC, 323 pRCC, and 91 chRCC
- 分别与癌旁组织差异分析
- 基因高低表达分组与病人癌症阶段(Stage),生存时间的关系
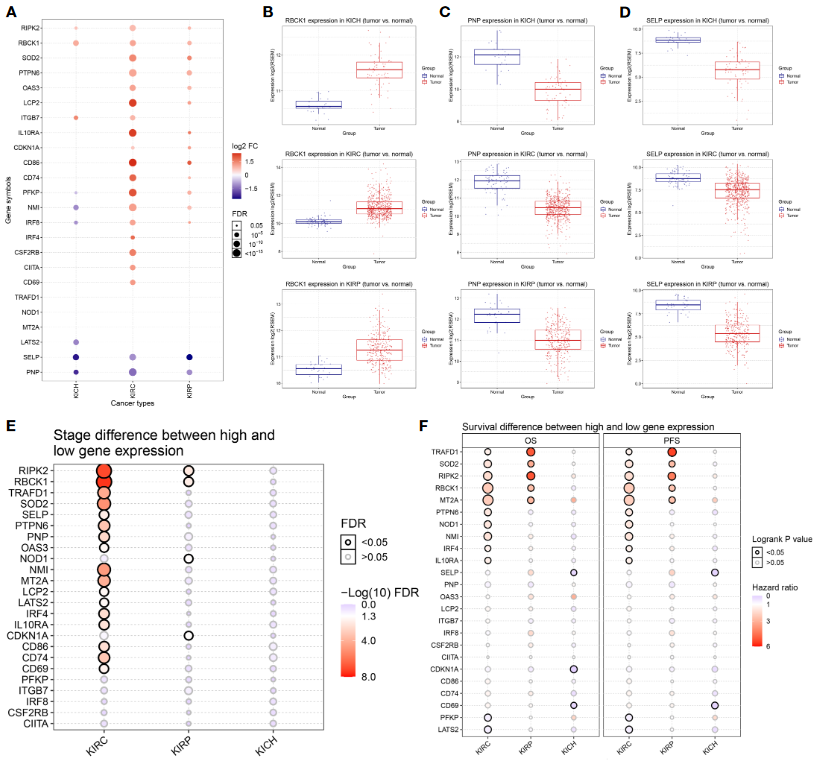
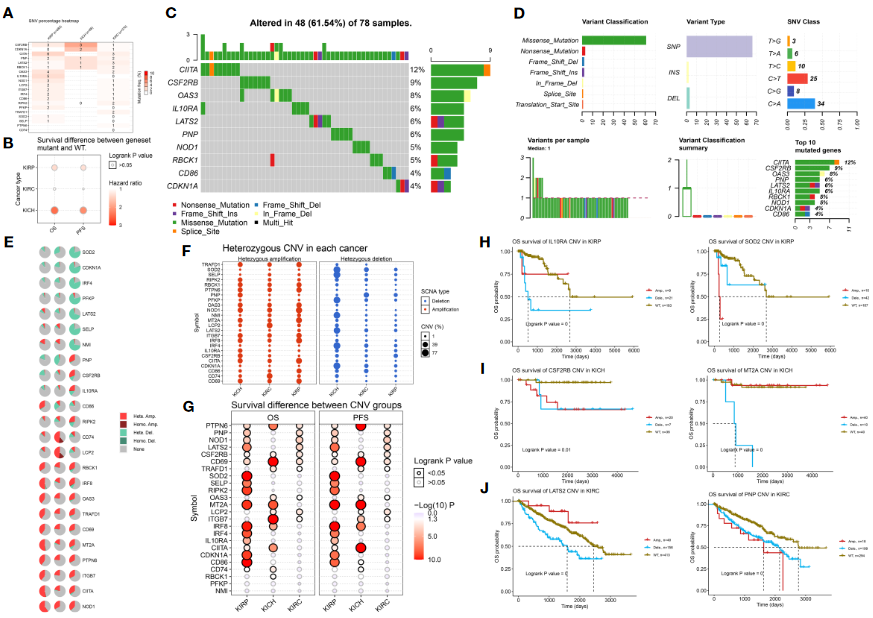
- (2)肿瘤突变数据:SNV,CNV,genetic mutation
- 基于GSCA网站完成:http:// bioinfo.life.hust.edu.cn/GSCA
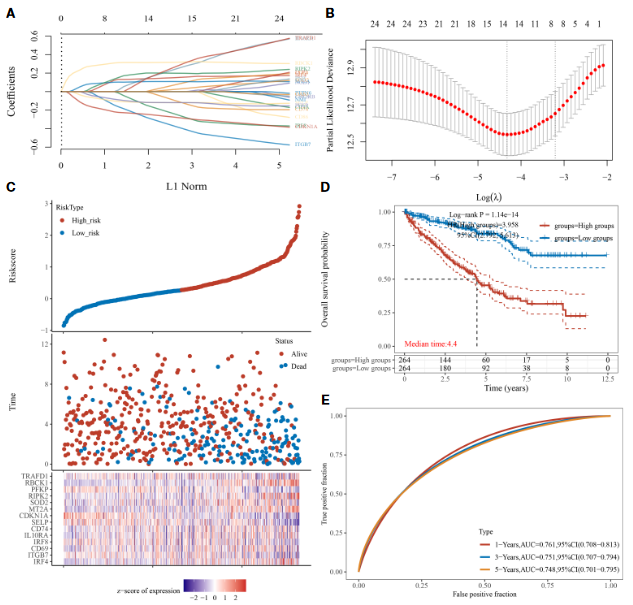
2、预后模型筛选基因
- 分别从ccRCC,RCC的OS与RFS(2×2)的4种组合进行套路分析
- LASSO回归建模
- 计算risk score,以中位数分组
- 生存分析,AUC分析,免疫浸润相关性分析
- 基于ccRCC的OS预后模型,对高低风险组差异基因分析,通路富集
- 分别以OS与PFS为目标的诺模图分析发现RBCK1为关键基因
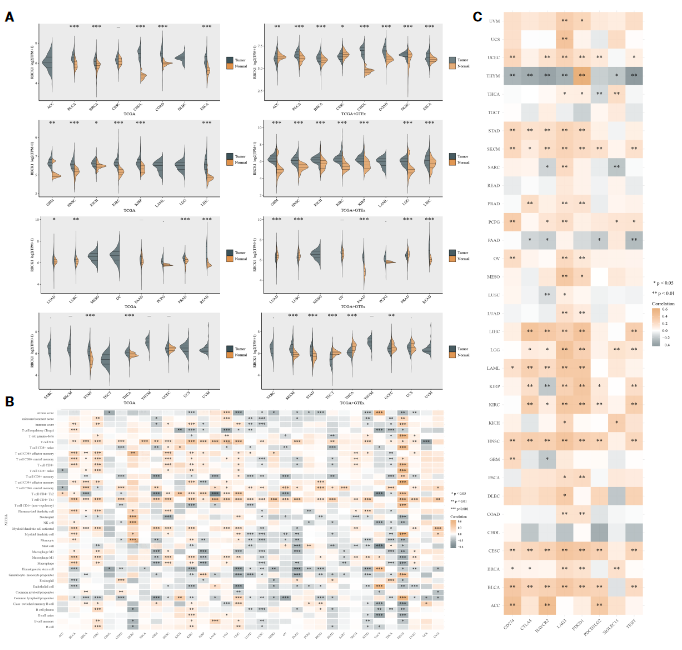
3、单基因分析
- 基于单细胞数据集(TISCH database),发现基因高表达的免疫细胞类型
- 泛癌分析(1)与癌旁组织的差异表达;(2)与免疫检查基因的相关性
- 基因表达高低分组与TIDE score、免疫检查点基因表达相关性
- ICGC验证TIDE score
- FUSCC数据集验证差异表达,免疫浸润、生存分析
【03】ccRCC的免疫预后基因
标题:Immune-Associated Gene Signatures Serve as a Promising Biomarker of Immunotherapeutic Prognosis for Renal Clear Cell Carcinoma
期刊|日期:Frontiers in Immunology | 24 May 2022
- 1、筛选候选基因
- TCGA:539 tumor/72 normal → 9459 DEGs
- InnateDB+ImmPort数据库 → 2660 immune genes
- 上述取交集 → 946 个,用于WGCNA鉴定与tumor相关的两个模块,取40 hub genes
- 2、建立预后模型
- 40个候选基因经cox回归,筛选得到24个
- 再经多变量回归,鉴定5基因组成的模型SHC1,WNT5A, NRP1, TGFA, IL4R
- 计算risk score,根据中位数分组,生存分析具有显著性
- 使用ICGC的生存数据做进一步验证
- 3、预后模型衍生分析
- risk score与其它临床指标的独立性
- risk score分组与通路GSEA的关系
- risk score分组与基因突变的关系
- risk score分组与免疫浸润的关系
- risk score分组与TIDE score的关系
【04】ccRCC与单基因
标题:PIK3C2A is a prognostic biomarker that is linked to immune infiltrates in kidney renal clear cell carcinoma
期刊|日期:Frontiers in Immunology | 30 March 2023
- PIK3C2A
- 1、初步表达分析
- 泛癌分析:差异表达、COX相关性(OS/DFS/PFS)
- TCGA KIRC分析(下调) + GEO验证 + HPA验证 + WB验证
- 单细胞分析:高表达数据类型/对照前后差异表达
- 2、临床预后分析
- 高低表达分组的生存分析/AUC
- 高低表达分组的差异临床指标分析
- 基因与其它临床指标的预后独立性
- 3、实验siRNA敲除后的影响
- 4、其它衍生分析
- 基因表达与免疫浸润的关系
- 基因表达与免疫细胞相关marker的关系
- 与PIK3C2A基因表达所有相关的基因-→通路
【05】PCa与巨噬细胞
标题:Multi-omics analysis reveals a macrophage-related marker gene signature for prognostic prediction, immune landscape, genomic heterogeneity, and drug choices in prostate cancer
期刊 | 日期:Frontiers in Immunology | 14 April 2023
- 1、筛选signature gene
- scRNA-seq
- macrophage cluster →307 marker genes
- Consensus分群 → C1 versus C2+C3
- 生存分析/临床指标/免疫功能/差异基因通路
- 38个 Cox genes
- macrophage cluster →307 marker genes
- TCGA → 18304 DEGs
- 21 交集基因,经lasso回归,9个基因
- scRNA-seq
- 2、基因层面分析
- scRNA seq细胞类型表达、RT-qPCR验证差异表达
- 基因表达与临床表型、CNV/SNP、ICB基因、免疫治疗IPS
- 3、risk score层面分析
- riskscore与临床表型、免疫浸润、肿瘤相关指标评价
- 根据中位数分为高低组
- 生存分析/AUC,差异分析/通路
- CNV/SNP,药物敏感度分析(分子对接)
- 构建诺模图及相关分析
- GEO数据验证
- GSE70768, GSE46602
- “inSilicoMerging”包去批次