A single-cell and spatially resolved atlas of human breast cancers
NatGenet. 2021 September

1、scRNAseq测序样本
文章对26个乳腺癌样本进行了单细胞测序,包括11 ER+,5 HER2+,10 TNBC。
对质控得到的130246个细胞根据marker基因注释为8种主要的细胞类型。
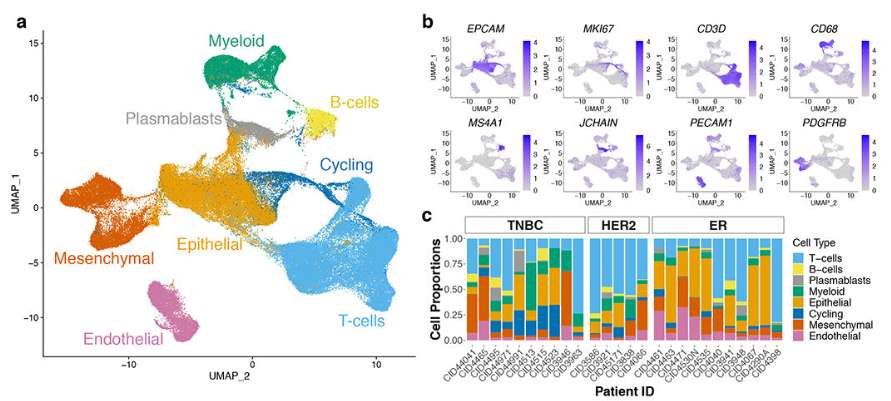
其中对于上皮细胞(epithelial cell)使用inferCNV鉴定出恶性肿瘤细胞与正常上皮细胞。
2、鉴定乳腺癌亚型
(1)使用下述方法将上述测序样本分别注释为4种亚型之一(LumA, LumB, Her2, Basal)
首先将每个样本的单细胞表达矩阵合并为pseudo-bulk:
- 方法1:应用PAM50分类器自动注释
- 方法2:与TCGA已注释亚型样本进行层级聚类,基于相似度注释
(2)将标注亚型的样本分为训练集与测试集。在训练集中,基于差异分析鉴定出每种亚型的marker基因,作为“scSubtype” gene signatures(Supplementary Table 4)
-
Basal_SC: 89
-
HER2E_SC: 102
-
LumA_SC: 46
-
LumB_SC: 65
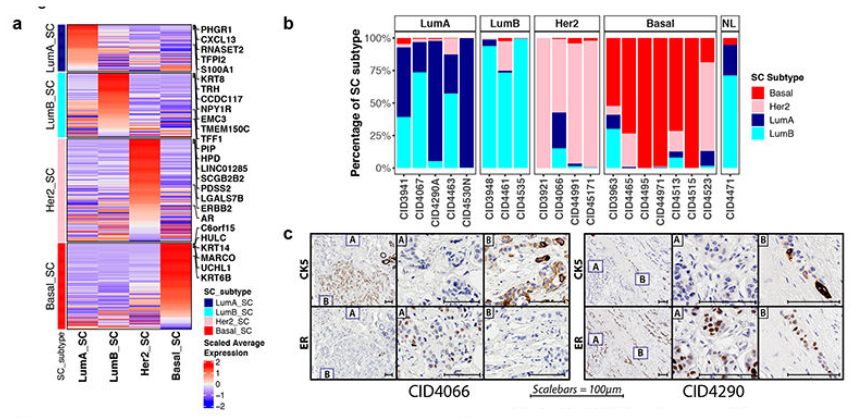
(3)使用上述的signatures对于单细胞表达矩阵对应的样本进行亚型注释
- 首先计算每个细胞对于4个signatures的表达分数,取最高的标签注释细胞;
- 然后取样本中大多数细胞注释标签注释为该样本的亚型。
结合测试集以及Bulk RNA-seq说明有较好的预测效果。
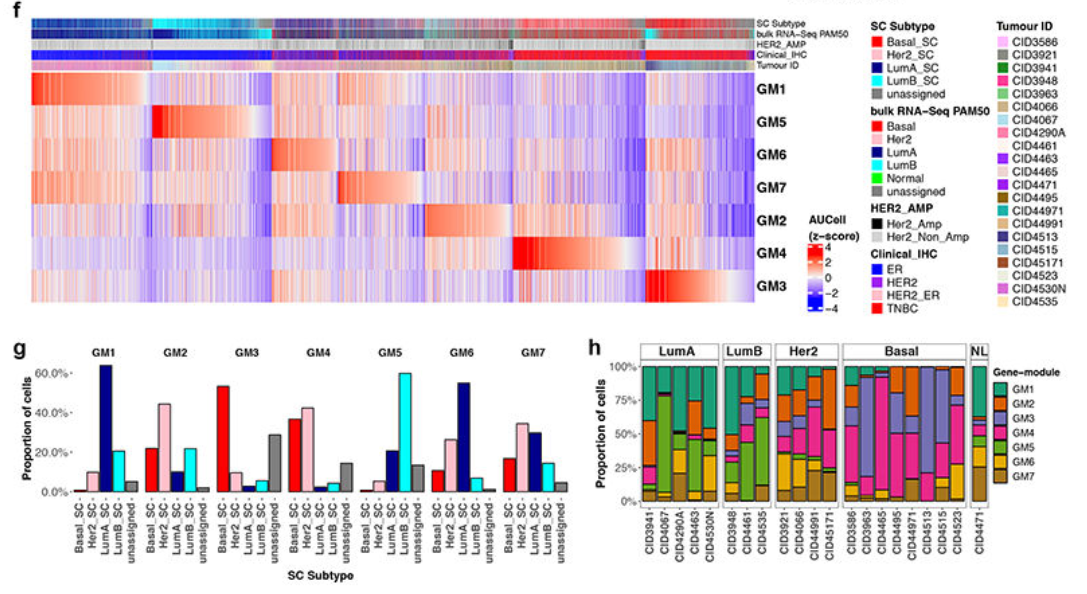
3、驱动肿瘤异质性的模块基因集
(1)对至少有50个肿瘤细胞的肿瘤样本单细胞表达矩阵进行基因的聚类分析(integrative clustering)得到574个 gene-signatures。
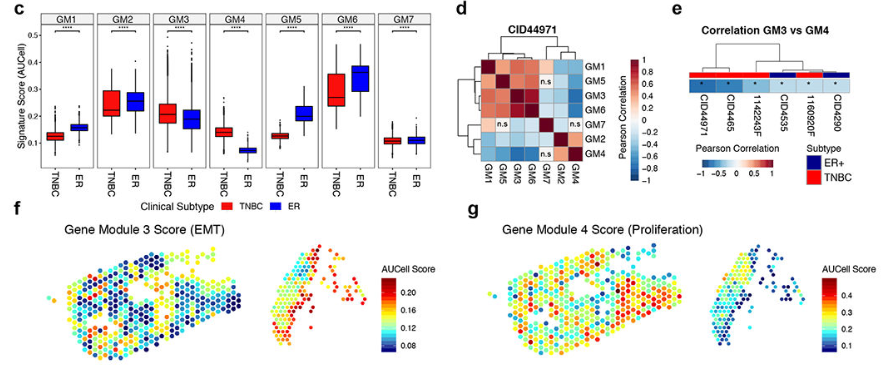
(2)基于gene-signature之间的Jaccard相似度系数,进一步聚类( Spherical k-means based consensus clustering)得到7个模块,对于模块取出现频率最高的200个基因。
(3)对于每个模块的组成基因进行通路富集分析;
(4)对于每个肿瘤细胞,计算对于7个模块的表达分数,再进行层级聚类,可以一定程度解释肿瘤异质性。
例如GM1 → LumA,GM2 → LumB
4、免疫微环境研究
这一步主要是对免疫细胞进行亚类分析,包括T细胞与固有免疫细胞(myloid、B、plasmablast)
此外对其中的4个样本进行CITE-seq测序进行细胞表面蛋白表达分子的辅助验证。
(1)将淋巴细胞分为了18个亚类,包括4个CD4亚群、5个CD8亚群等
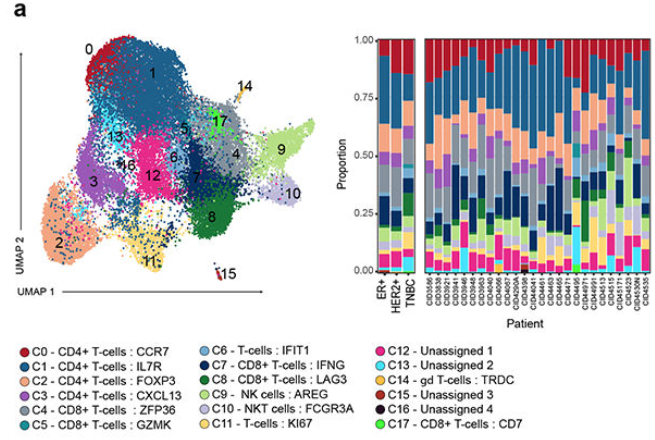
此外将B细胞分为两个亚类:naive and memory
(2)将Myeloid细胞分为了13个亚群,包括3个Monocytes亚群、6个Macrophages亚群等。
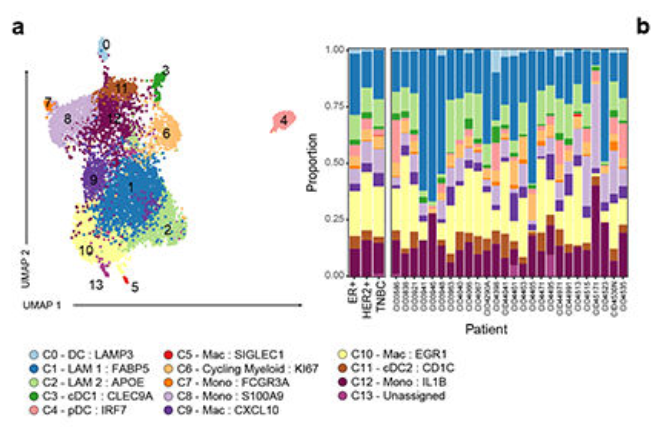
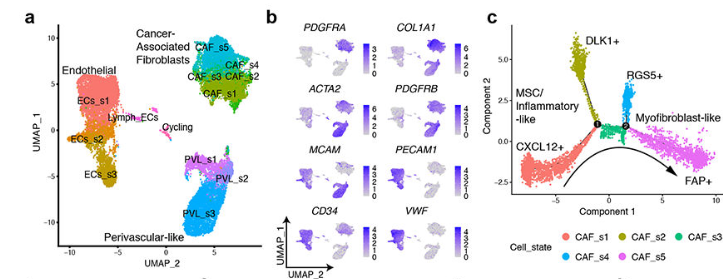
(3)Stromal细胞分为3大类:CAFs, perivascular-like cells(PCL), endothelial cells,分别从轨迹分析解读不同发育阶段特征。
5、空间转录组解释乳腺癌异质性
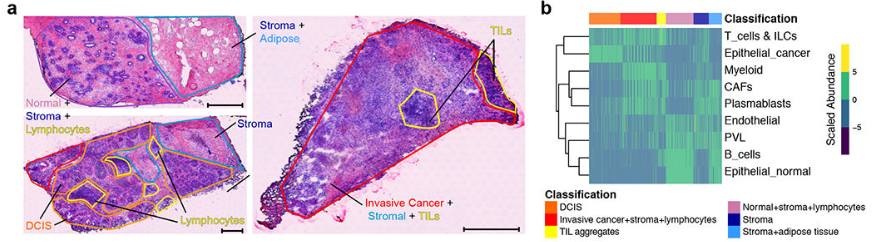
(1)对于6个乳腺癌样本进行空间转录组测序,其中前4个属于于上述单细胞测序的样本(2 ER+, 2 TNBC);另外2个来自外部数据(TNBC)。
(2)使用Stereoscope模型对每个spot进行细胞类型注释,结果与病理切片注释大致相同。
(3)计算上述得到的7个模块在空间转录组spot表达特征,例如模块与亚型的差异性,模块与模块的相关性等。
6、发现细胞间相互作用
主要阐述此前研究较少的基质细胞与免疫细胞相互作用情况
(1)主要计算方式是基于spot中两两细胞类型表达比例的相关性
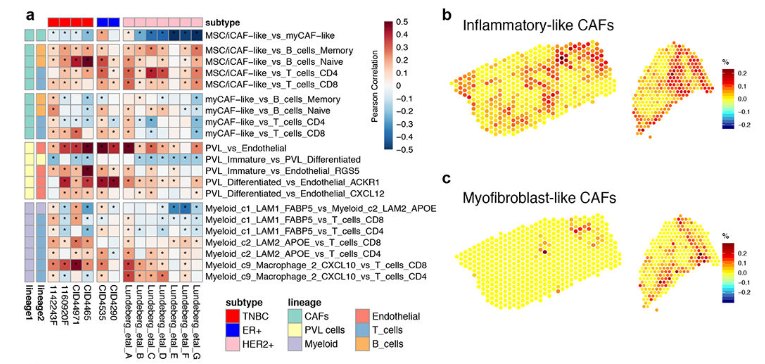
例如 iCAF与MyCAF存在负相关性;iCAF/MyCAF与淋巴细胞存在明显的相关性。
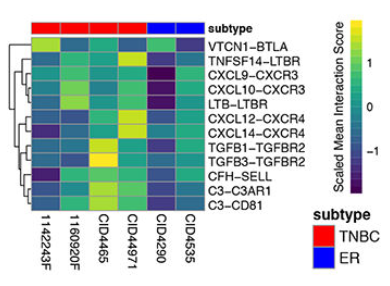
(2)通过计算高表达配(受)体iCAF spot与高表达受(配)体的CD4/CD8 spot在空间转录组上的距离进一步验证这两类细胞所存在的相互作用类型。
7、定义乳腺癌新的亚型
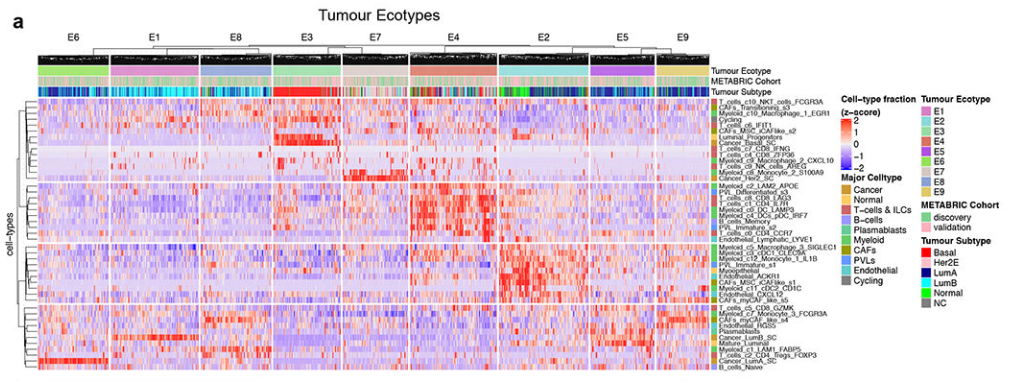
(1)根据上述的再聚类结果,共得到乳腺癌的45种亚群以及相应的gene signatures。
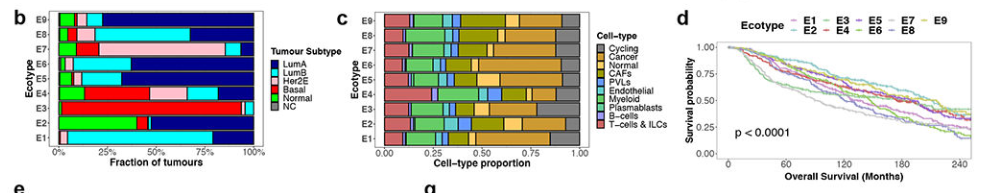
(2)对METABRIC的BC bulk RNA-seq队列使用CIBERSOFTx算法,基于上述的signatures计算每个样本的45种细胞亚群的比例。然后进一步聚类得到9类肿瘤亚类。即每一类肿瘤都有相似的细胞类型组成模式。
(3)结合肿瘤样本的生存资料,进一步分析较好/较差预后肿瘤亚类,以及对应的细胞类型组成特点。
8、关于数据获取
(1)文章对于数据可视化制作的网页
(2)单细胞以及Bulk RNAseq测序数据:
- Processed:GSE176078
- Raw: EGAS00001005173
(3)空间转录组数据
-
自测 – DOI: 10.5281/zenodo.4739739
-
外部 – DOI: 10.5281/zenodo.3957257
(4)文章分析涉及的代码脚本
https://github.com/Swarbricklab-code/BrCa_cell_atlas
以上是我自己对于这篇文章中要点所做的简单概述。其中每一点,对应原文都有较详细的阐述,例如特定细胞亚群的特征与作用。
作为一篇体量较大的资源型文章,在日后涉及乳腺癌特定细胞类型的相关研究时,都可以使用这个数据集进行初步尝试与探索或者补充验证。