A single-cell analysis of breast cancer cell lines to study tumour heterogeneity and drug response
Nature Communications | 31 March 2022
DOI: https://doi.org/10.1038/s41467-022-29358-6

这篇文章对来自32个乳腺癌细胞系的35276个细胞进行了单细胞转录组测序(Drop-seq),数据已上传到 GSE173634。文章具体内容主要是针对测序结果,从多个角度展开衍生的分析。
https://figshare.com/articles/dataset/Single_Cell_Breast_Cancer_cell-line_Atlas/15022698
32种细胞系的分型信息如下,其中MCF12A为non-cancer细胞系。
| CCLs | ER | PR | HER2 | Subtype | |
|---|---|---|---|---|---|
| 1 | AU565 | - | - | + | H |
| 2 | BT20 | - | - | - | TNA |
| 3 | BT474 | + | + | + | LB |
| 4 | BT483 | + | +/- | - | LA |
| 5 | BT549 | - | - | - | TNB |
| 6 | CAL51 | - | - | - | TNB |
| 7 | CAL851 | - | - | - | TNB |
| 8 | CAMA1 | + | +/- | - | LA |
| 9 | DU4475 | - | - | - | TNA |
| 10 | EFM19 | + | + | - | LA |
| 11 | EVSAT | - | +/- | + | H |
| 12 | HCC1143 | - | - | - | TNA |
| 13 | HCC1187 | - | - | - | TNA |
| 14 | HCC1500 | + | +/- | - | LA |
| 15 | HCC1937 | - | - | - | TNA |
| 16 | HCC1954 | - | - | + | H |
| 17 | HCC38 | - | - | - | TNB |
| 18 | HCC70 | - | - | - | TNA |
| 19 | HDQP1 | - | - | - | TNB |
| 20 | HS578T | - | - | - | TNB |
| 21 | JIMT1 | - | - | + | H |
| 22 | KPL1 | + | - | - | LA |
| 23 | MCF12A | - | - | - | Basal-like |
| 24 | MCF7 | + | + | - | LA |
| 25 | MDAMB361 | + | +/- | + | LB |
| 26 | MDAMB415 | + | +/- | - | LA |
| 27 | MDAMB436 | - | - | - | TNA |
| 28 | MDAMB453 | - | - | + | H |
| 29 | MDAMB468 | - | - | - | TNA |
| 30 | MX1 | - | - | - | TNB |
| 31 | T47D | + | + | - | LA |
| 32 | ZR751 | + | +/- | - | LA |
1、BC细胞系scRNAseq图谱概况
32种细胞系的35276个单细胞测序结果
- 平均每个细胞系1069个细胞
- 平均每个细胞3248个基因表达信息
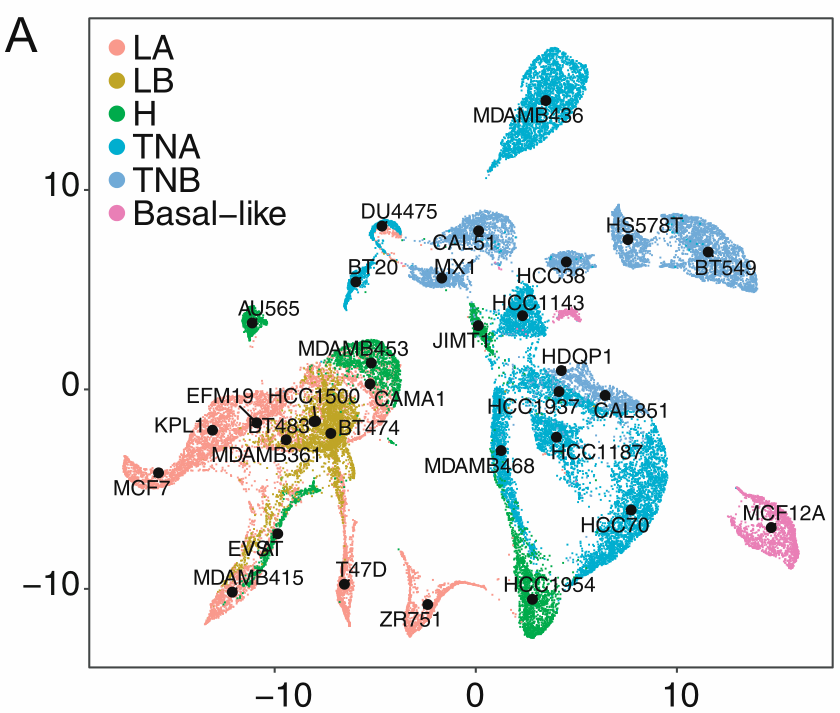
(1)如下图,对单细胞数据的降维可视化发现不同亚型的细胞系可基本被分开。
- 其中Luminal super-cluster混杂部分Her2亚型
- TNBC被分为2个不同的super-cluster(TNA、TNB)
(2)不同细胞系对于ESR1、ERBB2、PGR、EGFR以及MKI67等5种经典标志物基因的表达情况
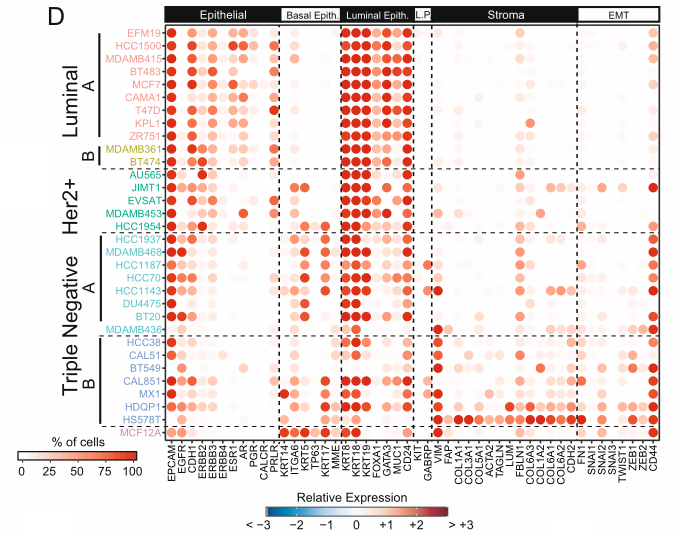
(3)不同细胞系对于48个基于文献的标志物基因的表达情况。
2、聚类分群发现新的BC标志物基因
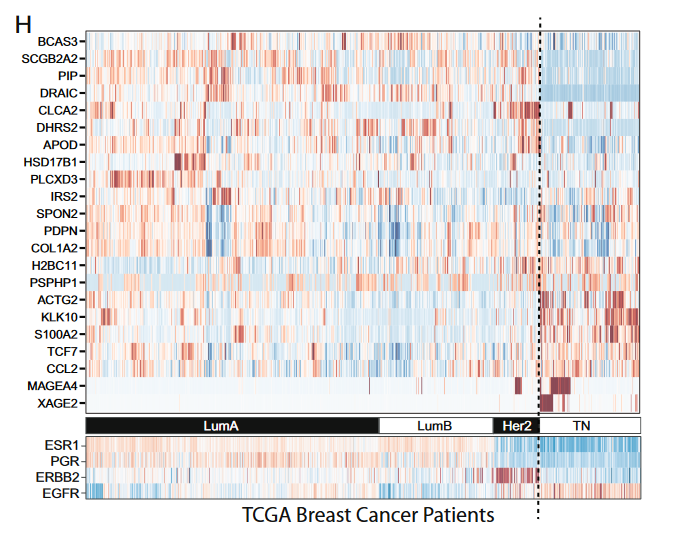
将325276个细胞经聚类、分为22个cluster。
从每一个cluster中,挑选一个cluster marker gene(例如相较其它cluster最差异表达的)。
- 首先经文献查询,这22个marker gene具有研究的价值。
- 观察TCGA的937病人样本的表达数据中这22个基因的表达情况,并结合病人的BC注释分型信息,分析差异表达(如下图所示)。
- 最后,文章测试了这个22个基因用于区分TCGA样本亚型的能力,并与PAM50进行比较。
3、根据细胞系结果分析BC病人测序样本的异质性
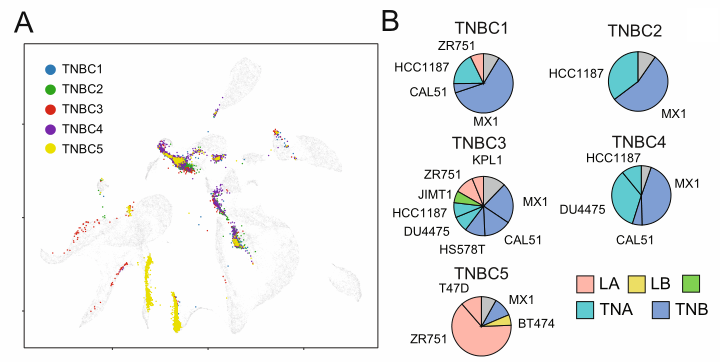
(1)对于BC病人的scRNAseq测序结果,分析每个细胞与哪种BC细胞系最相似。
分析过程本质上是分类预测,判断样本的一个细胞属于哪一种乳腺癌细胞系。
Mapping算法大致如下图所示,核心是利用UMAP降维转换以及KNN聚类算法。
- 首先基于细胞系数据做测试,分为训练集与验证集,正确率在75%以上
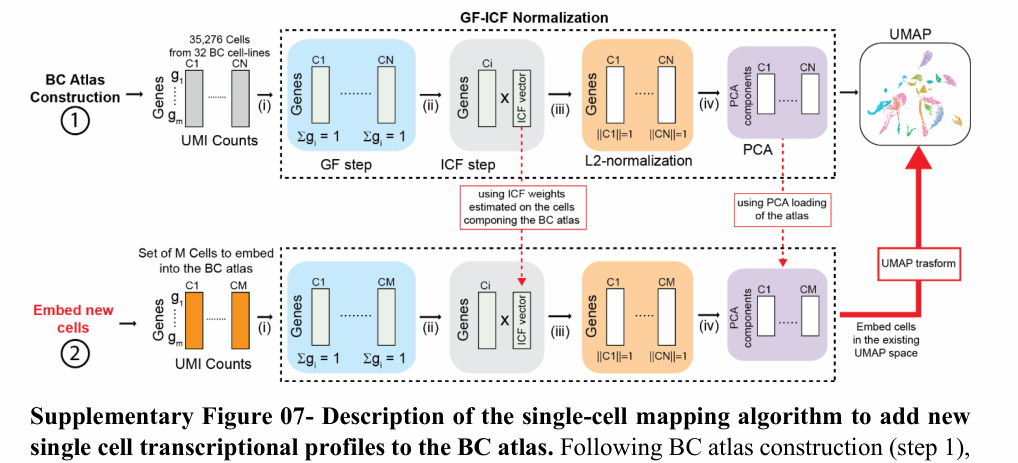
- 然后从GEO(GSE173634)中获得5个TNBC样本的scRNAseq结果以及BC空间转录组数据进行测试。
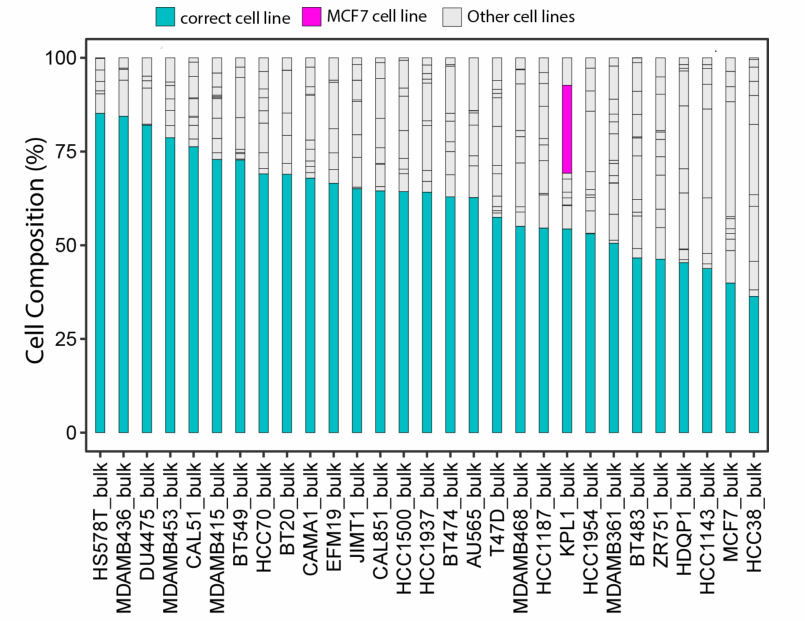
(2)对于BC病人的bulk RNAseq测序结果,使用Bisque工具预测样本的细胞系组成比例。
- 首先对CCLE中BC细胞系的bulk RNAseq数据进行验证,正确率在40%~80%
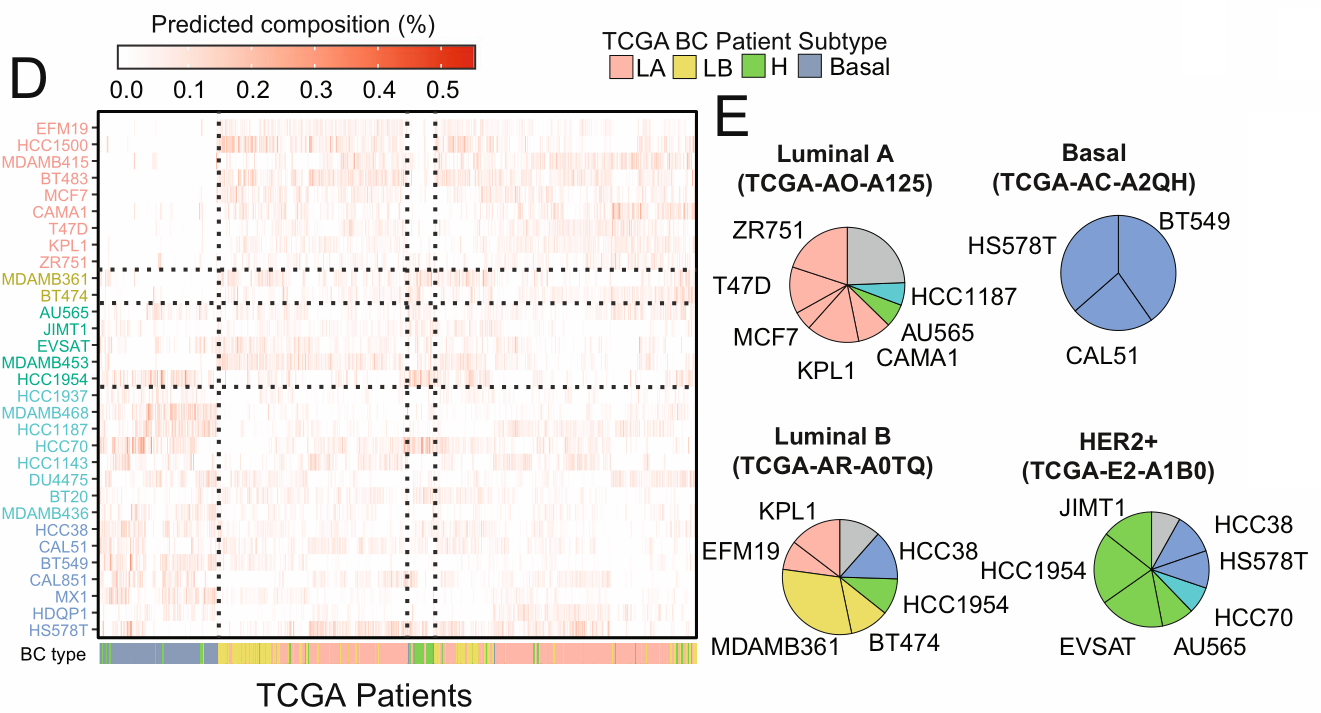
- 然后对TCGA BRCA的表达数据,预测样本的细胞系组成,对比样本本身的亚型注释信息验证准确性。
4、单细胞水平的药物敏感度预测
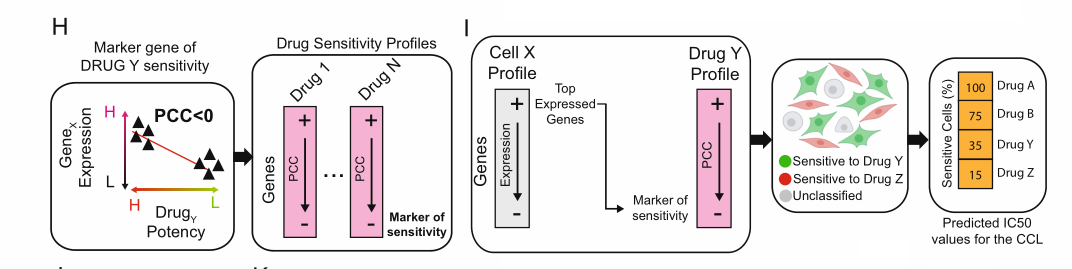
(1)从CTRPv2数据库收集得到450个药物作用于658个癌症细胞系的药物敏感度数据;
- 其值越小,表明细胞系对于该药物越敏感(药物越有效)
(2)对于一个药物的658个细胞系的敏感度实验结果与基因在这658个细胞系的表达情况做相关性分析(PCC)。
-
如果二者呈负相关,说明细胞系中的该基因表达水平越高,药物该细胞系效果越显著。
-
因此对于每一个药物都可以得到一个相关系数为排名依据(由高到低)的基因列表。
(3)对于一种细胞系中每一个细胞的测序结果,取Top250高表达基因,分别对上述450个drug gene list做GSEA富集分析,如果ES显著小于0,表明该药物可能对这个细胞会有良好的效果。
- 因此可以预测出一种细胞系中不同种细胞可能有不同的适应药物。
(4)文章把上述的方法包装为一个工具,为DREEP (DRug Estimation from single-cell Expression Profiles),详见https://github.com/dibbelab/singlecell_bcatlas
(5)最后文章以MDA-MB-361细胞系的单细胞数据进行示例分析以及药物实验验证。
以上是文章的简要整理,其中分析部分还有一大点涉及细胞系的基因表达异质性与动态性,尚未完全理解,因此没有整理。
关于这篇文章,最重要的还是提供了全面的乳腺癌细胞系的单细胞转录组数据。其次将基因表达与药物敏感性进行关联的药物发现思路值得借鉴与学习。