Discovery of drug mode of action and drug repositioning from transcriptional responses
August 2, 2010 | PNAS | IF=11.2
10.1073/pnas.1000138107

1、合并PRL
cMap 1309种化合物作用于5种细胞系的6100个干扰转录组的差异基因结果。对于同一种种化合物由于不同浓度,或者作用于不同细胞系会得到多个差异基因列表(下面简称list)。
为了得到一种化合物的综合差异基因列表,通过合并多个原始的list(由up到down的降序ranked list),得到Prototype Ranked List (PRL)。
(1)计算同一化合物所有list两两间的相似性,文章具体使用的是Spearman’s Footrule系数。
(2)对于最相似的两个list,使用Borda Merging Method方法合并产生一个新的list,代替原有旧的两个list。
(3)重复上述步骤(1)、(2)直至只剩下一个list,即为该化合物的PRL。
对于PRL排名最靠前的基因,表示被化合物显著上调的基因;排名靠后的基因,表示被化合物显著下调的基因。
RankAggreg包
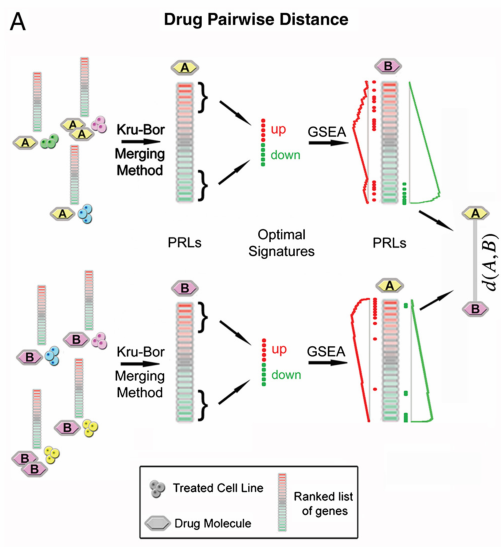
2、计算PRL距离
基于富集分析原理,计算两个PRL的距离(相似性)
如下公式:对于药物d的PRL,p表示排名靠前的Top N个基因,q表示排名靠后的Last N个基因。
分别计算p与q相对于药物x的PRL富集分数(Enrichment score)
- ES>0, 表示富集到PRL的排名靠前部分
- ES<0, 表示富集到PRL的排名靠后部分
如果对于p的ES接近1,q的ES接近-1,则TES结果接近0,那么说明药物d的PRL距药物x的PRL较近(相似)。
如果对于p的ES接近-1,q的ES接近1,则TES结果接近2,那么说明药物d的PRL距药物x的PRL较远(不相似)。 $$ TES_{d,x} = 1-\frac{ES^p_x-ES^q_x}{2} $$ 同理可计算药物x的PRL距药物d的PRL的远近程度。
药物d至x的距离不一定等于药物x至d的距离。
最后有两种方式评价两个药物的综合距离
(1)平均TES $$ D = \frac{TES_{A,B}+TES_{B,A}}{2} $$ (2)最近距离 $$ D = \frac{min(TES_{A,B},TES_{B,A})}{2} $$
3、构建网络并分群
3.1 构建网络
- 根据所有化合物间两两距离(1309种化合物的856086个两两距离),筛选Top5%的距离关系。(Top5%聚类作为threshold)
- 然后构建有1302个药物作为节点,41047条距离作为权重的边的Drug Network。
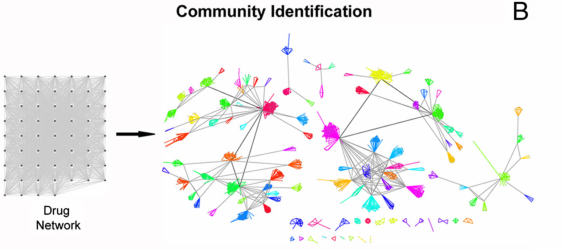
3.2 分群鉴定
- 基于 affinity propagation算法,对Drug Network进行分群,鉴定community以及每个community的exemplar核心节点。
- 共鉴定得到106个community
R包apcluster可实现affinity propagation算法:https://cran.r-project.org/web/packages/apcluster/vignettes/apcluster.pdf
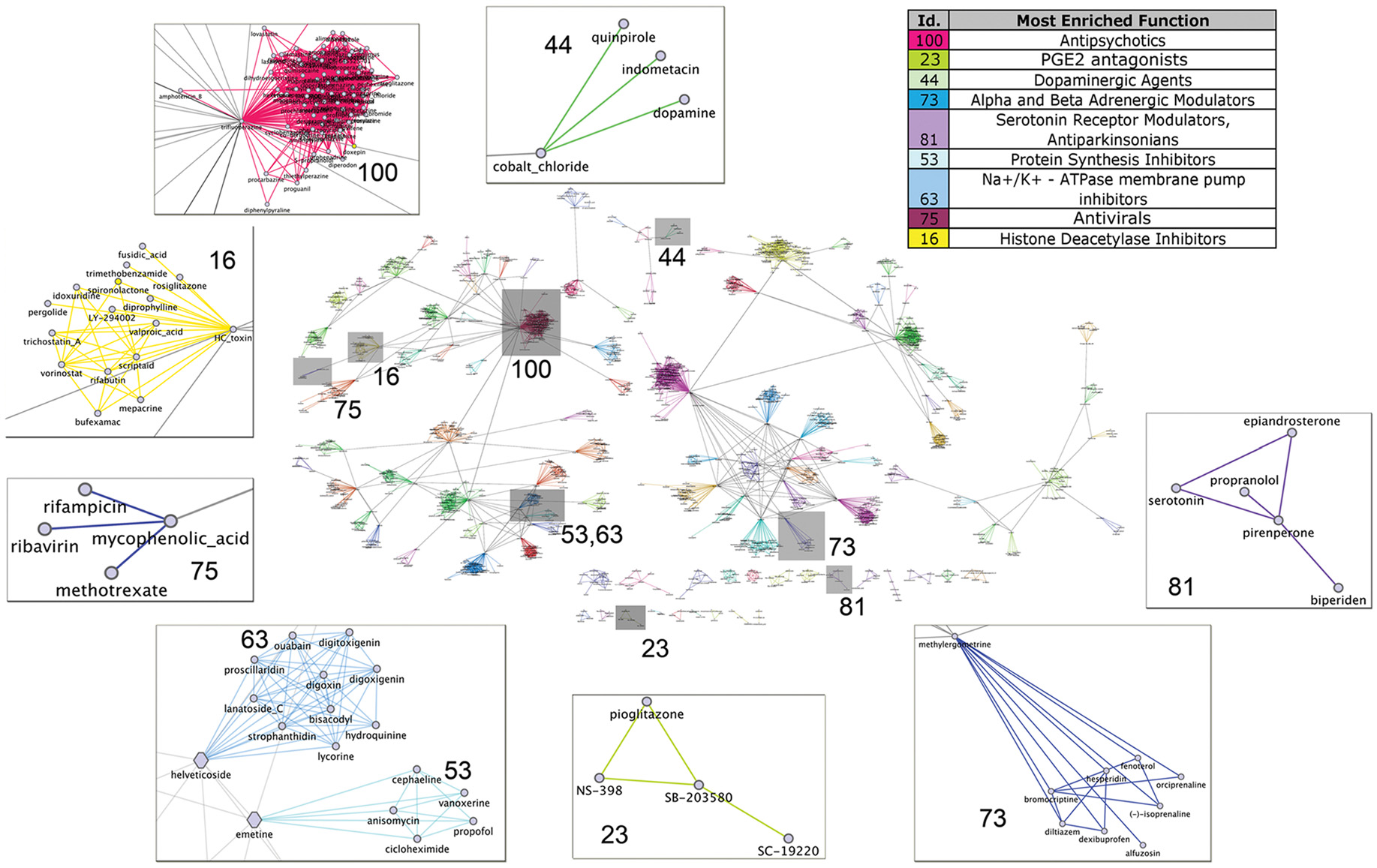
3.3 分群验证
(1)在同一群的药物是否具有相同的MOA(包括ATC、Target、pathway)以随机分布作为背景,计算odd ratios
- 共有77个community具有显著富集的MOA(odd ratios>1)
(2)文章进一步说明了具有相同的MOA的化合物是否分到一个community里
4、小分子MOA预测
4.1 预测流程
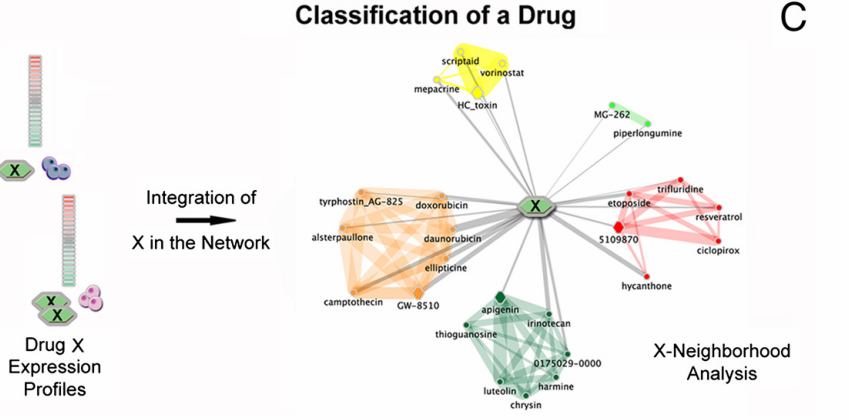
在如上建立了drug network并鉴定出具有MOA意义的community之后,可通过此网络预测新的小分子的MOA。
(1)得到新的小分子干扰实验的PRL
(2)计算该小分子与网络中所有节点的距离,保留距离低于阈值的边。
(3)计算该小分子分别与网络中所有community的距离→weighted geometric average of the distances
- 如下公式,计算药物x到与community C的距离,|Cx|表示community C的组成节点数。
$$
\sqrt[|C_x|]{\Pi_{d\in C_x} D(d,x)/|C_x|}
$$
4.2 可行性验证
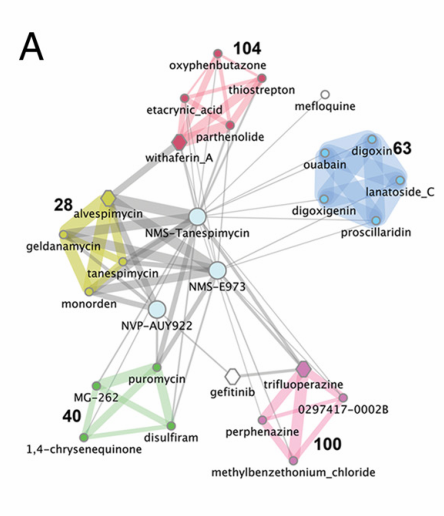
文章以9个已知MOA的化合物作为输入,用于验证可行性。
如下图所示,三个测试化合物MMS-Tanespimycin、NMS-E973、NVP-AUY922为已知的HSP90抑制剂。
根据上述方式计算,相距最近的community为 n.28,该community主要有HSP900抑制剂组成;其次接近的为n40、n104。
最后也通过WB实验验证了上述预测的预测结果。