(1)Computational Drug Repurposing for Alzheimer’s Disease Using Risk Genes From GWAS and Single-Cell RNA Sequencing Studies
https://www.frontiersin.org/articles/10.3389/fphar.2021.617537/full
Front Pharmacol, 2021/06, IF 5.81
(2)gene2drug: a computational tool for pathway-based rational drug repositioning
https://academic.oup.com/bioinformatics/article/34/9/1498/4721786
Bioinformatics,2018/15, IF 6.92
(3)Drug-set enrichment analysis: a novel tool to investigate drug mode of action
https://academic.oup.com/bioinformatics/article/32/2/235/1743543
Bioinformatics,2016/01, IF 6.92
一、DSEA与gene2drug
Napolitano等人分别于2016年、2018年在Bioinformatics提出了DSEA与gene2drug方法,分别回答了如下两个问题。
1、功能简介
(1)DSEA:给出特定的几个类似的药物,发现这些药物可以特定作用于哪些通路? https://dsea.tigem.it/
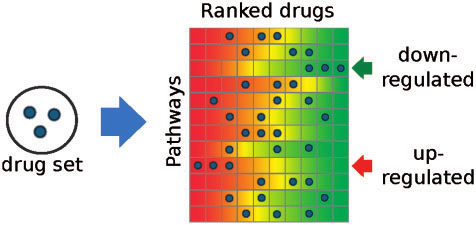
(2)gene2drug:给出特定的几个通路,发现这些通路可以被哪些药物所调控?https://gene2drug.tigem.it/
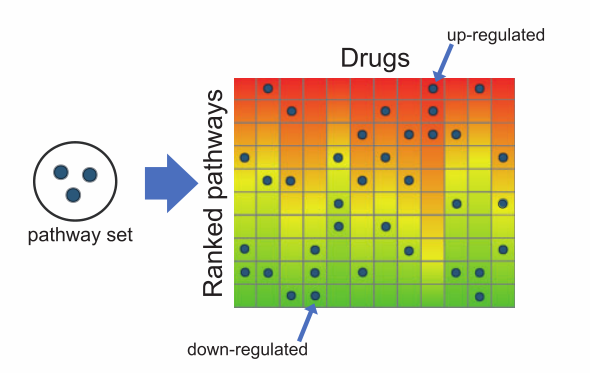
目前已经提供了
gep2pep的bioconductor包同时支持上述的两种分析,分别称为CondSEA与PathSEA。https://bioconductor.org/packages/release/bioc/vignettes/gep2pep/inst/doc/vignette.html
2、数据与计算方法
- 通路数据主要收集在MsigDB的8类基因集(DSEA),DSEA与gene2drug文章所使用的数据略有差异。
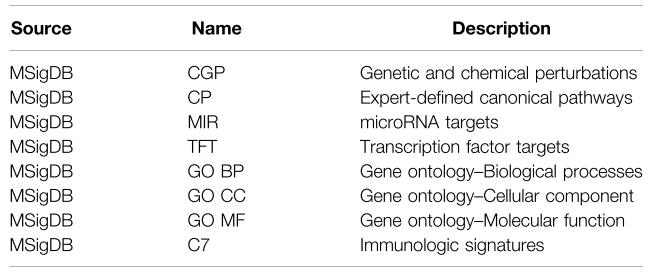
-
药物干扰数据主要收集自Cmap的1309个 Ranked gene list(First–most up; Last–most down),分别对应1309个compound。
-
使用Kolmogorov–Smirnov (KS) test方法(https://github.com/franapoli/signed-ks-test),计算每个药物对于所有通路的GSEA分析,得到富集分数与P值。富集分数范围在-1~1之间,越接近1表示药物可正向调控这个通路。
3、两次GSEA分析
如上本质上是将药物–基因差异表达矩阵转为药物–通路调控矩阵。
DSEA与gene2drug方法本质都是基于药物–通路调控矩阵的再一次GSAE分析。
-
对于DSEA,则是通路为主体的ranked list。First 是最正向调控该通路的药物,Last是最负向调控该通路的药物。
在用户分析时,提供若干感兴趣的药物,发现整体上这些输入药物所正向/负向调控的通路。
-
对于gene2drug,则是药物为主体的ranked list。First是该药物最正向调控的通路,Last是最负向调控的通路。
在用户分析时,提供若干感兴趣的通路,发现整体上能够正向/负向这些输入通路的药物。

二、基于通路的AD药物重定向

1、收集基因list
1.1 GWAS risk gene
GWAS(genome-wide association analyses)全基因组关联分析通过SNP位点与病人表型信息鉴定疾病相关的risk gene
作者参考Jansen等人2019年的大型AD GWAS研究(https://doi.org/10.1038/s41588-018-0311-9)收集到255个late-onset AD的风险基因【Table S1A】
1.2 scRNAseq DEG
scRNAseq单细胞数据可进行细胞类型水平的差异基因分析。
作者参考Mathys等人2019年的大型AD单细胞数据研究(https://doi.org/10.1038/s41586-019-1195-2)收集得到717个来自3种细胞类型在AD early与late间的差异基因。【Table S1】
- Asr,astrocytes,星形胶质细胞–532;
- Ex,excitatory neuronal,兴奋神经元–103;
- Oli,oligodendrocytes,少突胶质细胞–82
2、预分析
2.1 富集分析
对上述的4个基因list使用Enrichr工具进行富集分析(GO BP)
2.2 靶点交集分析
根据Cummings等人2019年的AD研发药物综述(https://doi.org/10.1016/j.trci.2019.05.008)总结的处于phase 1, 2, and 3阶段的药物,在TTD、DrugBank、PubChem数据库收集药物的作用靶点。【Table S2】
| Phase | Total target genes | Unique target genes | Total number of pathways (adjusted p-value < 0.05) |
|---|---|---|---|
| Phase 3 | 49 | 37 | 33 |
| Phase 2 | 110 | 90 | 67 |
| Phase 1 | 34 | 23 | 21 |
结合上述3类药物的靶点信息,与之前得到的4个基因list做交集分析。结果发现
- GWAS 基因list与之有5个基因交集(TREM2, CD33, CHRNA2, PRSS8, and ACE)
- Ex差异基因list与之有3个基因交集(TKT, APP, and GABRA1)
- 其它两个基因list无交集基因。
The analysis suggests that other drugs targeted these AD-associated risk genes ca potentially be repurposed for AD.
3、gene2drug分析
- 4个 如上所述的基因list、8个 通路基因集
- 使用 gep2pep包计算每个基因list中的基因在每个基因集的相关通路,然后进行PathSEA分析,得到以P值作为排序依据的Ranked Drug list。
- 每个基因list对于每个通路基因集都得到一个drug list,共32个drug list。
- 由于CP、MIR、TFT通路基因集得到drug list包含较少的药物结果,所以剔除,保留20个
- 使用adjusted p-values,将每个基因list的5个drug list合并为一个drug list
- 挑选出每个drug list的Top5 drug,共20个候选药物
- 文献验证
- PubMed:收集药物的有效性、作用机制研究
- PubChem:确定药物的毒性与否
- ClinicalTrials:查看药物的临床研究情况

