1、算法简介
1.1 关系拆解
通过计算方法( 例如相关性Pearson correlation, 互信息mutual information等)构建的相互关系网络中,节点两两之间的关系通常包括直接关系与间接关系两部分,即如下图所示Total = Direct + Indirect
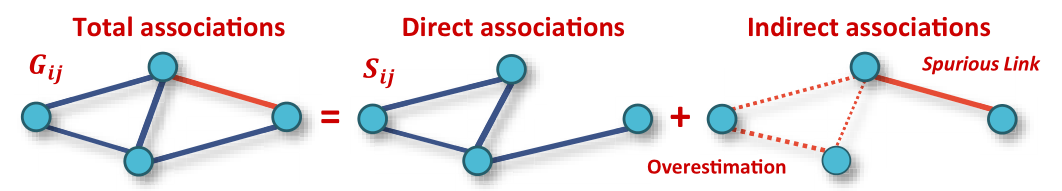
PNAS的一篇文章提供了一种从计算网络中鉴定节点之间direct association的新方法–iDIRECT(Inference of Direct and Indirect Relationships with Effective Copula-based Transitivity)
Disentangling direct from indirect relationships in association networks
PNAS 2022 Jan 11
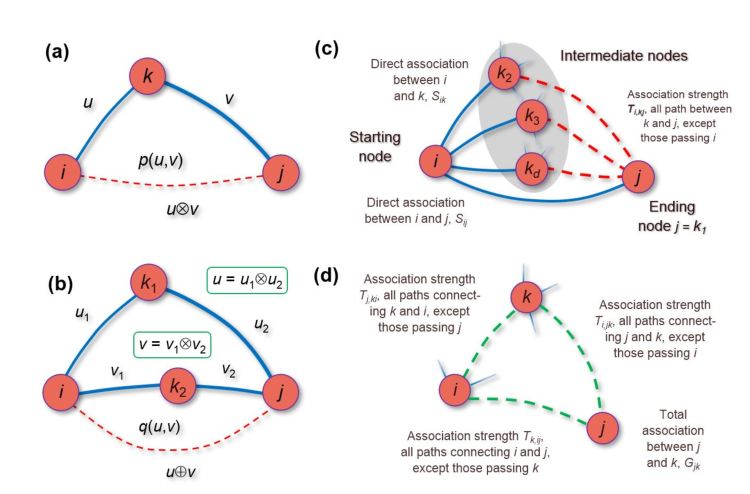
- 对于Sequential paths(如下图A),即两个节点之间存在1至多个中间节点连接二者。可通过简单的关系乘法计算这两个节点在这条path的indirect关系。
$$ u \otimes v = uv $$
- 对于parallel paths中(如下图B),即两个节点之间存在多条path。此时并没有简单的相加,而是采用Archimedean copulas计算 combined probability。从而保证
u⊕v ∈ [0,1] for all u,v ∈ [0,1]。其中u、v表示每条path的indirect association
$$ u\oplus v = \frac{u+v-2uv}{1-uv} $$
- 对于存在direct与indirect association的节点对。其中G表示observation association;S表示direct association
$$ G_{ij} = S_{ij}\oplus S_{ik_2}G_{k_2j} \oplus S_{ik_3}G_{k_3j} \oplus … \oplus S_{ik_d}G_{k_dj} $$
- 文章对上式做了进一步的改进:在计算节点i的邻居k(假如为k2)与节点j的关系时,排除self-looping path(k-i-j)后再进行计算k2与j的total association。
$$ G_{ij} = S_{ij}\oplus S_{ik_2}T_{i,k_2j} \oplus S_{ik_3}T_{i,k_3j} \oplus … \oplus S_{ik_d}T_{i,k_dj} $$
1.2 算法对比
文中提到之前已经有人提出相关方法,例如network deconvolution (ND)、global silencing (GS)等
作者认为以前的方法中存在3点不足
-
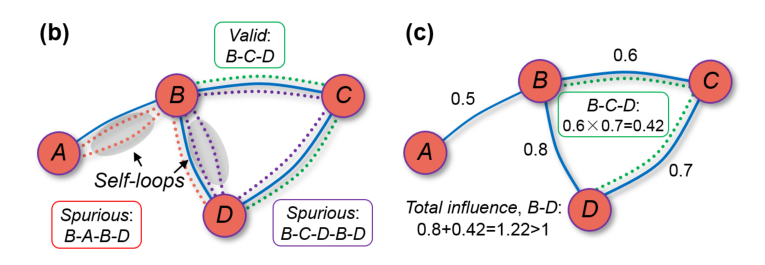
(1)interaction strength overflow:对于parallel path采用简单的相加,可能会存在 over-estimation。
- 本文采用了Archimedean copulas方式计算多条path之和(如上图b所示)
-
(2)self-looping:对于indirect association,考虑了包含自身的冗余路径。
- 本文提出在计算indirect path时,一种计算transitivity matrix的新思路,用以去冗余。(如上图c所示)
-
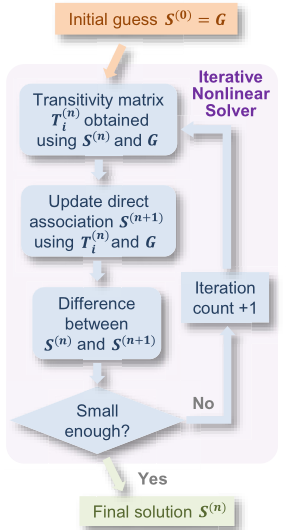
(3) ill-conditioning:对于这一点,暂时没有太理解。作者认为ND、GS等方法求解 direct association时借助转置矩阵
G-1。但由于关系矩阵一般近奇异,转置操作具有高度不确定性。而本文使用了称为nonlinear solver方法,进行推算迭代、直至稳定。Specifically, ill-conditioning means that the association matrix is close to singular and is highly unreliable to invert.
2、Python脚本
作者已将代码封装为Python脚本,可直接使用。
- https://github.com/nxiao6gt/iDIRECT
- 为了适应不同python版本,作者提供了对应python版本的脚本文件
|
|
3、应用示例
在文章Result中除了第一部分简单介绍了iDIRECT的算法原理,后续部分都是通过在不同类型/背景网络中的应用(同时对比其它算法)说明本方法的优势。
简单介绍一下在优化基因调控网络关系中的示例:
3.1 DREAM5
DREAM5有一项重构基因网络挑战项目,其中有一个模拟的基因表达数据(GeneNet-Weaver (GNW) version 3.0),同时给出了gold-standard 基因调控关系(0/1)。参赛者采用不同的计算方法试图重构网络,然后DREAM5基于gold-standard对预测结果进行评价。
-
评价方式是从0到1遍历不同cut-off得到的true-positive rate/false-negative rate得到的AUPR与AUROC。
-
然后进行随机1,000 random simulations,得到的AUPR与AUROC分布,作为背景,从而计算上述方法得到的AUPR与AUROC的P值
原文描述为:
Those links were then scored based on the true network using the Challenge organizer’s script (details in Mate- rials and Methods), which was just -log(p) of the empirical P-values of the predicted AUPR from 1,000 random simulations.
3.2 iDIRECT优化
- 文章对于参赛者已提交的、基于不同算法得到network使用iDIRECT,以期望从中鉴定出direct association;
- 同时对比ND与GS两种方法的结果;
- 在结果评价上,同时考虑了AUPR与AUROC两个指标
$$ \Theta_{PR} = -log_{10}P_{PR} $$
$$ \Theta_{ROC} = -log_{10}P_{ROC} $$
$$ \Theta_{total} = (\Theta_{PR} + \Theta_{ROC})/2 $$
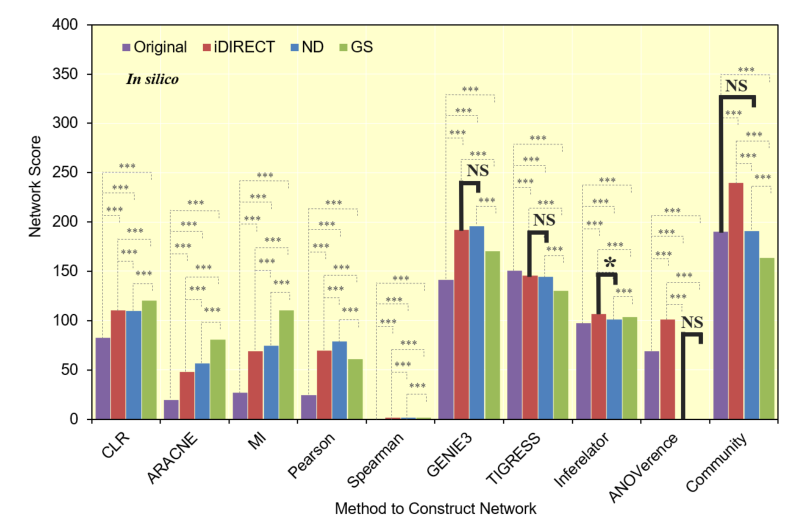
AUPR represents the average precision when recall (true-positive rate) varies from zero to one;
AUROC represents the average true positive rate when the false-negative rate varies from zero to one.