本次的两篇文章属于同一团队,第一篇文章侧重提出计算方法;第二篇文章侧重于应用方法,发现生物学规律
Paper1:提出方法
SIGN: similarity identification in gene expression
Bioinformatics 2019 / 2 IF = 7

1.1 TSC score
文章根据2009年学者提出的modified RV coefficient,使用transcriptional similarity coefficient(TSC)分数,用以表征两个矩阵的相似性。计算公式如下:其中P1矩阵与P2矩阵的纵轴(Gene Row)需要保持一致,而两个矩阵的样本数(Sample Column)没有要求。 $$ TSC(P_1,P_2) = \frac{\sum_{i}(P_{10}×P_{20}){ij}} {{\sqrt{\sum{ij}(P_{10}){ij}^2}}× {\sqrt{\sum{ij}(P_{20})_{ij}^2}}} $$
- 其中
Pm0的计算公式如下。P'表示原始P矩阵的转置矩阵,Diagonal()方法仅保存原始矩阵的主对角线值,其余值标记为0。
$$ P_{m0} = P_m × P’_m - Diagonal(P_m × P’_m) $$
计算的TSC score范围在[-1, 1]之间,值越高表明两个样本(群)具有相同的基因表达模式。
根据RV coefficient,符合以下特征规律:
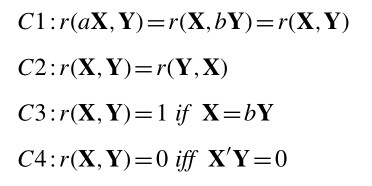
举例计算,实现需要了解矩阵乘法的原理。

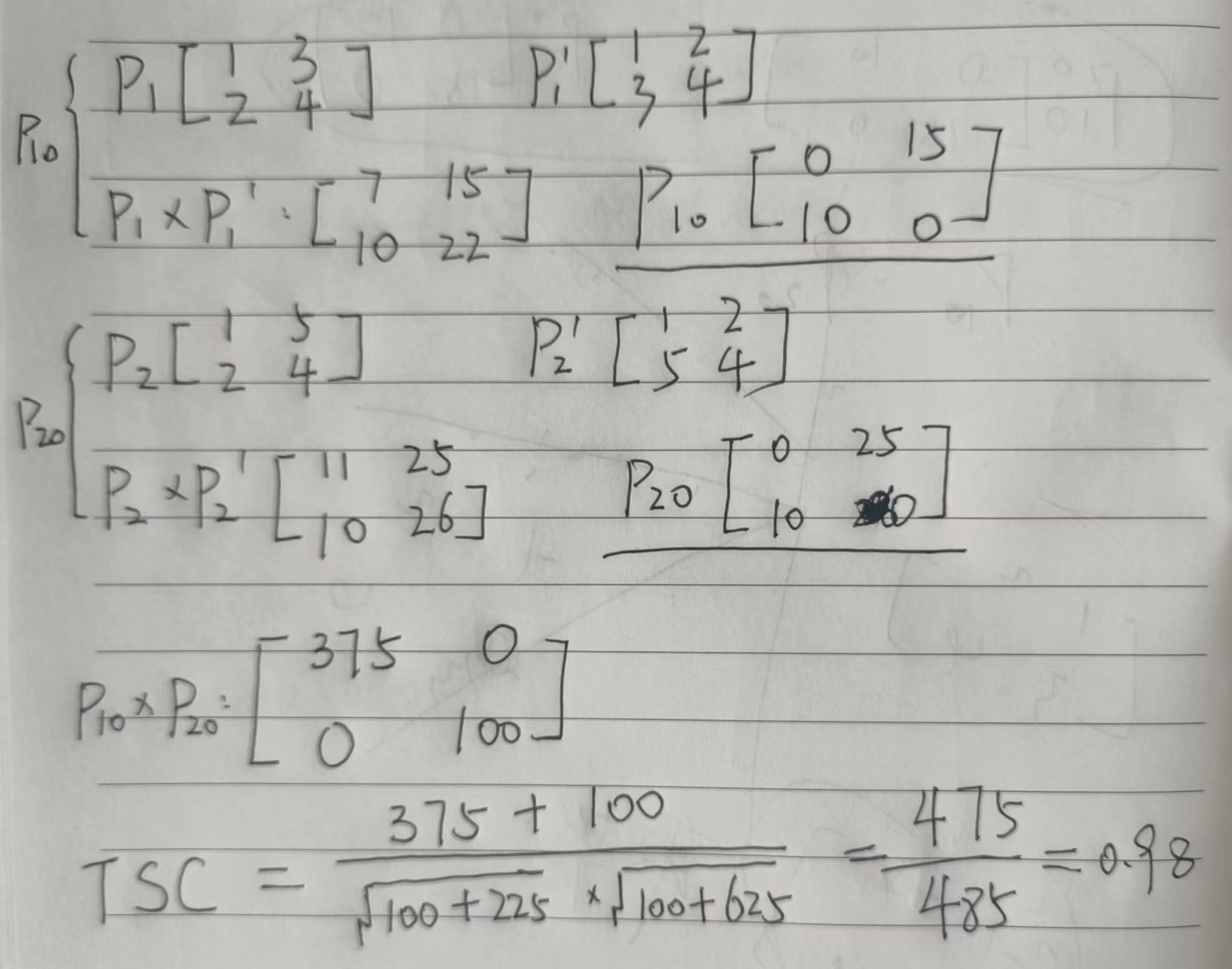
1.2 简单应用
1.2.1 乳腺癌分型
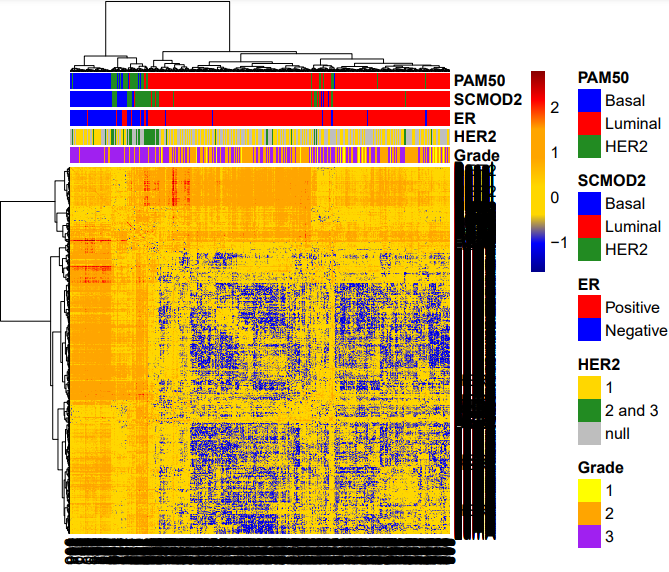
文章根据一个乳腺癌数据集表达矩阵,计算其中两两样本间分别关于ESR1、ERBB2相关基因集的相似性。基于此计算样本间的欧几里得距离(详见原文代码),用于层次聚类。
根据样本已知注释评价聚类结果,如下图所示具有较好的一致性。
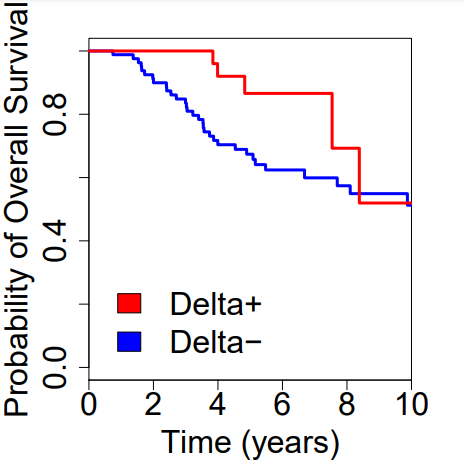
1.2.2 生存分析
同样的数据集,根据不同给药处理分为四组,每组单独分析。在每组中,找到生存最好、最差的10%样本作为标准,计算其余每一样本分别与之的相似性,进行比较,得出Delta结果。 $$ Delta = (similarity,to,good,survival,cohort) - (similarity,to,poor,survival,cohort) $$ 如果Delta>0, 表示该样本与生存良好的样本集更接近;反之Delta<0, 表示与生存较差的样本集更接近。
然后根据Delta正负性对每组样本进行分组生存分析,用以评价Delta的生存预后价值。
1.3 数据分享
1.3.1 SIGN包
文章编写了一个R包 SIGN(Similarity Identification in Gene expressioN)用于计算两个基因表达矩阵的相似性,已经上传到CRAN。
其核心代码如下
|
|
1.3.2 codeocean
文章已将绘制Fig2的代码、数据上传到 CodeOcean,可通过http://bit.ly/2PMwegY访问,进行复现分析。
Paper2:应用发现
Pathway-Based Drug Response Prediction Using Similarity Identification in Gene Expression
Frontiers in Genetics 2020 / 09 IF=4.6

2.1 应用思路
-
拉帕替尼(Lapatinib)和曲妥珠单抗(trastuzumab)是针对HER2+型乳腺癌的药物,但并非所有HER2+型乳腺癌患者均有效(40%)。
-
因此文章想鉴定出根据患者的基因表达情况可以预测HER2+型乳腺癌患者对上述药物是否响应的biomaker。
-
一方面考虑单基因作为biomarker的区分效果,另一方面使用上面提及的TSC score方法评价通路作为biomarker的预测效果。
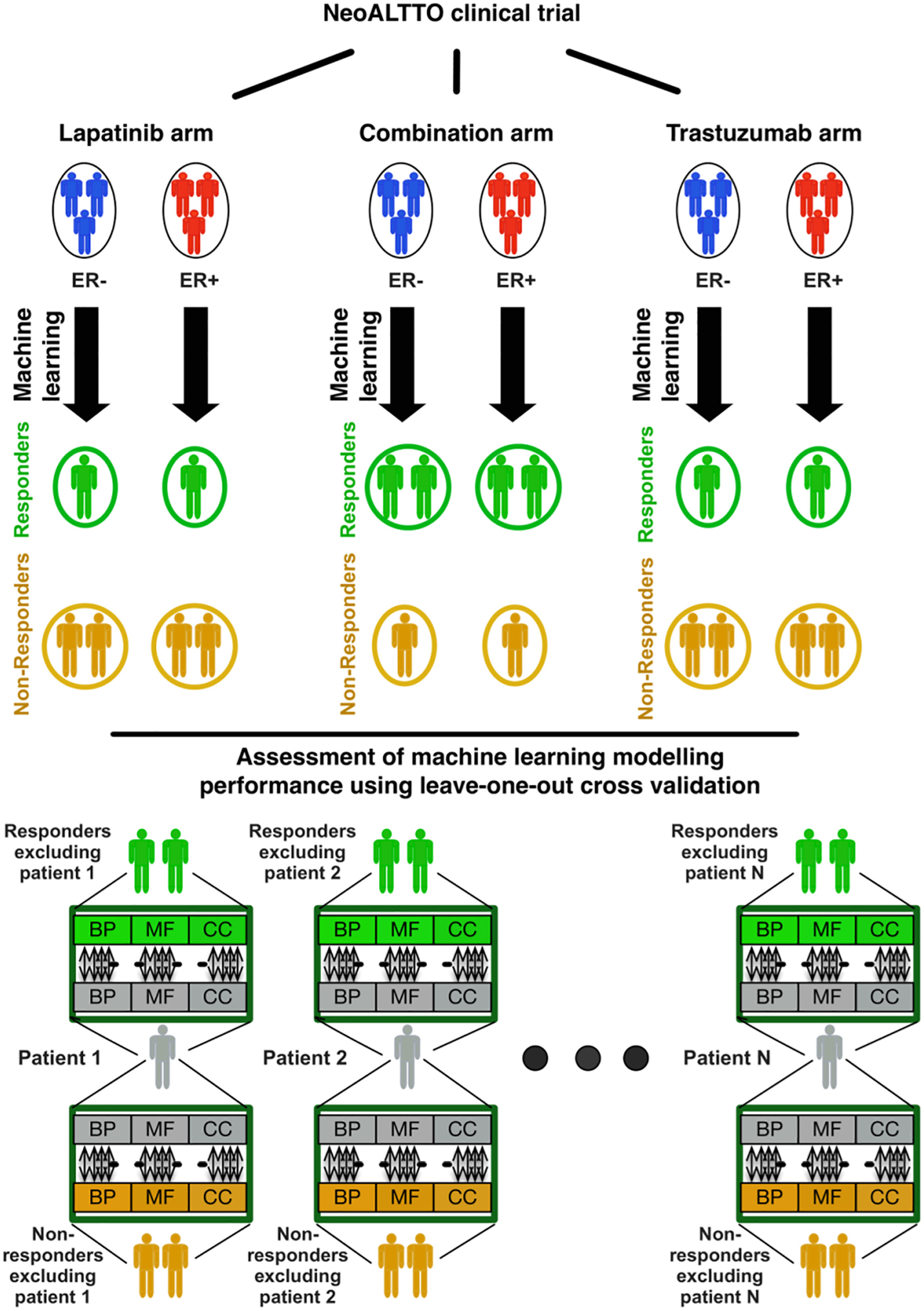
2.2 研究步骤
2.2.1 数据与方法
-
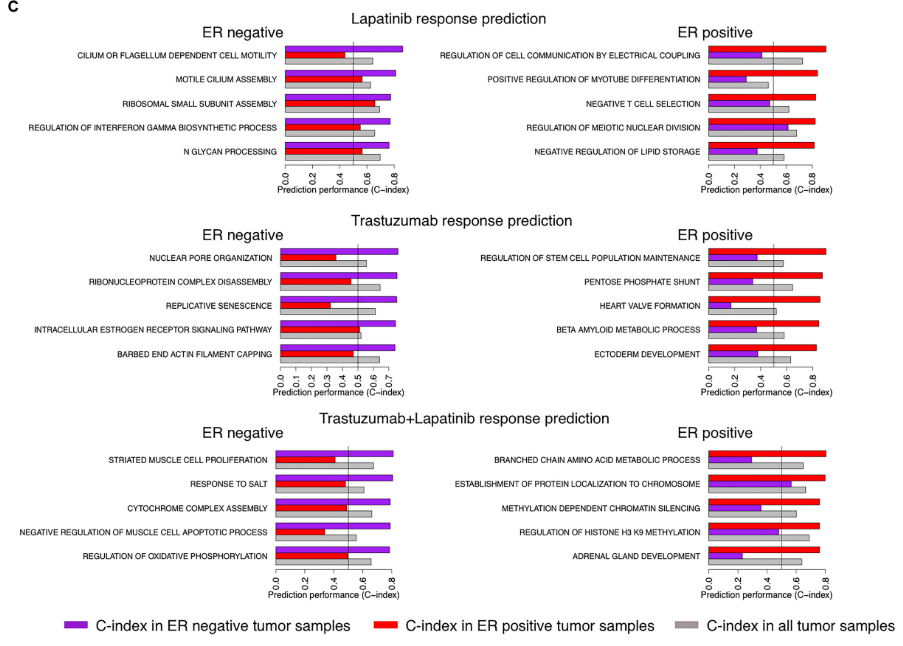
使用来自NeoALTTO clinical trial数据集,根据治疗方式分为Lapatinib、Trastuzumab,以及联用组。并进一步根据ER状态分为阴性组与阳性组。
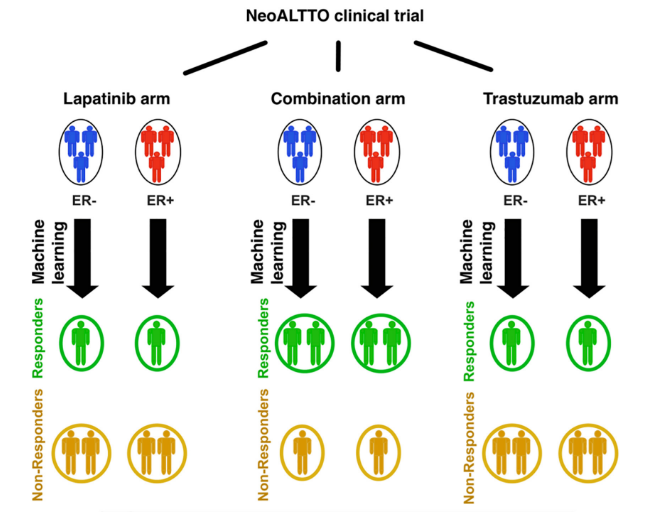
Relying on the common knowledge on ER being one of the main drivers in breast cancer development and progression (Fuqua,1997), we stratified our analyses based on the ER status.
-
根据Rate of pathological complete response (pCR),将患者分为Responders,与Non-responders。相当于贴标签。
-
使用C-index(concordance index)值用于评价gene/pathway的预测分类性能
2.2.2 单基因biomarker分析
根据患者的基因表达与响应状态关系,计算C-index。使用置换检验评价结果的显著性。
结果发现最好的单基因C-index值为0.68,即使是marker基因ERBB2的C-index值仅为0.59。
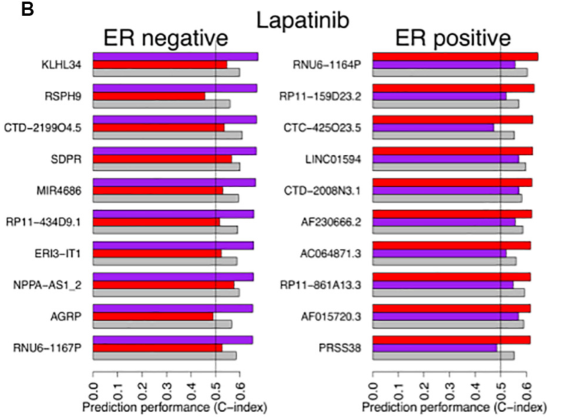
2.2.3 通路biomaker分析
-
通路选择:GO term中基因数10~30之前的通路
-
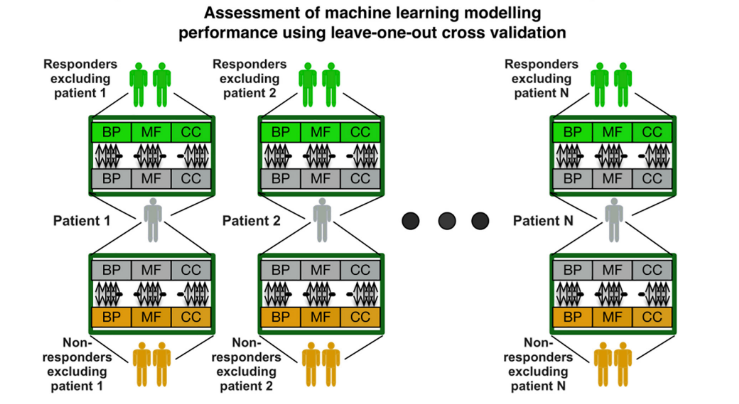
TSC预测:对于每一组来说,选择其中一个病人进行预测。分别计算其与剩余所有Responder与Non-responders 的相似度。
将该样本定位为距离相距较近的一组。对所有样本逐一按此法预测。
-
留一法交叉验证:选择一个病人,分别计算其与剩余样本中任意5个Responder的通路相似度,以及任意5个Non-Responder的通路相似度,比较二者结果的中位数,贴标签;重复100次取众数。计算C-index。
- 结果发现,每组的Top通路的C-index可达到0.8,优于其它机器学习的预测结果。