A network approach reveals driver genes associated with survival of patients with triple-negative breast cancer
April 19, 2021; IF=5.4
https://doi.org/10.1016/j.isci.2021.102451

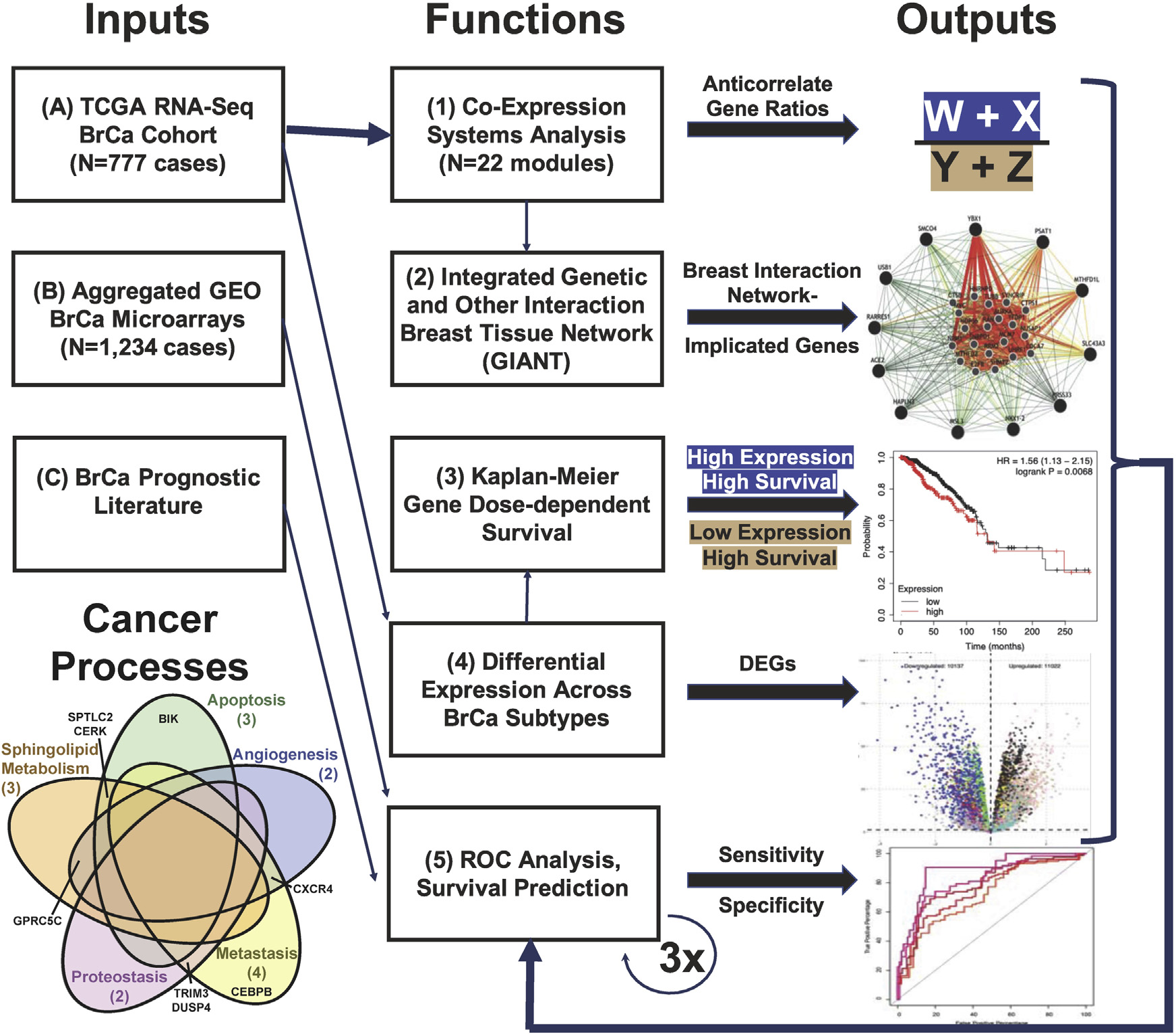
1、WGCNA分析
(1)对773个来自TCGA的BRCA(包括92个TNBC)的31338个基因表达数据进行WGCNA网络共表达分析
- 计算邻接矩阵的相关性指标选择 bicor(biweight midcorrelation)
- 得到22个模块,计算模块相关的ME与kME
- ME:每个样本对于每个模块的特征值
- kME:每个基因对于每个模块的相关性
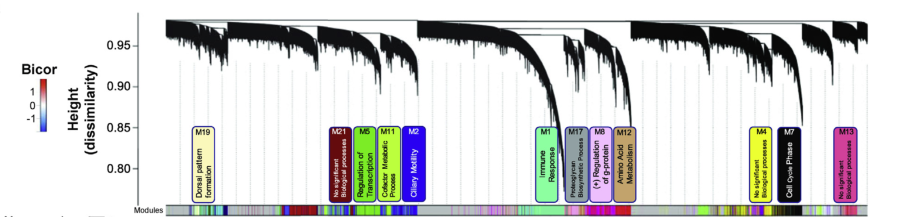
(2)使用方差分析,根据WGCNA鉴定的样本模块ME特征值与BRCA样本的subtype,发现与亚型分型最相关的12个模块
- 前期根据每个模块的基因组成进行富集分析,注释每个模块的相关通路(GO BP)
- 其中M12模块与TNBC分析最正相关(bicor = 0.69);M2模块与TNBC最负相关(bicor = -0.69)
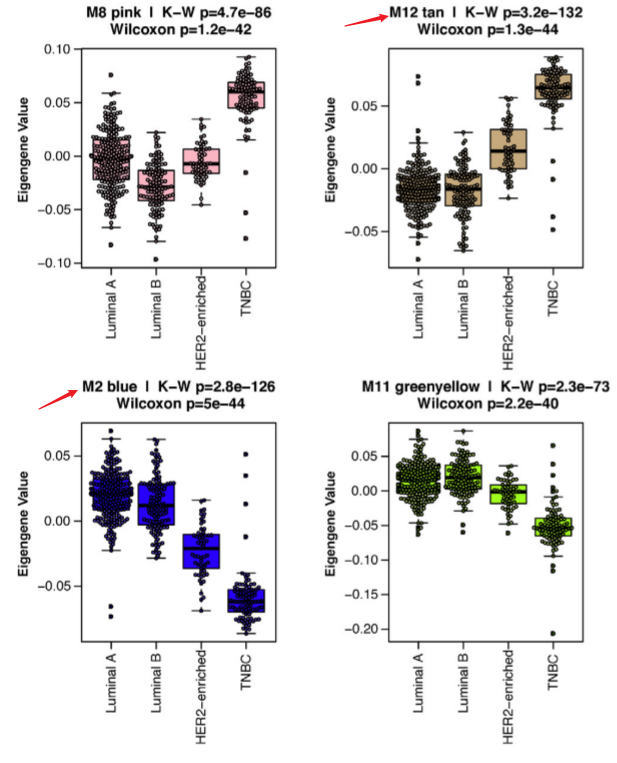
(3)对模块进行聚类分析,发现22个模块可以分为3类;其中的两类与TNBC相关,但相反(anti-correlated)
- 对于每个模块的样本ME值与样本TRAIT数据进行相关性分析,其中重点关注与样本的TNBC二分类以及生存时间的相关性。目标是希望找到与TNBC二分类正相关与生存时间负相关的模块。
- 发现M12为代表的一类模块(M7,M21,M17,M8)与TNBC正相关,生存时间负相关,统称为M12-like module
- 发现M2为代表的一类模块(M11,M5,M19)与TNBC负相关,与生存时间正相关,统称为M2-like module
- 综上找到与TNBC分型相关的两大类模块,包括9个模块
与TNBC正相关表明,模块特征值越大,越符合TNBC;与TNBC正相关表明,模块特征值越小,越符合TNBC。
2、差异分析与生存分析
(1)差异分析:使用T检验,分析TNBC分别与其它三种BRCA subtype的差异基因 BH FDR校正
- 取三次差异分析结果中具有相同方向的显著TNBC DEG,共1518个。
(2)模块ME生存分析:基于TCGA的773样本的RFS状态进行生存分析
- 首先分析每个模块的ME高低分组与生存的相关性:分为M12-like与M2-like两类模块讨论,总体来说M2-like模块低表达组(lower ME)的样本更容易复发;M12-like模块与之相反。
(3)单基因生存分析:基于GEO的1234个样本的RFS状态进行生存分析
- 根据之前的差异分析结果,标记M12 TNBC top20(相对Luminal A)的上调基因;标记M2 TNBC top20(相对Luminal A)的下调基因。进行单基因生存分析。
- 结果发现M12模块的PSAT1、ART3等5个基因;M2模块的TFF1、SCUBE2等6个基因具有生存显著相关性。
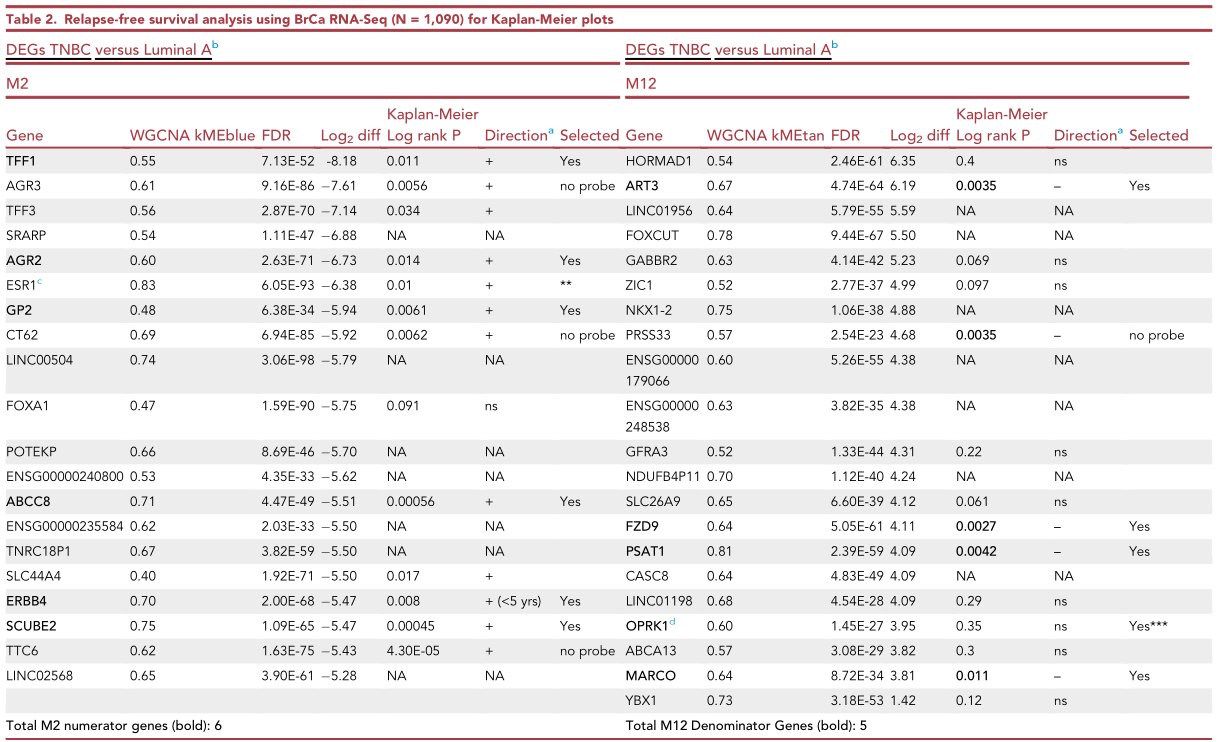
3、GIANT分析
(1)挑选出M2-like、M12-like的hub基因(Top10,kME>0.6)
(2)使用GIANT数据库(http://giant-v2.princeton.edu/)搜索在乳腺组织中,与这些hub gene高相关的基因。
- 对于M2、M12模块也加入哪些虽然不是hub,但是生存高相关的基因。
(3)结果发现M7,M21,M17,M8,M12,M2这6个模块,具有较高的连接度关系
- M12模块的hub基因,PSAT1,YBX1,MTHFD1L具有最高的连接度。
- M2模块中整体连接度比较高,hub基因ESR1,TFF1,SCUBE2等基因比较特殊。

4、预后模型
-
基于GEO的1234个BRCA样本的RFS状态,训练基因组合模型,使用ROC曲线评价模型对于BRCA以及TNBC的预测性能。
-
如下式,定义一个score:分子来自与TNBC负相关的M2-like module的基因;分母来自与TNBC正相关的M12-like module的基因。
-
分子与分母的基因数保持相同,即基因数比(1:1,2:2,3:3);每个基因的权重设置为1。
-
若M2相关基因的表达值越低、同时M12相关基因的表达越高;则score值就越低→越符合TNBC。
$$ score = \frac{M2-like ,gene(s)}{M12-like ,gene(s)} $$
-
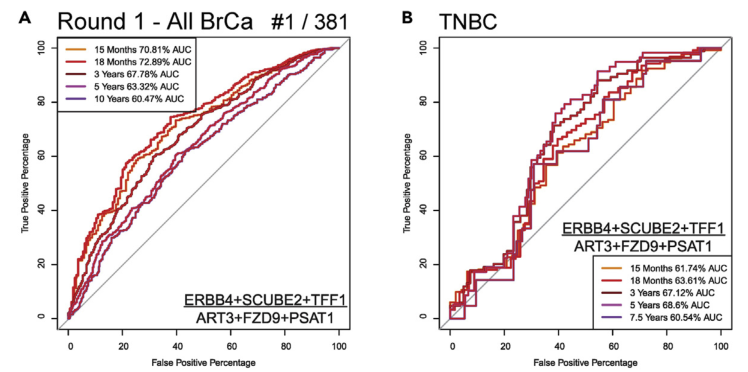
4.1 第一轮ROC
选用来自第二步中挑选的11个来自M2(5)、M12(6),与生存相关的TNBC差异基因。
- 共有381种模型组合可能;
- 对BRCA预测最佳模型的基因组合为(ERBB4 + SCUBE2 + TFF1/ART3 + FZD9 + PSAT1),但是在TNBC中表现不佳。
- ERBB4/FZD9组合在TNBC的5年生存率预测中效果最佳
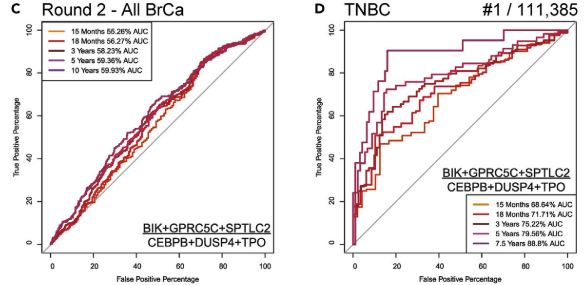
4.2 第二轮ROC
扩大基因的选择范围,考虑更多的module 差异基因或者hub基因
-
分子包括ERBB4、SCUBE2等24个来自M2-like 模块;分母包括ART3, FZD9,PSAT1等34个来自M12-like 模块;
-
共有11385种组合可能(包括1:1, 2:2, 3:3);
-
针对TNBC的Top最佳模型在TNBC中性能明显优于上一轮,但在BRCA数据中预测性能最低;
-
最佳基因组合为(BIK +GPRC5C+ SPTLC2/CEBPB + DUSP4 + TPO),而且大些基因大多出现在Top100 TNBC模型中。
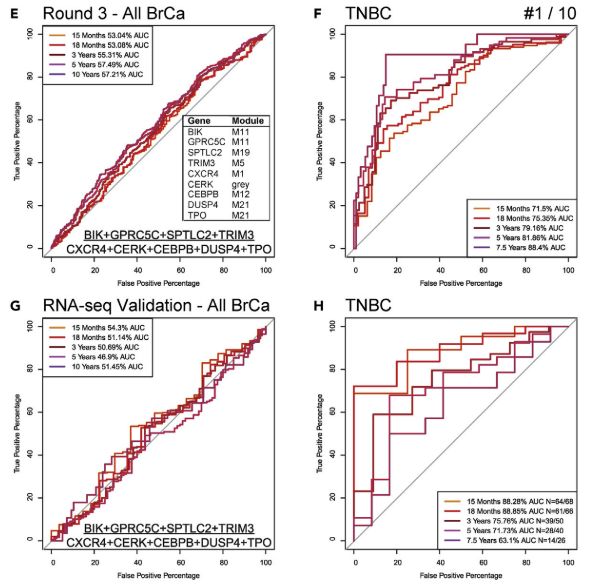
4.3 第三轮ROC
- 在第二轮的最佳TNBC模型6基因的基础上,手动根据文献添加补充候选基因;
- 最终敲定最优的9基因模型对TNBC具有良好的特异性,对BRCA性能一般。
- 最后在TCGA的BRCA数据上取得了验证。
小结:这篇文章目的是找到一个特异性针对TNBC的预后模型的基因组合。首先使用WGCNA找到与TNBC分型相关的模块,然后结合差异分析、生存分析以及组织间基因相互作用分析从关键模块定位到关键基因。关于模型基因组合,第一次见到本文的计算(类似于传统riskscore)方式,觉得比较新奇;结合三轮ROC分析,不断增加候选基因,筛选了1W+的基因组合,最终获得了一个理想的模型。