题目: Integrated analysis to identify the prognostic and immunotherapeutic roles of coagulation-associated gene signature in clear cell renal cell carcinoma
期刊 | 日期:Frontiers in Immunology | 17 March 2023
DOI:https://doi.org/10.3389/fimmu.2023.1107419
凝血相关基因 coagulation-related genes,CRGs
透明细胞肾细胞癌 clear cell renal cell carcinoma,ccRCC
0、主要数据方面
- TCGA-KIRC转录组数据
- GSE159115单细胞数据(ccRCC+normal)
- CRGs来自MsigDB数据库(HALLMARK_COAGULATION, MALLMARK_ COMPLEMENT)
1、两个角度分析CRGs
1.1 差异基因分析
- TCGA-KIRC中normal与tumor的DE CRGs(limma, P<0.05)
- scRNAseq中normal与tumor epi细胞群差异分析
- 使用SCEVAN软件区分肿瘤细胞
- 取上述二者的交集基因,共16个
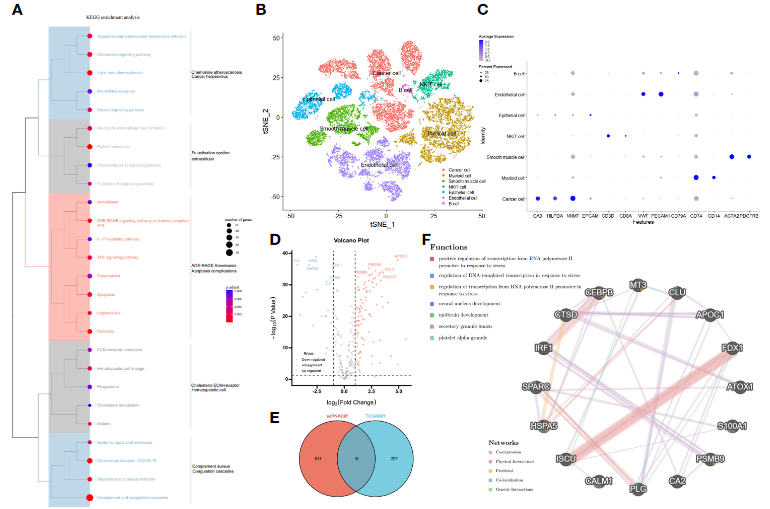
1.2 样本亚群分型
- 使用CRGs将TCGA-KIRC样本首先分为4类,进一步合并为2类
- ConsensusClusterPlus包
- (maxK=7, reps=100, pItem=0.8, pFeature=1, distance=“manhattan”, clusterAlg=“pam”)
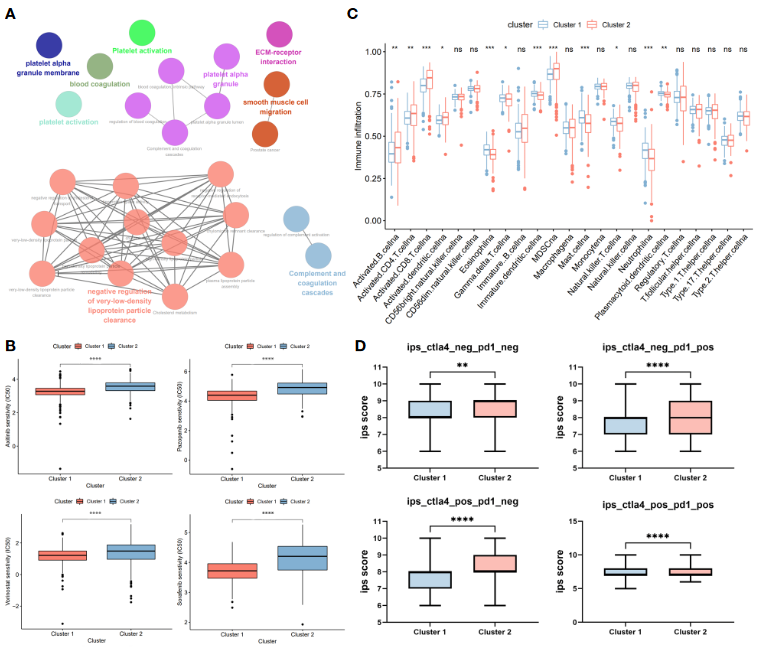
- 样本类群之间差异性
- 生存分析
- 通路分析–ClueGO(Cytoscape)
- 免疫浸润分析–ssGSEA
- 免疫治疗差异–TCIA数据库
- 药物敏感度差异–pRRophetic
2、诊断与预后模型
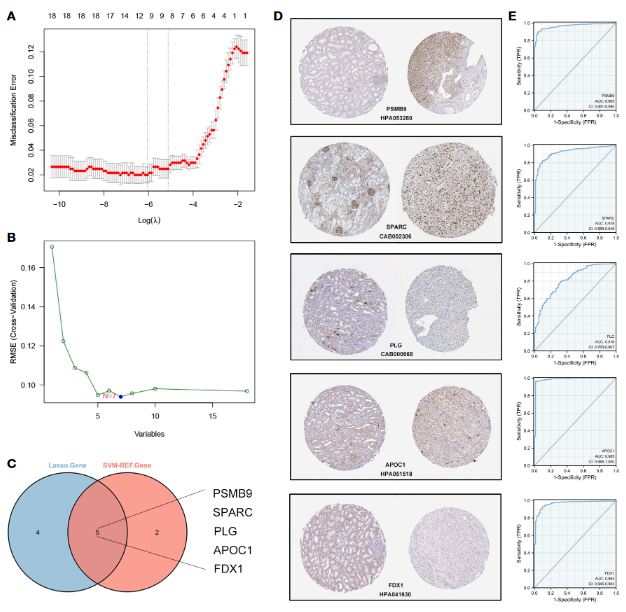
2.1 诊断模型
- 基于16个hub基因,分别使用LASSO与SVM方法识别ccRCC与normal(TCGA)间显著差异的基因组合
- 对得到的5个交集基因,先结合HPA数据库验证差异表达,在计算AUC指标评价
2.2 预后模型
- 将TCGA数据集分为训练集与测试集,再加一个外部测试集(E-MTAB-1980)
- 基于279个CRGs进行单变量cox回归分析,进而lasso回归,得到8个基因组合;根据权重系数计算riskscore
- 在每份数据集中,根据riskscore中位数分组进行生存差异分析,计算AUC指标
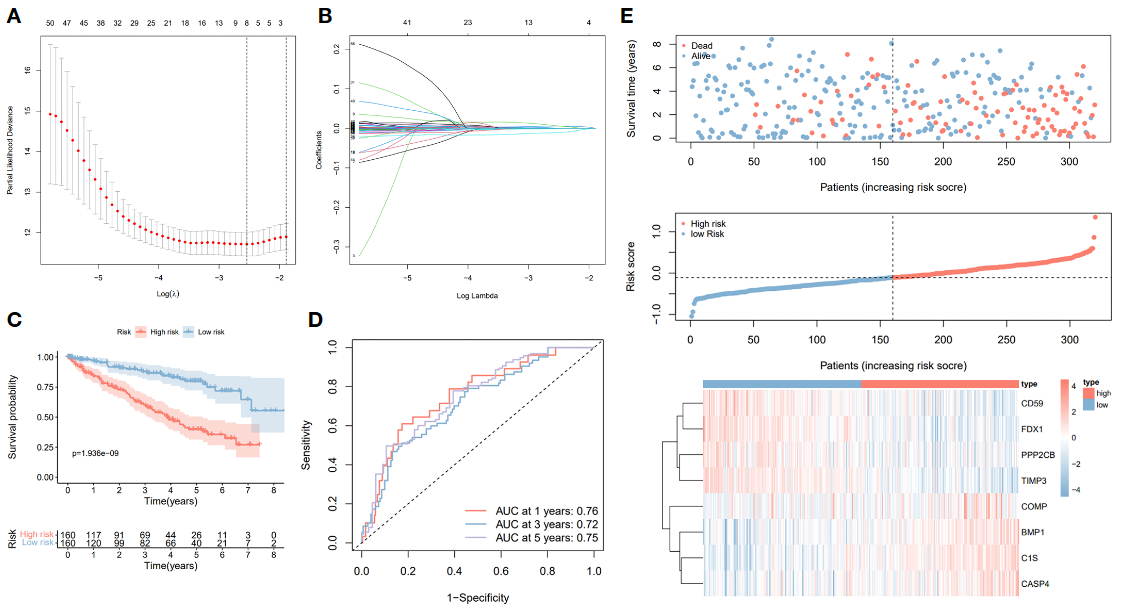
- 综合Riskscore以及Stage, Grade, Age,分析因素独立性,绘制综合诺模图,再次进行AUC评价。
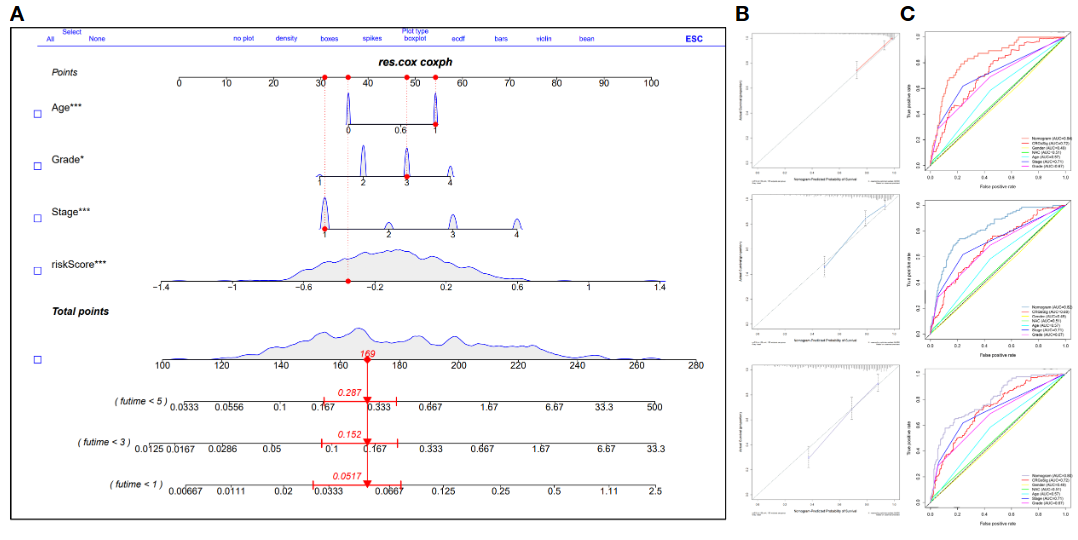
文章最后一部分结合多个数据集,以及自己的湿实验(临床样本+细胞系)进一步证明了一个key CRG的差异表达。
文章小结:属于是一类基因在一种肿瘤的分析套路。首先基于结合TCGA与scRNA-seq筛选hub基因,在结合lasso与svm筛选diagnostic gene。然后分别中TCGA亚群分析以及常规预后分析流程阐释CRGs的应用价值。部分数据分析手段以及可视化具有借鉴意义。