题目 : Integrative Analysis From Multicenter Studies Identifies a WGCNA-Derived Cancer-Associated Fibroblast Signature for Ovarian Cancer
期刊 | 日期 : https://doi.org/10.3389/fimmu.2022.951582 | 06 June 2022
概述:发现6个基因(COL16A1, COL5A2, GREM1, LUM, SRPX, TIMP3)组成的卵巢癌肿瘤相关成纤维细胞相关的signature

1、数据收集与预处理
主要收集16个数据集,2742个卵巢癌病人转录组数据及对应的预后信息。
- TCGA队列:372个病人, FPKM标准化(另收集突变数据)
- GPL570平台的6个GSE数据集,597个病人,log2(TPM+1)标准化(下同)
- 对于相同平台的不同数据集使用sva::Combat校正批次(下同)
- GPL96平台的4个数据集,409个病人;
- GPL7759,GPL2986,GPL14951,各一个数据集
- PD-1,PD-L1治疗后的两个GSE数据集
主要使用GPL570与TCGA两个队列,其余数据用于后续验证以及衍生分析
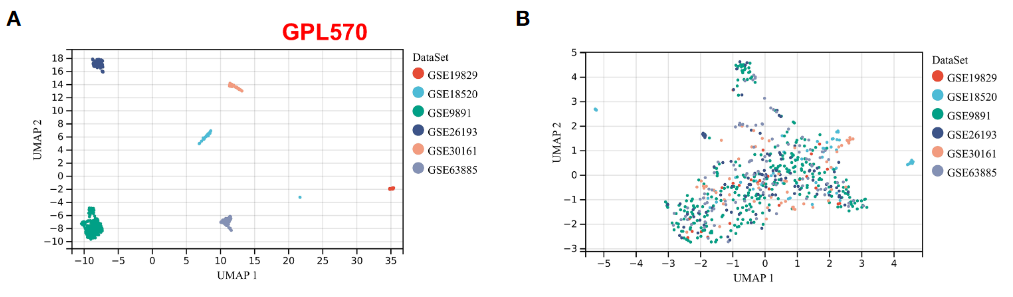
2、CAF相关hub基因筛选
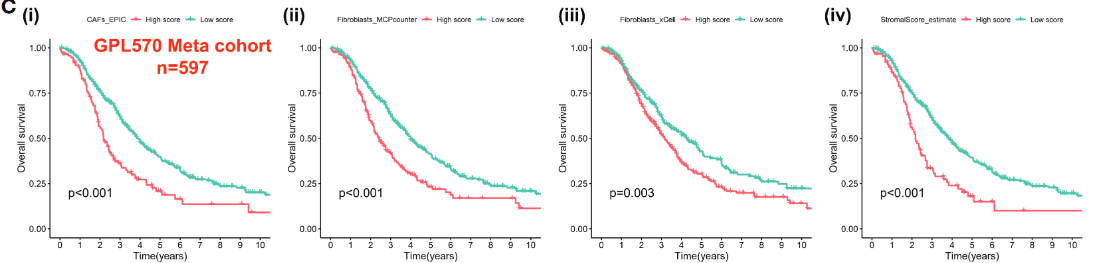
(1)基于IOBR包的4种算法,计算每个样本的CAF abundances and stromal scores;
(2)根据score将病人分为高低两组,TCGA与GPL570生存分析具有均显著意义
- 较高的CAF infiltration、stromal score与较差的生存预后相关
每个队列的组别划分阈值不相同可以理解,但作者并没有明确提及确定阈值的依据,包括后面的risk score分组分析。
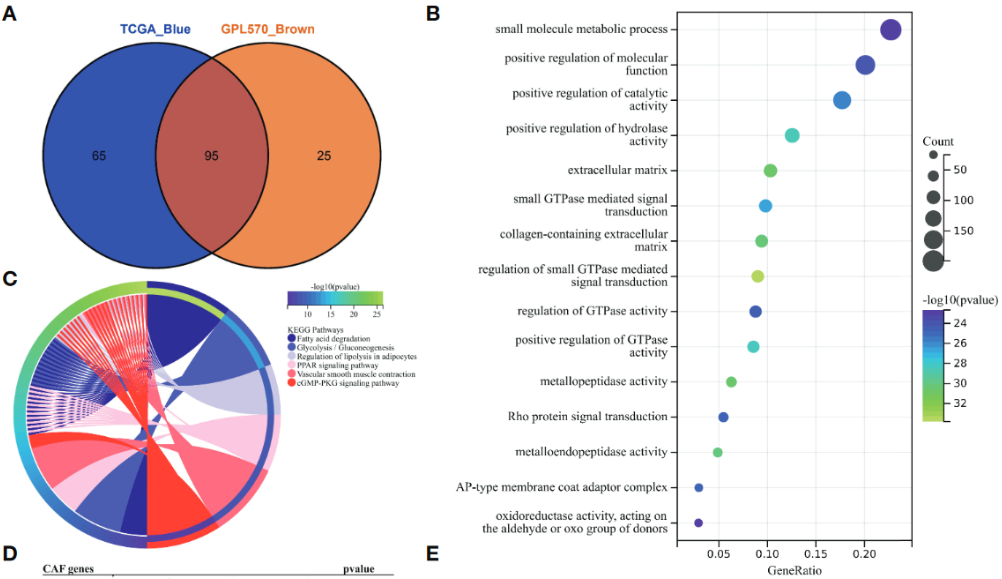
(3)分别对TCGA与GPL570进行WGCNA分析,各自鉴定出与样本CAF abundances and stromal scores最相关的一个模块,再取交集,得到95个CAF相关基因。
(4)对CAF相关基因进行初步的GO,KEGG通路富集分析,阐释相关通路。
3、构建预后模型signature
将GPL570队列用于训练集, 构建模型;将TCGA队列用于验证模型。
(1)针对上述95个CAF相关基因进行Univariate Cox regression analysis,得到63个显著基因(p<0.05)
(2)再经LASSO regression analysis,最终得到6个用于构建signature的marker基因
- COL16A1—0.0924;COL5A2— -0.0031;GREM1— 0.0847;
- LUM— 0.0069;SRPX— 0.0649;TIMP3— 0.0425
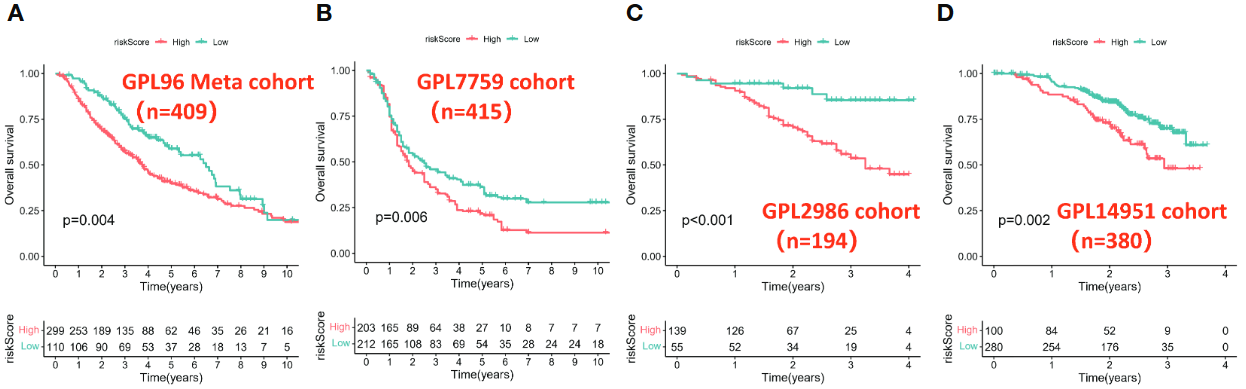
(3)根据上述基因权重,计算每个样本的risk score,进一步分为高低风险组,进行生存分析
- 分组具有显著生存预后意义,高分数→差预后
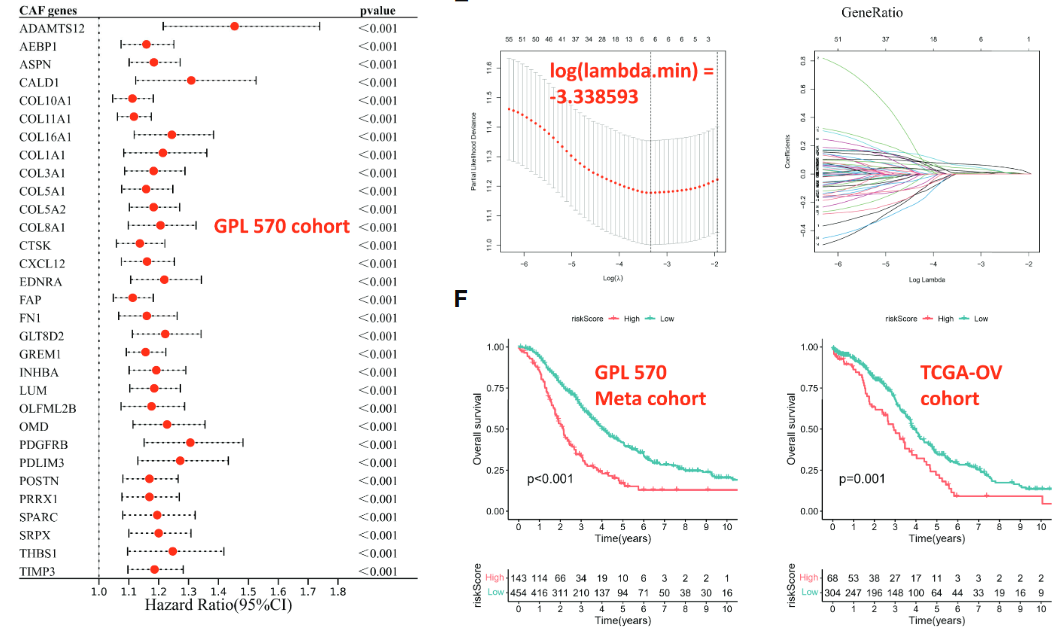
(4)其余4个GPL队列的数据验证,发现风险分组均具有显著意义。
4、预后模型衍生分析
通过不同角度论证所发现的signature具有重要意义
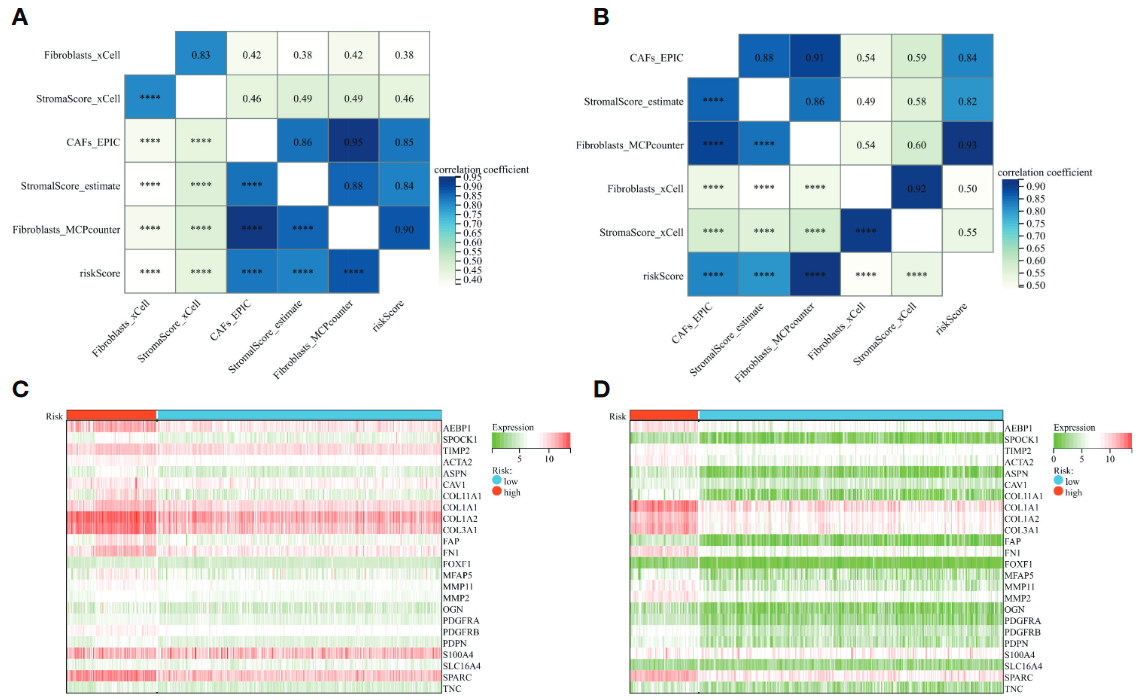
4.1 signature与CAF的关系
- 样本risk score与CAF浸润打分的相关性
- 样本risk score与已知CAF marker表达的相关性
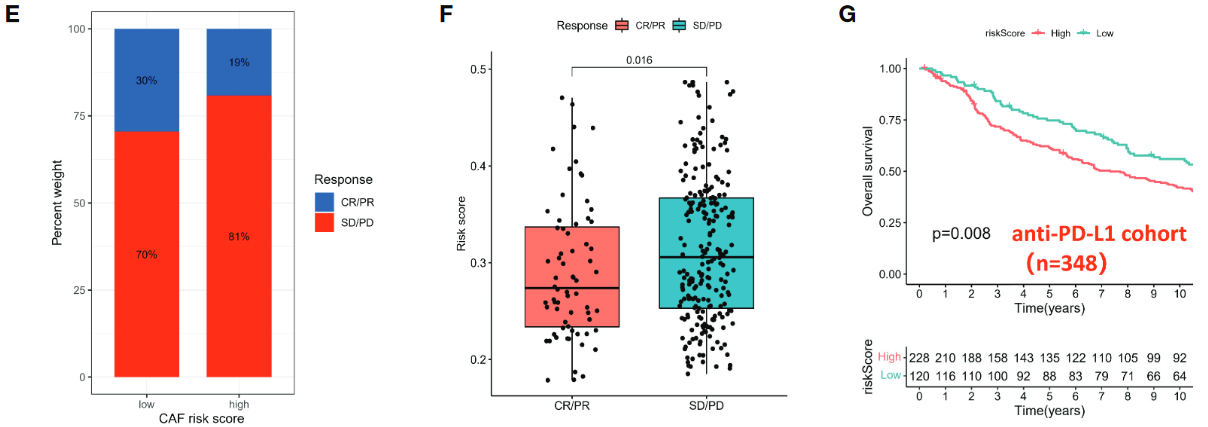
4.2 signature与免疫治疗的关系
PD-1/PD-L1可降低免疫效应,促进肿瘤生长; PD-L1 and PD-1 blockade 肿瘤免疫治疗手段
stable disease (SD)/progressive disease (PD) – 无效
complete response (CR)/ partial response (PR) – 有效
- high risk score样本与SD/PD有关
- high risk组与较差的生存预后相关
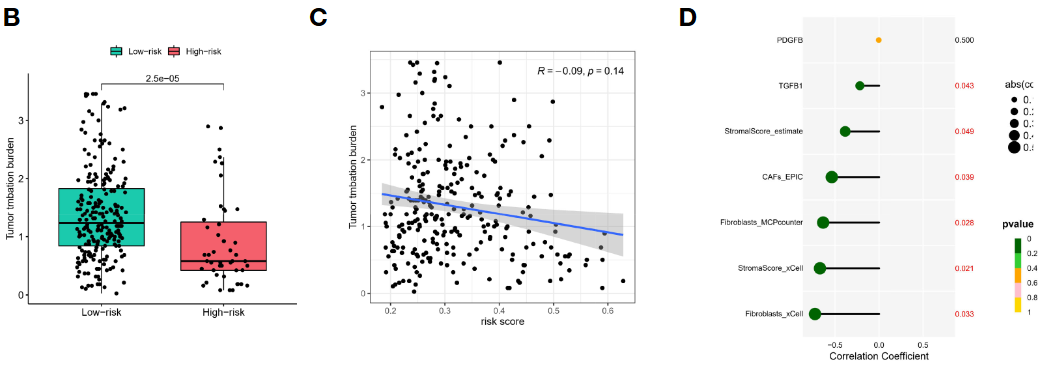
4.3 signature与肿瘤突变负荷
一般认为肿瘤突变负荷TMB越高,免疫治疗效果越好
- 结合TCGA突变数据,分析high/low risk score组的差异突变基因;
- 进一步分析TMB分数与risk score的相关性(maftools包的tmb函数)
4.4 signature的相关通路
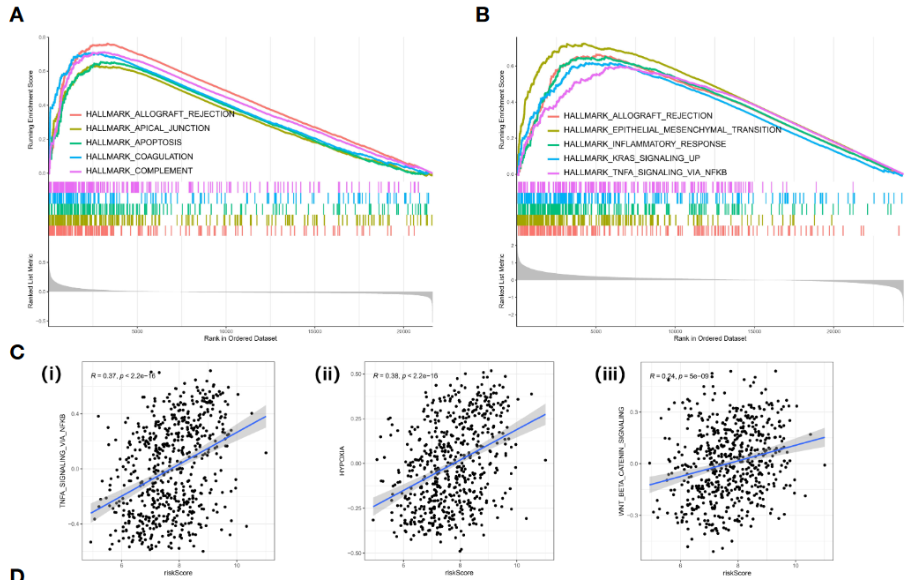
- high/low risk score组的GSEA显著富集通路
- 样本risk score与ssGSEA分数的高相关性通路
4.5 其余分析
(1)BRCA是否突变以及突变型与risk score的关系
(2)结合pRRophetic包预测对high/low risk score组的IC50具有显著差异的药物
(3)siganture在纤维原细胞系与卵巢癌细胞系表达差异
(4)OV scRNAseq中高表达siganture的细胞类型
5、小结与体会
- 本篇数据挖掘文章亮点之一是收集了大量样本数据,共2742个样本,并且均包含预后数据。其中主要使用的是GPL570所包含的仅600个样本数据。
- 分析思路主要分为3步(1)确定CAF相关基因范围;(2)建立CAF signature;(3)signature后续衍生分析
- 前面两步是很常规的signature挖掘过程;
- 后面一步则取决于个性的发挥。本篇文章花了相当的笔墨在这一步骤,可感觉有些分析实在有仅为了充实内容之嫌。
- 最后关于文章的存疑是目前仍未发现作者如何针对每个数据集的risk score确定分组阈值的方法,而这直接决定了生存分析结果,不知作者有意为之还是如何。