标题 Nicheformer: a foundation model for single-cell and spatial omics https://doi.org/10.1038/s41592-025-02814-z
发表 nature methods, 2025年10月
通讯 Fabian J. Theis
Github:https://github.com/theislab/nicheformer
1. 研究背景
-
传统的单细胞转录组(scRNA-seq)虽能捕获细胞异质性,但由于解离过程丢弃了空间结构(Niche),导致无法分析细胞间的通信机制。现有的单细胞大模型(如 scGPT, Geneformer)主要基于解离数据,对空间依赖性的特征捕获不足。
-
主要思路:能否通过在大规模空间组学数据上进行预训练,让模型学习到基因表达与空间微环境之间的隐性关联,并将这种能力迁移到非空间的 scRNA-seq 数据中?
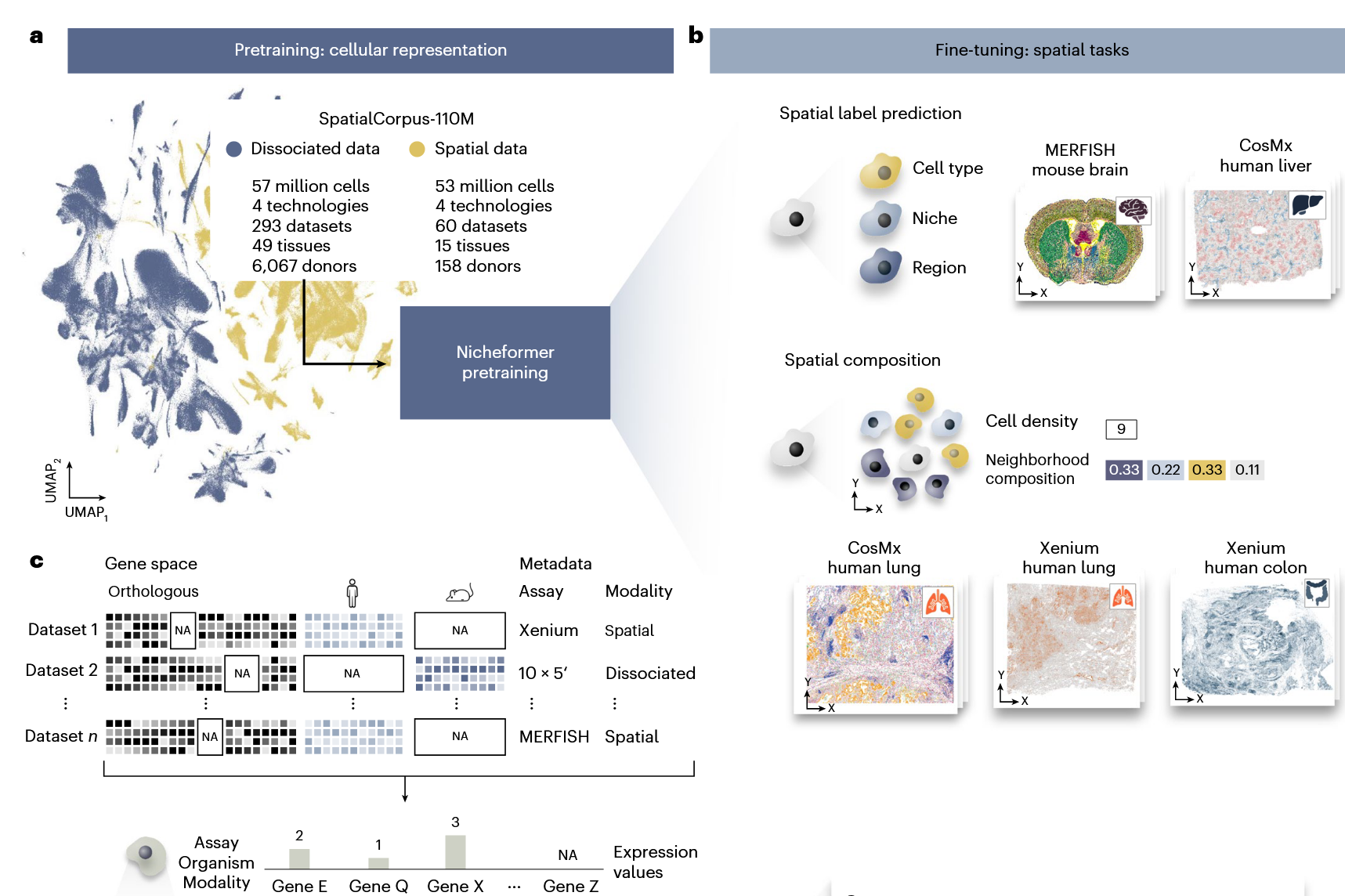
2. SpatialCorpus-110M 数据集
- 数据规模:包含 1.1 亿个细胞。其中 5,700 万为scRNA-seq,5,300 万为具有空转细胞。
- 覆盖广度:
- 人类与小鼠(通过 Ensembl ID 进行同源基因映射,最终 20,310 个total基因)。
- 多种imaing based空转平台(Xenium, MERFISH, CosMx, ISS)
- 73 个不同的组织器官
3. 模型架构与训练细节
3.1 输入编码
基本参考Geneformer的模式
- Rank Value Encoding:不直接输入表达量,而是根据基因表达量在细胞内的排名进行编码,以增强对测序深度和噪声的鲁棒性。
- 归一化策略 (Technology-specific Mean):为了处理跨平台差异,模型对每个基因基于特定技术的非零均值(Non-zero mean)进行归一化处理。
- Context Tokens (Meta Tokens):在基因序列头部插入 4 个特殊的 Token:物种 (Species)、性别 (Sex)、组织 (Tissue) 和平台 (Assay)
3.2 任务类型
- Pre-training:Masked Token Prediction (MTP),通过掩盖 15% 的基因 Token 让模型根据上下文预测缺失基因,从而学习基因间的协同表达关系。
- Downstream Adaptation:
- Linear Probing (线性探针):冻结编码器,验证 Embedding 层的生物学表征质量。
- Fine-tuning (微调):针对特定任务(如 Niche 预测、细胞密度回归)调整全量参数。
4. 主要结果
Result-1: 注意力机制的解释性
- 早期层 (Early Layers):注意力倾向于在所有基因间均匀分布,进行初步特征提取。
- 中间层 (Middle Layers):注意力集中在具有特定生物学功能的基因簇上。
- 深层 (Final Layers):注意力高度向 Context Tokens 偏移。这表明模型在最后阶段会利用“组织”或“平台”信息来最终修正表达模式的表征。
Result-2: 空间特征的迁移 (分类模型)
- 预测细胞所属cell type / niche / region
- transfer from spatial to scRNA-seq : 基于空转的分类器用于scRNA-seq的预测,然后再于ground truth作比较
Result-3: 邻域组成与密度预测 (回归模型)
- Neighborhood Composition (组成预测):
- 定义:预测细胞周围一定半径(根据平均覆盖 10, 20, 50, 100 个邻近细胞的物理距离动态调整)内的细胞类型比例。
- 意义:将定性的空间分类转化为定量的比例回归,证明了模型对复杂细胞交互模式的感知。
- Neighborhood Density (密度预测):
- 模型成功区分了健康组织与肿瘤组织的细胞分布密度特征(肿瘤区通常具有更高的细胞堆积密度)。
5. 不足之处
作者主要提到的limitation
- 论文提到,在提取细胞最终 Embedding 用于分类任务时,不应包含 Meta Tokens 的向量,而应仅取基因 Token 的平均值(Average Gene Token Embedding)。因为 Meta Tokens 包含过强的技术批次信息,会干扰生物学本质的聚类。
- 目前的输入序列没有显式的 $X, Y$ 坐标。未来可能引入 Graph Transformer 或空间位置编码(Spatial Positional Encoding)来直接处理几何拓扑关系。
- 目前主要针对转录组,未来可扩展至 ATAC-seq 或蛋白质组学数据。
我的想法
- Nicheformer本质还是single cell foundation model,只不过其训练数据一半是来自于空转数据。在预训练过程中还是以单个细胞为输入,不考虑其空间位置。
- 而加入空转数据的优势在学习到空转表达模式后,在下游的SFT任务中,可结合空间已知的信息设计微调模型。
- 其主要假设是在于根据细胞的表达谱,能够反推其周围空间信息。即观测到的细胞表达谱=自身信号 + 空间影响。